简单重复序列(simple sequence repeat,SSR)标记具有多态性丰富、操作简便、稳定性强等优点,被广泛应用于大豆种质资源遗传多样性研究[4,5,6]。大豆种质资源的遗传多样性是长期进化的产物,是大豆生存(适应)和发展(进化)的前提[7]。黑龙江省是我国春大豆的主产地,大豆遗传多样性的研究也受到育种者的重视。秦君等[8]利用60对SSR标记对黑龙江省140份代表性大豆品种进行分析,结果表明地方品种的遗传丰富度要高于育成品种,建议进一步开发地方品种重要基因;马淑梅等[9]利用40对引物研究俄罗斯远东大豆与黑龙江大豆发现,前者的遗传多样性高于后者;何海燕等[10]利用筛选后的9对SSR引物对黑龙江省25份大豆品种进行标记,结果表明黑龙江省大豆种质资源间有相对较宽的遗传基础,存在较大的遗传变异性;高运来等[11]利用经过筛选的43对SSR标记,对黑龙江省83个主栽大豆品种进行遗传多样性分析,结果表明黑龙江省大豆主栽品种间遗传组成相近,遗传基础狭窄,多样性低,建议拓宽黑龙江大豆的遗传基础。全面系统地评价黑龙江省大豆栽培品种的遗传多样性,掌握不同品种的遗传基础及品种间的亲缘关系,可为大豆优质种质资源的利用提供理论基础。
1 材料与方法
1.1 材料
试验于2017年在黑龙江大学农学楼进行。供试材料为东农57、合丰23、合丰46、黑河18和合丰47等202个大豆品种,包括黑龙江省育成品种187个,黑龙江省外育成品种15个,将试验材料按育种单位划分为10个类型(表1)。
表1 供试材料育成单位及编号
Table 1
| 类型Type | 育成单位Breeding unit | 编号-材料Code-material |
|---|---|---|
| A | 东北农业大学大豆研究所 | 1-东农24,2-东农36,3-东农38,4-东农42,5-东农43,6-东农47,7-东农48,8-东农49,9-东农50,10-东农51,11-东农52,12-东农53,13-东农54,14-东农55,15-东农56,16-东农57,17-东农5号,18-东农67 |
| B | 中国科学院东北地理与农业生态研究所 | 19-东升1,20-东升3号,21-东升9,22-东生7,23-东生8 |
| C | 黑龙江省农业科学院克山分院 | 24-丰收06,25-丰收11,26-丰收20,27-丰收24,28-丰收25 |
| D | 黑龙江省农业科学院佳木斯分院 | 29-合丰1,30-合丰14,31-合丰16,32-合丰23,33-合丰24,34-合丰25,35-合丰26,36-合丰28,37-合丰29,38-合丰32,39-合丰34,40-合丰36,41-合丰37,42-合丰38,43-合丰39,44-合丰41,45-合丰42,46-合丰43,47-合丰44,48-合丰46,49-合丰47,50-合丰48,51-合丰50,52-合丰51,53-合丰52,54-合丰53,55-合丰54,56-合丰55,57-合丰56,58-合农59,59-合农60,60-合农61,61-合农64,62合农65,63-合农66,64-合农68,65-合农75,66-合农76,67-合丰05,68-合丰06,69-合丰22,70-合丰30,71-合丰35,72-合丰40,73-合丰45,74-合丰57,75-合丰58,76-合丰60,77-合丰61,78-合丰62,79-合农62,80-合农63,81-合农67,82-合农69,83-合农70 |
| E | 黑龙江省农业科学院黑河分院 | 84-黑河35,85-金源55,86-黑河10,87-黑河11,88-黑河12,89-黑河14,90-黑河15,91-黑河16,92-黑河18,93-黑河19,94-黑河20,95-黑河21,96-黑河22,97-黑河23,98-黑河24,99-黑河25,100-黑河26,101-黑河33,102-黑河39,103-黑河43,104-黑河44,105-黑河46,106-黑河49,107-黑河50,108-黑河51,109-黑河52,110-黑河53,111-黑河56 |
| F | 黑龙江省农业科学院大豆研究所 | 112-黑农37,113-黑农40,114-黑农44,115-黑农48,116-黑农51,117-黑农52,118-黑农58,119-黑农60,120-黑农61,121-黑农62,122-黑农64,123-黑农65,124-黑农66,125-黑农68,126-黑农71,127-黑农83,128-龙豆1号,129-黑农69,130-黑农84,131-黑农85,132-哈05-5675,133-哈06-1939 |
| G | 黑龙江省农业科学院绥化分院 | 134-绥农22,135-绥农24,136-绥农26,137-绥农04,138-绥农25,139-绥农31,140-绥农35,141-绥农36,142-绥农38,143-绥农40,144-绥农41 |
| H | 农垦地区育种单位 | 145-北豆14,146-北豆5号,147-垦豆18,148-垦豆3,149-垦豆40,150-垦豆41,151-垦豆43,152-垦丰14,153-垦丰16,154-垦丰17,155-垦丰20,156-垦丰22,157-垦丰23,158-垦鉴27,159-垦鉴43,160-垦农23,161-垦农26,162-垦农28,163-北豆10,164-北豆19,165-北豆21,166-北豆22,167-北豆34,168-北豆41,169-北豆42,170-北豆47,171-北豆49,172-北丰11,173-北丰14,174-宝农1,175-垦豆25,176-垦豆38 |
| I | 黑龙江省其他育种单位 | 177-北疆2号,178-北疆九1,179-华疆2,180-华疆4,181-嫩奥1,182-嫩奥3,183-绥杨1号,184-北疆1,185-育丰115,186-育丰20,187-哈北46-1 |
| J | 黑龙江省外育种单位 | 188-登科1,189-登科8,190-登科5,191-赤豆1,192-赤豆3,193-俄豆-1,194-吉育95,195-长密30,196-长农18,197-长农24,198-长农26,199-长农27,200-长农28,201-长农29,202-长农31 |
1.2 试验方法
1.3 数据分析
1.3.1 多样性分析 通过软件PowerMarker version 3.25计算SSR位点的等位片段数、每个位点的多态性信息量PIC值和群体的平均基因多样性指数He。
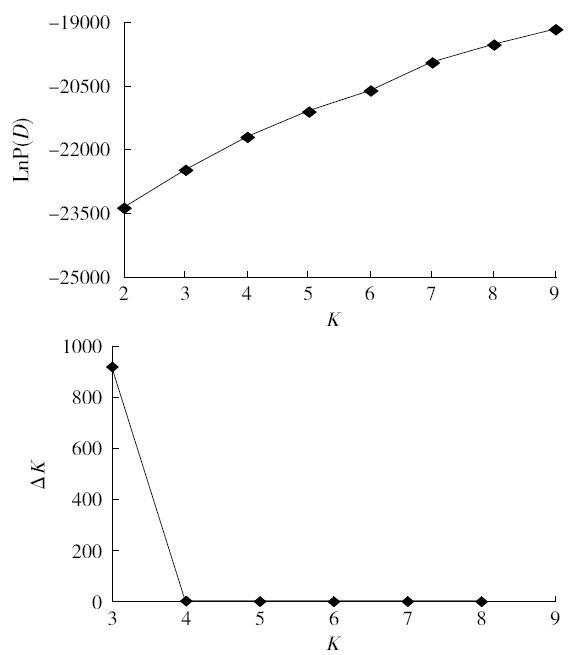
1.3.2 群体结构分析 利用Structure 2.2软件对202份大豆品种进行群体结构分析,将群体数K的取值范围设定为2~9,将MCMC(markov chain monte carlo)开始时的预迭代长度(length of burn-in period)设为10 000次,再将其后的迭代次数设为100 000次,然后依据似然值最大的原则从后验概率LnP(D)中选取合适的K值。如果LnP(D)随着群体数的增加而增加,导致无法确定最合适的亚群数,那么需要利用系数ΔK来分析亚群构成。ΔK具体算法为|L″(K)|的平均数除以L′(K)的标准差,其中L′(K)=L(K)-L(K-1),|L″(K)|=|L′(K+1)-L′(K)|,L(K)=LnP(D)[18]。
1.3.3 主成分分析及聚类分析 采用Ntsys 2.10软件,对202份大豆材料的遗传相似性系数GS(genetic similarity coefficient)进行计算,GS=2MXY/(MX+MY),其中,MXY为品种X和品种Y在所有检测位点上共有片段的数目,MX为品种X在所有检测位点上的总片段数,MY为品种Y在所有检测位点上的总片段数,通过建立两两品种间遗传相似系数矩阵,得到主坐标分析聚类图,再以大豆品种单株材料间的遗传距离GD(GD=1-GS)为基础[19],运用软件MEGA7.0绘制邻接法(neighbor-joiningn,NJ)聚类图。
2 结果与分析
2.1 基于SSR引物扩增结果分析
利用均匀分布于大豆20条染色体上的187对SSR引物对202份大豆品种进行扩增发现,除4对引物satt237、satt332、sat-087、satt441外,另外183对引物均表现有多态性。在全部试验材料中总共检测到了808个等位变异,不同SSR位点等位变异数在2~10个,平均每对SSR引物的等位变异数为4.42个,其中satt703、satt311的等位变异数最多,均为10个,有20对引物只检测到2个等位变异。试验材料中平均多态性信息含量指数为0.406,其中引物sat-332的PIC值最高,为0.813,其等位基因频率最低,为24.3%;satt417与satt575的PIC最低,均为0.0098,等位基因频率最高,均为99.5%。群体的平均基因多样性指数(He)为0.45。
通过10个育成单位类型的多样性比较发现,类型D中的平均等位变异数最多,为3.70个,PIC值最高,为0.42;类型B中的平均等位变异数最少,为2.29个;类型G的PIC值最低,为0.37。
2.2 群体遗传关系及遗传结构特征分析
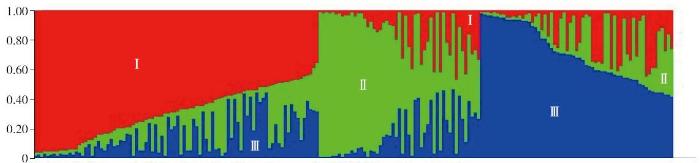
利用Structure 2.2分析大豆群体遗传结构,在K取值2~9时,LnP(D)随着K值的增大而增大。当K值为3时,ΔK值最大(图1),因此可以将202份大豆品种分为3类不同血缘,分别由Ⅰ类(90个品种)、Ⅱ类(51个品种)、Ⅲ类(61个品种)表示(图2)。不同血缘间存在一定程度的遗传混杂,Ⅰ类血缘中包含有90个品种,除了类型B、类型I没有分布以外,其他类型均以不同比例分布,其中包含类型D中的品种最多,为38个,占比为42.2%;Ⅱ类血缘中包含有51个品种,10个类型的品种在此均有分布,类型A和类型E占有比例最高均为21.6%,类型B最少只占2.0%;Ⅲ类血缘中包含有61个品种,类型C中的品种没有分布,类型D和类型E占有比例较高分别为23.0%和21.3%,类型J占比最少,仅为1.6%。
图1
图2
2.3 主坐标分析与聚类分析
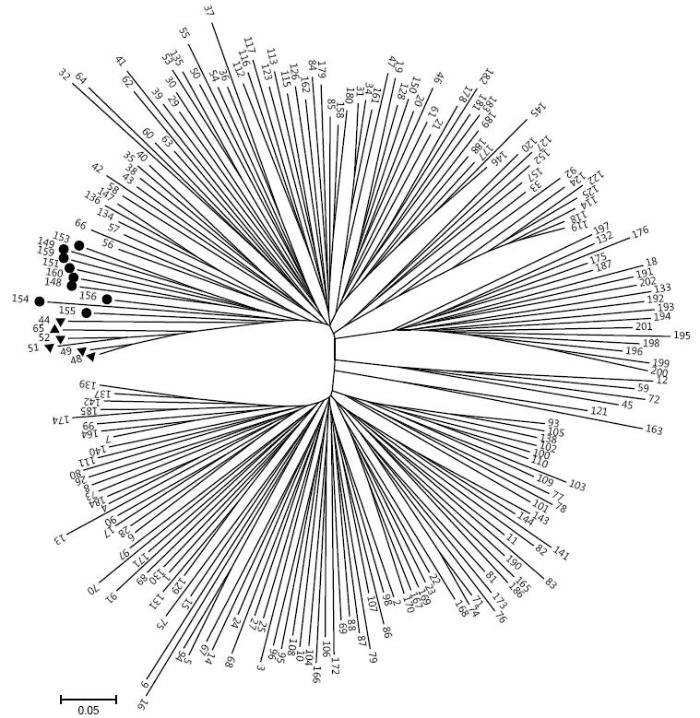
对全部试验材料的SSR标记遗传相似系数分析发现,其值介于0.283~0.930,平均为0.519。东农57与合丰23相似系数最小,为0.283,表明二者亲缘关系最远;合丰46与合丰47相似系数最大,为0.930,表明二者亲缘关系最近。
图3
图3
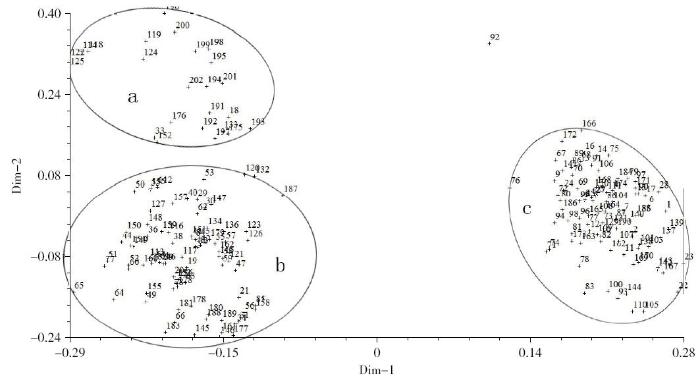
依据SSR标记对202个大豆品种进行主坐标分析的二维散点图
图中材料编号同
Fig.3
Two-dimensional scatter plot of principal coordinate analysis of 202 soybean varieties based on SSR markers
The material codes in the figure are the same as table 1, the same below
图4
图4
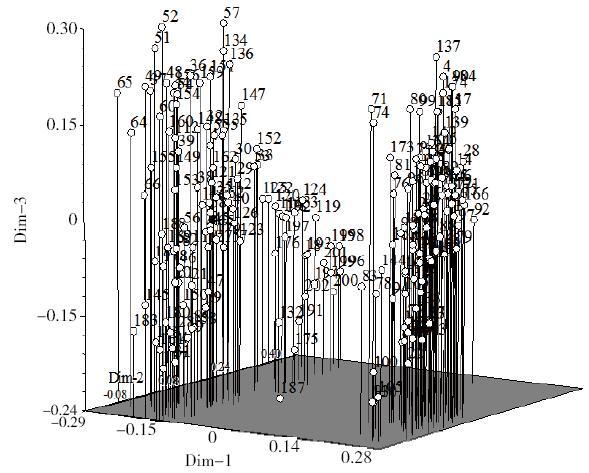
依据SSR标记对202个大豆品种进行主坐标分析的三维散点图
Fig.4
Three-dimensional scatter plot of principal coordinate analysis of 202 soybean varieties based on SSR markers
如图3所示,202个大豆品种除编号92品种(黑河18)外,其余均分布在3个类群中。第1和第2主坐标中的位置远近代表了其亲缘关系的远近,其中类群c和类群a、类群b在坐标系中相距较远,表明其遗传相似系数较小,种源间的遗传差异较大,即血缘关系较远;类群a与类群b相对距离较近,种源间的遗传差异较小,即血缘关系较近,同时也说明2个类群之间的基因存在一定的交流。
利用前3个主坐标排序得到图4,可以看出三维与二维分类基本一致。只是在第1、2主坐标分类的基础上,202个品种在第3(Dim-3)主坐标方向上可进一步细分,这能够从不同角度更加直观显示不同品种之间的遗传差异。
图5
3 讨论
3.1 黑龙江省栽培大豆遗传多样性
3.2 黑龙江省栽培大豆群体结构
在此基础上,进一步发掘不同SSR标记的“稀有”等位变异,并以遗传群体结构为基础,进一步结合每个大豆品种的农艺性状进行深入分析,发掘与重要农艺性状相关的优异等位变异,开展分子聚合设计育种。
4 结论
187对SSR引物平均每对引物扩增出多态性位点4.42个;平均多态性信息含量指数为0.406,平均基因多样性指数为0.45;187对引物可将202个黑龙江省栽培大豆品种全部区分;品种间的遗传相似系数为0.283~0.930,平均值为0.519;同一个育种单位育成的部分品种具有较高遗传相似性。202个大豆品种分为3个类群,类群中的部分材料血缘不是独立的而是相互渗透的。
参考文献
大豆基因组解析与重要农艺性状基因克隆研究进展
DOI:10.11983/CBB16087
URL
[本文引用: 1]

自2010年大豆(Glycine max)基因组论文在Nature杂志上正式发表以来,以中国为主的多国科学家已对众多不同地理来源的野生大豆、农家品种及栽培品种进行了基因组重测序,并通过比较基因组学等技术手段,揭示了大豆基因组在进化与驯化过程中的基本规律。利用大豆基因组信息,科学家已成功克隆了调控大豆生育期、生长(结荚)习性、种子形态性状与组分、结瘤与养分利用效率、生物及非生物胁迫抗(耐)性等重要农艺性状的功能基因,并初步揭示了其相关的作用机理,取得了一系列突破性进展,为今后进一步精细解析大豆基因组与相关性状的基因调控网络奠定了坚实的基础,同时为通过分子(模块)设计育种来培育新品种创造了必要条件。
黑龙江省1986-2000年大豆育种研究成就与展望
DOI:10.3969/j.issn.1000-9841.2004.02.012
URL
[本文引用: 1]

主要论述1986年-2000年黑龙江省大豆育种研究的成就和展 望.在此期间,各育种单位经黑龙江省农作物品种审定委员会审定推广108个大豆优良品种,其中黑龙江省农科院育成81个,占75%.育成品种在生育期、产 量、抗病虫性、品质等方面得到了提高和改善,育种技术得到了发展.并对育种存在主要问题和今后的发展进行了探讨.
中国大豆进口依赖性及其对大豆进口安全的影响
DOI:10.11861/j.issn.1000-9841.2015.03.0503
URL
[本文引用: 1]

为判断大豆进口来源的可靠性,增强进口安全性,通过综合考察我国从某国进口大豆量占我国进口大豆总量的比重、某国对我国出口大豆金额占该国出口大豆总金额的比重、某出口国的大豆产量占世界大豆产量的比重等指标,测算了我国大豆进口对主要来源国的依赖系数,并进一步评价了进口安全性。结果表明:我国大豆进口主要依赖美国和巴西,但对美国的依赖迅速减弱,系数从2000年的0.893 1降至2013年的0.180 8,而对巴西的依赖缓慢增强,系数从2000年的0.159 0上升至2013年的0.197 2。总的来看,大豆进口仍处于相对安全的状态。
Assessing the numbers of SNPs needed to establish molecular IDs and characterize the genetic diversity of soybean cultivars derived from Tokachi nagaha
DOI:10.1016/j.cj.2016.11.001
URL
[本文引用: 1]

The development of a core set of SNP molecular markers that could be widely used in soybean genetic research would greatly facilitate research into the genetic diversity of soybean. We conducted an analysis of Tokachi nagaha and 137 of its descendant soybean cultivars using 4044 SNP markers with the goal of determining the appropriate number of single-nucleotide polymorphisms (SNPs) needed to construct unambiguous molecular IDs and characterize genetic diversity based on a genetic distance matrix correlation method.When the number of SNPs was held constant, the number of accession pairs that could be distinguished increased as the polymorphism informative content (PIC) value of the SNPs increased. A core panel of 20 selected SNPs from 11 linkage groups with a mean PIC value of0.3703 and a range of 0.3640 0.3749 was able to identify almost all of the accession pairs in our study [9445 pairs (99.92%)]. The eight accession pairs that could not be identified with this core SNP set all originated from the same province and some of them had the same parental cultivars. The molecular IDs of the 138 accessions were constructed using the core 20 SNPs. It is known that both the number of SNPs and PIC values should be considered when SNPs are selected for use in the analysis of genetic diversity. In this study, when the PIC value was 0.3460, the correlation coefficient between the genetic distance matrices associated with a panel of 200 SNPs and the total population was >0.800, indicating satisfactory correlation. Our high-accuracy, high-resolution core SNP panel for germplasm fingerprinting and our findings about assessing genetic diversity will likely markedly improve the management and utilization efficiency of soybean germplasm resources.
Development of soybean aphid genomic SSR markers using next generation sequencing
DOI:10.1139/g11-002
URL
PMID:21529140
[本文引用: 1]

Abstract Simple sequence repeats (SSRs) or microsatellites are very useful molecular markers, owing to their locus-specific codominant and multiallelic nature, high abundance in the genome, and high rates of transferability across species. The soybean aphid (Aphis glycines Matsumura) has become the most damaging insect pest of soybean (Glycine max (L.) Merr.) in North America, since it was first found in the Midwest of the United States in 2000. Biotypes of the soybean aphid capable of colonizing newly developed aphid-resistant soybean cultivars have been recently discovered. Genetic resources, including molecular markers, to study soybean aphids are severely lacking. Recently developed next generation sequencing platforms offer opportunities for high-throughput and inexpensive genome sequencing and rapid marker development. The objectives of this study were (i) to develop and characterize genomic SSR markers from soybean aphid genomic sequences generated by next generation sequencing technology and (ii) to evaluate the utility of the SSRs for genetic diversity or relationship analyses. In total 128 SSR primer pairs were designed from sequences generated by Illumina GAII from a reduced representation library of A. glycines. Nearly 94% (120) of the primer pairs amplified SSR alleles of expected size and 24 SSR loci were polymorphic among three aphid samples from three populations. The polymorphic SSRs were successfully used to differentiate among 24 soybean aphids from Ohio and South Dakota. Sequencing of PCR products of two SSR markers from four aphid samples revealed that the allelic polymorphism was due to variation in the SSR repeats among the aphids. These markers should be particularly useful for genetic differentiation among aphids collected from soybean fields at different localities and regions. These SSR markers provide the soybean aphid research community with the first set of PCR-based codominant markers developed from the genomic sequences of A. glycines.
鲁麦14对山东新选育小麦品种的遗传贡献
DOI:10.3724/SP.J.1006.2012.00954
URL
Magsci
[本文引用: 1]

为了解骨干亲本鲁麦14对山东新选育小麦品种的遗传贡献,对鲁麦14及其6个衍生品种(系)进行了全基因组SSR扫描分析。在350个SSR位点上共检测到662个等位变异,每个位点1~5个等位变异,平均1.9个, 位点平均多态性指数(PIC)为0.21。UPGMA聚类分析表明,济麦22和鲁麦14聚为一类、青丰系列4个品种(系)聚为一类,这两类形成一个大的分支,而济麦20与这些品种的关系较远。在所检测的350个位点中,与鲁麦14相同的位点数,济麦22有235个(67.1%), 济麦20有210个(60.0%), 青丰1号有229个(65.4%), 青农2号有247个(70.6%)。这些相同位点多数以一个大的染色体区段传递至子代,且有的区段在6个衍生品种(系)中共享,如5A的gwm304–barc360–gwm415–barc1区段和6D的barc196–gdm127–barc123区段等。济麦22在21条染色体上都有与鲁麦14相同的位点,但染色体间差异较大,相同位点比例超过70%的染色体有3A、4A、7A,2B、4B、7B、1D、3D和4D;相同位点比例最低的是3B,仅46%。同一连锁群上,位点之间多呈连续区段分布,大多与已发现的重要性状分布簇相吻合。因此认为,鲁麦14的优良遗传背景对济麦22有重要贡献。在育种实践中,除需关注重要基因的导入外,还应注意骨干材料主体背景的选择。
中国栽培及野生大豆的遗传多样性、地理分化和演化关系研究
<p>栽培大豆的起源和演化是大豆生物学和农学基础研究的重要命题之一. 本研究选用60对细胞核SSR标记(nuSSR)和11对叶绿体SSR (cpSSR)标记, 在检测由393份地方品种和196份野生材料组成的全国代表性样本的细胞核、叶绿体基因组变异的基础上, 分析了栽培、野生地理生态群体间的遗传演化关系. 结果表明: (ⅰ) 野生群体核、质遗传多样性都明显大于栽培群体, 核、质等位变异数分别为1067:980和57:44个; 栽培大豆980个核等位变异中有377个(38.5%)为驯化后新生等位变异, 44个质等位变异中出现了7个(15.9%)新生等位变异. (ⅱ) 栽培生态类群中, 以南方3个地理生态类群(中南、华南、西南)遗传多样性较高; 野生生态类群中以长江中下游野生类群遗传多样性较高. (ⅲ) 从分子方差分析、遗传聚类与地理类群间关联分析及群体特有等位变异三方面证实我国栽培、野生大豆地理生态分化有其遗传分化的基础. (ⅳ) 以材料为单位的聚类结果表明与栽培大豆近缘的野生材料大多来自长江中下游及西南-中南野生地理类群; 进一步分析地理群体间的遗传距离, 并作UPGMA聚类, 发现各栽培地理类群与长江中下游野生大豆群体的遗传距离一致, 小于与包括本区在内的其他野生群体的距离, 结合该区野生群体特有的cpDNA等位变异NTCP10-117在所有栽培生态类群中都有分布的现象, 推论在南方野生群体中的长江中下游野生祖先可能是栽培大豆共同的野生祖先.</p>
黑龙江省大豆种质遗传结构及遗传多样性分析
DOI:10.3724/SP.J.1006.2009.00228
URL
Magsci
[本文引用: 1]

<p><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">利用</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">22</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">个表型性状和</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">60</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">个微卫星</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">(simple sequence repeat, SSR)</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">位点对黑龙江省</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">140</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">份代表性种质</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">(78</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">份地方品种和</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">62</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">份育成品种</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">)</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">进行分析</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">根据</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">UPGMA (unweighted pair group method with arithmetic mean)</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">和</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">Model-base</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">对</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">SSR</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">数据进行遗传结构划分。结果表明</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">参试品种可分为</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">2</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">大类群</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">第</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">II</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">类群的各项多样性指标均高于第</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">I</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">类群</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, 2</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">个类群遗传距离为</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">0.2427</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">;</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">PCO</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">结果显示这</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">2</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">个类群分布在不同区域</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">这与地理来源和育成年代密切有关。依据品种类型分为育成品种和地方品种两组</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">后者的各项多样性指标均高于前者</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">两组间的遗传距离为</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">0.1131</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">。依据表型数据的</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">PCO</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">分析表明</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">分布区域与品种类型有关</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">与</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">SSR</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">结构分类的结果吻合度低</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">两组品种主要在</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">3</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">个主成分的</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">6</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">个表型性状上有所不同。它们不是</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">2</span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">个相对独立的遗传群体</span><span lang="EN-US" style="font-size: 9pt; font-family: "Times New Roman"; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt; mso-fareast-font-family: 宋体">, </span><span style="font-size: 9pt; font-family: 宋体; mso-ascii-font-family: 'Times New Roman'; mso-hansi-font-family: 'Times New Roman'; mso-bidi-font-family: 'Times New Roman'; mso-font-kerning: 1.0pt; mso-ansi-language: EN-US; mso-fareast-language: ZH-CN; mso-bidi-language: AR-SA; mso-bidi-font-size: 10.5pt">根据分子标记和表型分类各有特点;建议在种质遗传多样性研究中将分子数据和表型数据结合起来。</span></p>
俄罗斯远东及黑龙江省大豆种质资源遗传多样性和群体结构分析
DOI:10.7505/j.issn.1007-9084.2017.01.004
URL
[本文引用: 1]

利用40对SSR引物对引自俄罗斯远东地区的大豆46份和来自黑龙江省的大豆44份共90份大豆种质资源进行遗传多样性和群体结构分析.结果表明,俄罗斯远东地区大豆遗传多样性高于黑龙江省大豆,UPGMA树状聚类和Structure遗传结构分析将90份材料分为3个类群,每个类群都包括不同地理来源的中、俄大豆,但主体明显:类群Ⅰ、类群Ⅱ主要为俄罗斯远东地区大豆资源,类群Ⅲ主要为黑龙江省大豆资源;中、俄大豆没有明显分开,表明俄罗斯远东地区和黑龙江省大豆种质个体间亲缘关系较近,拓宽黑龙江大豆的遗传基础还需从更大范围内引入俄罗斯种质.
黑龙江省大豆种质资源遗传多样性的ISSR分析
DOI:10.11861/j.issn.1000-9841.2011.01.0037
URL
[本文引用: 1]

采用ISSR分子标记技术对黑龙江省25个大豆品种进行遗传多样性分析。结果表明:从28对随机引物中筛选出9对多态性明显、条带清晰、反应稳定的引物;9对引物共扩增出65条带,其中58条为多态性条带,多态性比率为89.23%;Shannon多样性指数平均为0.4604,每个位点的有效等位基因数为1.5139;25个品种间的遗传相似性系数变幅范围为0.4462~0.8923,平均为0.6692;UPGMA聚类分析在GS=0.698处,将25份大豆材料分成5大类。
黑龙江省主栽大豆品种遗传多样性的SSR分析
DOI:10.3969/j.issn.1674-3466.2009.05.005
URL
Magsci
[本文引用: 3]

<P><FONT face=Verdana>使用合丰25等来自13个育种单位的13份大豆(<EM>Glycine max</EM>)品种对大豆20个连锁群上的100对SSR标记进行筛选, 最终保留了扩增稳定且多态性较高的43对SSR标记, 分析了黑龙江省83个主栽大豆品种的遗传多样性。结果表明, 在所有供试材料中共鉴定出等位变异157个, 每个位点2-7个, 平均为3.65个。品种间遗传相似系数为0.216-0.937, 平均为0.638 4, 表明黑龙江省大豆品种的遗传相似性较大, 故拓宽黑龙江省大豆品种的遗传基础具有重要意义。</FONT></P>
82份苜蓿种质资源的群体结构分析
种质资源遗传多样性和群体结构的分析是进行复杂性状关联分析的前提条件。本文从140对SSR引物中筛选到16对多态性引物,对82份苜蓿(Medicago spp.)材料进行产量性状差异的分析、遗传多样性与群体结构分析。结果如下: 1.在扬州地区,苜蓿一年刈割了3茬,生长天数分别为65天、40天、35天,所有参试材料都能够正常的生长。苜蓿在整个生长过程中冬季不枯黄,但停止生长。不同苜蓿品种(系)的初花期相差1天至10天。 2.苜蓿材料不同茬次的平均生长高度为第三茬第一茬第二茬,全年生长高度最高的是Creno,最矮的是CUF101。苜蓿种质资源不同茬次的干草产量为第一茬第三茬第二茬。全年草产量最高的是多仑、最低的是草原2号。 3.对苜蓿种质产量性状的特点分析表明,参试苜蓿品种(系)的遗传变异非常丰富。8个产量相关性状有较为丰富的变异,变异系数为第三茬干草产量分枝数第一茬干草产量第二茬干草产量第三茬刈割高度第二茬刈割高度第一茬刈割高度鲜干比。在82份苜蓿材料中变异系数最大的是多仑,为23.46,变异系数最小的是1075,为6.05。 4.筛选的16对SSR引物在82份苜蓿种质材料中共检测到190个等位变异,平均每对引物检测到11.88个,平均多态性比率为83.05%,范围从66.67-100%;平均基因多样性为0.2577,从0.1477-0.3637;平均多态信息量变化范围从0.0079-0.9432,平均为0.5501。 5.对82份苜蓿材料的群体结构进行分析,当K(类群数)=5时,似然值最大,因此将参试材料划分五大类群。以Q值=0.5为划分标准,由于82份苜蓿材料中的62份材料(占75.61%)在5个类群中的Q值大于或等于0.5,遗传基础比较单一,分到了某一个类群中:而其余的20份苜蓿材料(占24.39%)在每一类群中的Q值均小于0.5,遗传结构比较复杂,分到了混合群体中。划入特定类群中的62份材料,在各类群中占的比例分别为15.85%、8.54%、9.76%、26.27%、12.20%。苜蓿种质资源主成分分析与Structure遗传结构分析结果相一致。不同类群的遗传多样性比较结果显示,类群1遗传多样性水平最高,其次是类群2、类群3,类群4、类群5的遗传多样性水平相对较低。 6.不同类群的苜蓿种质资源生长差异很大,类群1产量高、分枝数多;类群2株高较矮,产量较低;类群3生长高度最矮;类群4植株生长高度高,分枝数少,鲜干比大,产量最低;类群5分枝数较少,鲜干比较小。不同类群的秋眠级类群5类群4类群3类群1类群2。
36份苜蓿种质材料遗传多样性的SSR分子标记
DOI:10.16742/j.zgcdxb.2016-02-04
URL
[本文引用: 1]

采用SSR分子标记方法,对36份不同苜蓿种质材料的基因组DNA进行扩增,并用6%的变性聚丙烯酰胺凝胶电泳对扩增结果进行了检测,结果表明:9对SSR引物共扩增出146个等位基因位点,扩增出的DNA片段大小在100-2000bp之间,其中多态位点72个,多态位点百分率43.57%;基因多样性指数平均值为0.1502,Shannon信息指数平均值为0.2047;多态信息量(PIC)平均值为0.8962。用Structure2.3软件对36份苜蓿材料进行结构分析,可将其划分为1个混合群体和4个特定类群,供试材料具有显著异质性,可用于苜蓿育种及优良基因挖掘利用。
分子标记与小麦群体遗传结构分析
DOI:10.3969/j.issn.1004-9398.2000.01.014
URL
[本文引用: 1]

小麦是我国主要的农作物之一,其遗传结构的研究,目前倍受分子生 物学家和小麦育种学家的高度重视.现代分子生物学的发展为小麦群体遗传结构的分析提供了一条更新更广阔的途径.本文论述了群体遗传结构研究方法的发展,着 重讨论几种主要的分子标记及其在小麦群体遗传结构分析中的应用.
大豆资源抗灰斑病1号生理小种优异等位变异的发掘
大豆灰斑病(Cercospora sojina Hara)是大豆生产区常发生的通过空气传播的真菌性病害。病害发生对大豆的经济产量影响很大。合理选育和种植抗病品种是大豆灰斑病综合防治中最有效、最经济的措施。对大豆资源进行抗病性鉴定,旨在发现优异抗病资源并发挖掘抗病基因优异等位变异,以实现优势资源抗病基因聚合。本研究以96个不同区域不同时期审定的大豆品种和68个野生大豆资源为试验材料,接种大豆灰斑病菌1号生理小种,建立一个非结构性和无亲缘关系的自然群体,进行抗性鉴定,并用178个SSR标记对164份大豆材料进行基因组扫描,采用STRUCTURE2.2对大豆群体进行群体结构分析,利用TASSEL软件的GLM方法对SSR标记和灰斑病1号生理小种的抗性进行关联分析,在P<0.05时,可认为标记与性状的关联显著,参考Breseghello等提出的无效等位变异(null allele)方法,计算等位变异的表型效应,发掘优异等位变异和相应的载体材料。主要结论如下:1、大豆育成品种资源鉴定结果:在96份育成品种中,鉴定出抗病品种16种,占供试验品种的16.67%;鉴定出中抗品种54种,占供试验品种的56.25%;鉴定出感病品种26种,占供试验品种的27.08%。2、野生大豆资源鉴定结果:在68份野生大豆资源中,鉴定出抗性资源16份,占参加试验资源的23.53%;鉴定出中抗资源31份,占供试验资源的45.59%;鉴定出感病资源21种,占供试验资源的30.88%。3、用STRUCTURE2.2对大豆群体进行结构分析,将164份大豆材料分为7个亚群,野生品种和育成品种血缘间相互独立;育成品种之间血缘相互融合,野生品种间血缘也不同程度相互融合。4、共检测到5个与抗性性状相关联的SSR分子标记,分别位于4、6、11、13和19号染色体上,引物分别为AW277661、satt363、satt430、satt656、satt652,其中贡献率最高的是satt363,值为59.71%。5、对5个SSR位点进行抗灰斑病1号生理小种抗病性等位变异的表型效应值分析,对抗性有增效作用的等位变异有28个,其中satt430效应值最大的是satt430-193,值为6.1714.09,典型载体材料九丰4号;AW277661效应值最大的是AW277661-177,值为10.59,典型载体材料野生大豆12C6277;satt363效应值最大的是satt363-214,值为7.33,典型载体材料野生大豆12NC1804;satt656效应值最大的是satt656-241,值为14.09,典型载体材料野生大豆12C8592;satt652效应值最大的是satt652-187,值为7.12,典型载体材料野生大豆12C6277;
大豆主要农艺性状的QTL分析
DOI:10.3969/j.issn.1004-8421.2017.01.023
URL
[本文引用: 1]

作为我国北方农业生产过程中最为重要的油料作物,大豆的种植质量以及产量都对我国民生事业的发展有一定的影响。因此,需要加强相关部门以及人员对于大豆种植的各种农艺性状的分析,从而促进我国的大豆种植的发展。本文基于此对大豆主要农艺性状进行QTL分析。
6个水稻杂交组合与其亲本的SSR标记多态性及其应用
DOI:10.3321/j.issn:1000-2030.2005.04.001
URL
[本文引用: 1]

选用分布于水稻12条染色体上的40对SSR引物对6个杂交水稻组合及其亲本的幼苗进行了SSR等位基因多态性分析.结果表明,位点RM224能够鉴别所有试验品种的F1及其亲本;Ⅱ优084和丰两优1号的F1及其双亲各有3对SSR引物可以区分;两优培九F1及其双亲有4对SSR引物可以区分.用位点RM224的引物对1份人为掺杂的两优培九F1样品进行了鉴定,试验结果与样本混合比例结果一致.
水稻耐盐碱胁迫优异等位变异的发掘
本研究以281个水稻品种构成的自然群体为试验材料,调查了盐、碱胁迫下水稻种子萌发能力相关性状,选择260对SSR标记对基因组进行扫描,采用Structure软件进行群体分析,利用GLM(General Linear Model)方法对标记与幼苗耐盐碱能力进行关联分析;并以试验材料表型均值为对照,鉴别出携带优异等位变异及载体材料。检测到15个与盐胁迫下种子萌发能力关联的SSR位点,其中第10条染色体上分别与标记RM184和RM171连锁位点的贡献率较大,达到29.51%和31.67%,优异等位变异RM184-211表型效应值最大为0.31,载体材料为‘滇屯502选早’。检测到与碱胁迫下种子萌发能力相关联的20个SSR位点,其中第3染色体与RM168和第4染色体与RM6314连锁位点的贡献率较大,分别达到39.17%和50.02%,优异等位变异RM6314-179表型效应值最大为0.58,载体材料为‘越6(N202)’。
基于SSR标记的太湖流域粳稻地方品种遗传多样性研究
DOI:10.3864/j.issn.0578-1752.2008.11.051
URL
Magsci
[本文引用: 1]

<P><FONT face=Verdana>【目的】评价太湖流域粳稻地方品种的遗传多样性。【方法】利用58对SSR引物,对基于主要农艺性状构建的太湖流域粳稻地方品种核心种质库的122个品种进行DNA水平的多态性分析,并与15个现代育成品种做比较。【结果】(1)地方品种群体中53个SSR位点共检测到216个等位片段,每个多态性位点等位片段数的变化范围为2~7个,平均为4.08个;71.7%的SSR位点具有3个以上等位片段;53个位点PIC值的变化范围为0.031~0.773,平均为0.413;Nei’s基因多样性指数He为0.378;品种间遗传距离平均为0.419;12条染色体中,第5染色体平均等位片段数最多,第11染色体平均PIC值最大。(2)现代育成品种群体检测到的等位片段数、位点PIC值、Nei’s基因多样性指数和品种间遗传距离均比地方品种群体相应值小,地方品种的遗传多样性大于现代育成品种。(3)聚类分析显示,在遗传相似系数0.63处,地方品种和现代育成品种可以明确区分开来。【结论】太湖流域粳稻地方品种具有丰富的遗传多样性,且与现代育成品种有较大的遗传差异,可用以拓宽育成品种的遗传基础。<BR></FONT></P>
26份大豆品种(系)遗传多样性分析及指纹图谱构建
利用53对SSR引物对来自黑龙江、内蒙古和新疆的26份大豆材料进行遗传多样性进行分析。结果表明:53对引物在供试材料间具有稳定的多态性,供试材料共测出137个等位基因,每个位点平均2.58个等位基因。多态信息含量指数在0.074 0~0.701 2之间。根据以上结果采用非加权类平均法进行聚类分析:26份大豆品种(系)间遗传相似系数变异范围为0.64~0.92,在相似系数0.68处可将26份大豆品种(系)分为4大类群,从53对引物中挑选8对多态性好、扩增带型清晰的SSR引物构建了26份大豆品种(系)的指纹图谱。构建的指纹图谱能够将26大豆品种(系)逐一区分。研究结果不仅为供试大豆品种鉴定、开展知识产权保护提供了依据,还为这些品种今后在育种中的应用提供理论指导。
川渝地区地方和育成大豆品种SSR标记多样性分析
DOI:10.7505/j.issn.1007-9084.2018.03.003
URL
[本文引用: 1]

利用基本均匀分布于大豆20条染色体的135对SSR标记,对232份包括6个地方品种地域亚群和1个育成品种亚群进行全基因组扫描.结果表明:所有的标记都有多态性,所有检测到的位点都是纯合基因型,说明所选用品种高度纯合,每个标记存在2~4个等位变异,平均2.66个.亚群多态性信息含量变异范围0.2751~0.3165,整个群体为0.3208;亚群内Nei遗传距离变异范围0.3258~0.3594,整个群体为0.3711,说明川渝地区大豆遗传变异较小.亚群间的遗传一致度(GI≥0.8862)较高,遗传距离(GD≤0.1208)较小,地方品种亚群间遗传差异更小,育成品种亚群与自然地域亚群的遗传差异相对较大.亚群间基因分化系数(Fst)平均为0.072 2,基因流(Nm)平均为3.214,说明不同亚群之间存在一定的基因交流.主坐标分析表明第一、二和三主成分分别解释总变异的4.97%、3.54%和3.33%.来自同一区域的品种资源基本聚集在同一亚群,聚类分析同样表明同一自然地域亚群品种资源虽不能完全聚集到同一个遗传类群中,但具有一定的聚集效应,说明川渝大豆品种资源遗传变异与地理位置有一定的关系.分子方差分析表明亚群内变异占总变异的97%,亚群间变异仅占总变异的3%.Man-tel收敛分析表明地方品种自然地域亚群的遗传距离与所处的地理位置距离(纬度和海拔)呈显著的正相关关系(R2=0.722).川渝地区大豆种质资源群体遗传丰富度不高,当前的育成品种未蕴含本地区所有遗传变异.
基于SSR标记的四川大豆与引进大豆资源遗传多样性和群体结构分析
DOI:10.11861/j.issn.1000-9841.2017.05.0657
URL
[本文引用: 1]

利用均匀分布于20条染色体的53对SSR标记(每条染色体上2~5对),对190份大豆资源进行遗传差异检测,随后根据标记试验结果进行遗传多样性分析、聚类分析、PCA分析和群体结构分析。53对SSR标记共检测到159个等位变异,每个位点等位基因范围为2~6个,平均每个位点的等位基因为3个,有效等位基因数Nei为1.4744±0.2375,多态性信息含量PIC为0.3050±0.1056;Shannon-Weaver指数值为0.4762±0.12490这些参数显示了190份大豆资源异质程度不是很高,遗传多样性丰富程度一般,总体遗传多样性处于中等水平。UPGMA聚类分析结果显示190份大豆资源(群体1:P1)被分为3个大类,且四川审定大豆品种与野生大豆资源、国外引进资源亲缘关系较远,随后将四川审定大豆品种31份、国外资源13份和野生大豆资源8份共52份材料(群体2:P2)单独进行聚类分析,52份材料也被分成3个大类。群体Ⅰ和群体2分别在K=3,K:2时得到合理群体结构。群体1的3个亚群分别是:亚群Ⅰ由地域来源丰富的78份材料组成,不包含野生大豆资源;亚群Ⅱ59份材料中含7份野生大豆资源;亚群Ⅲ53份材料只包括1份野生大豆资源zy05292。群体2分成两个亚群:亚群Ⅰ26份材料中含24份四川审定大豆品种和2份国外资源;亚群Ⅱ包含了6份审定大豆品种。供试的190份大豆资源蕴含了比较丰富的遗传变异,显示了较高水平的基因多样性。群体结构不能严格地按照地域、来源国家的划分而区分,这一现象显示了大豆资源存在着广泛的基因交流。从分析结果来看,四川大豆资源的种质创新可以充分地利用国外引进资源与直立型野生大豆资源,进而丰富四川大豆的基因多样性。
绥农号大豆血缘关系分析
DOI:10.3969/j.issn.1002-2767.2004.04.003
URL
[本文引用: 1]

通过对21个绥农号大豆品种进行系谱分析,明确了采用当地早熟、 高产、优质种质资源作为骨干亲本,加入国外血缘,改进血缘狭窄,丰富遗传基础,是育种成功的关键所在;三交组合方式后代类型丰富,易于选出丰产性突出、熟 期适中、抗病性及适应性好的优良品种和材料;在育种过程中要注重创造亲本材料.
合丰号大豆品种系谱分析
DOI:10.3969/j.issn.1000-9841.2002.02.012
URL
[本文引用: 1]

合丰号大豆32个品种84.4%来源于满仓金、荆山朴、丰收6号×克山四粒黄、丰收1号×蓑衣领四个族系.其基因库源于33个祖先亲本.细胞核祖先亲本有33个,细胞质祖先亲本有8个.核遗传贡献率较大的祖先亲本为金元(18.7%)、四粒黄(18.3%)、白眉(6.9%)、十胜长叶(5.9%)、小粒豆九号(5.6%)等.细胞质遗传贡献率最大的是四粒黄(46.7%),其次是小粒豆九号(16.7%)、白眉(13.3%).合丰号大豆直接利用亲本36个.利用国内优良亲本克4430-20、合交8009-1612等成功地育成了合丰25号、合丰35号等高产稳产大豆品种;利用国外抗病品种Ohio、Rampage、Wilkin直接作亲本育成高抗灰斑病品种4个.合丰号大豆品种有16个具有国外血缘,占品种总数的50%.随着年代的推移,合丰号大豆的细胞核、细胞质祖先亲本发生了变化.不同年代有其不同的主要贡献者,合丰号大豆遗传多样性是其成功的关键.制定一个祖先品种核质在新品种中达到新的更高水平协调的育种方案非常重要.
中国大豆品种生态区域划分的研究
DOI:10.3321/j.issn:0578-1752.2001.02.005
URL
[本文引用: 1]

在前人关于中国大豆栽培区域划分的基础上,根据我国各地256份大豆地方品种在南京分季播种、延长或缩短光照长度各处理条件下的生育期表现,结合供试材料的来源地地理与气候条件、播种季节类型、熟期组归属以及光温反应特性等因素,将我国大豆品种生态区划分为北方一熟制春作大豆品种生态区、黄淮海二熟制春夏作大豆品种生态区、长江中下游二熟制春夏作大豆品种生态区、中南多熟制春夏秋作大豆品种生态区、西南高原二熟制春夏作大豆品种生态区和华南热带多熟制四季大豆品种生态区等6大区,并在其中3个区内进一步划分亚区。