锌指蛋白[1]广泛存在于真核生物中,是一类具有“指状”结构域的蛋白,富含组氨酸和半胱氨酸[2]。保守的组氨酸和半胱氨酸残基通过与锌离子结合来稳定锌指结构域的空间结构[3]。基于锌结合氨基酸的排列特征,锌指蛋白可分为不同类型,如PHD(Plant Homeodomain Finger)、RING(Really Interesting New Genes)、LIM(Lin11,Isl-1 and Mec-3)等[4,5]。PHD结构域是由约60个氨基酸[C-X2-C-X(8-25)-C-X(2-4)-C-X(4-5)-H-X2-C-X(12-32)-C-X(2-3)-C]组成的保守基序(Cys4-His-Cys3),与RING(Cys3-His-Cys4)和LIM(Cys2-His-Cys5)高度相似[6,7,8]。
1 材料与方法
1.1 大豆PHD家族蛋白序列的搜索与鉴定
为了全面并准确鉴定大豆PHD家族基因,本研究采用3种方法预测大豆PHD家族成员。利用植物基因组数据库Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html),以“PHD-finger”为关键词进行搜索;利用拟南芥PHD家族蛋白序列,通过BLASTP比对搜索Phytozome大豆蛋白质组;利用拟南芥PHD家族蛋白构建隐马尔可夫模型,利用HMMER 3.0软件搜索大豆蛋白质组。对上述3种方法获得的候选序列,人工去除冗余序列,并利用SMART(http://smart.embl-heidelberg.de/)和Pfam(http:sanger.ac.uk/)进行分析,剔除不含完整PHD结构域的序列,最终确定大豆PHD家族成员。从Phytozome数据库获得大豆PHD家族基因的基本信息,包括基因登录号、染色体位置、基因长度、DNA、mRNA、编码序列和蛋白序列等。利用ExPASy数据库(http://www.expasy.org/)获取其分子量和等电点。利用PSORT1(http://psort1.hgc.jp/form.html)预测其亚细胞定位。
1.2 大豆PHD家族基因生物信息学分析
利用Rcircos本地软件[22]绘制大豆PHD家族基因在染色体上的位置图。通过植物基因组复制数据库PGDD(http://chibba.agtec.uga.edu/duplication/)获得基因复制信息共线性分析数据(Ka和Ks数值),利用Excel软件计算Ka/Ks,并利用Ka/Ks和Ka数据绘制散点图。利用MEGA 7软件内置的MUSCLE程序进行氨基酸序列比对,采用邻接法(Neighbor-Joining)构建系统进化树。其中,Bootstrap method值设为1 000,其余参数为默认。利用GSDS软件(http://gsds.cbi.pku.edu.cn/)绘制外显子-内含子结构图。大豆PHD家族成员功能结构域信息从Phytozome上获取。利用大豆PHD家族的蛋白序列,通过Blast2go[23]蛋白序列比对进行GO功能注释和聚类分析(参数为默认)。
1.3 大豆PHD家族基因表达特征分析
从Phytozome数据库获取大豆PHD家族基因在不同组织中的表达数据。苏打盐碱胁迫转录组测序数据由东北农业大学朱延明教授提供。高盐和干旱胁迫转录组数据从NCBI获取(GEO登录号:GSE57252)。提取PHD家族基因在不同组织及不同逆境胁迫处理的表达数据,利用Excel 2013软件结合Adobe illustrator CS6绘制热图。苏打盐碱、高盐、干旱胁迫处理,以0h为对照,|Log2变化倍数|>1为差异表达基因。
2 结果与分析
2.1 大豆PHD家族成员全基因组鉴定
本研究利用HMMER比对、BlastP比对及关键词(PHD-finger)搜索大豆蛋白质组,结合PFAM和SMART软件识别C4HC3基序(C-X2-C-X(8-25)-C-X(2-4)-C-X(4-5)-H-X2-C-X(12-32)-C-X(2-3)-C),最终获得95个大豆PHD家族成员。除Wei等[20]鉴定的6个大豆PHD家族基因GmPHD1-6外,将其余基因按照染色体位置分别命名为GmPHD7-GmPHD95(图1)。大豆PHD家族成员DNA序列在1 314~29 236bp不等,蛋白序列氨基酸数在132~2 335,分子量介于14.76~259.27kDa,等电点分布在4.76~9.96。由此可见,大豆PHD家族成员结构及理化性质差异较大,暗示进化过程中存在较大的分化。
图1
图1
大豆PHD家族蛋白PHD结构域氨基酸序列比对
Fig.1
Sequence alignment of the PHD domains within soybean PHD family proteins
2.2 大豆PHD家族基因染色体定位及复制分析
染色体定位结果显示,GmPHD95定位在第一个scaffold上;其余94个GmPHDs不均匀地分布在20条染色体上(图2)。此外,这些PHD家族基因在染色体上分布位置也不规则,例如1、5、13、18、20号染色体上PHD家族基因多分布在臂的下端,而6、16号染色体上的基因多分布在上端。
图2
图2
大豆PHD家族基因染色体定位和基因复制分析
Fig.2
Chromosome location and gene duplication of soybean PHD family genes
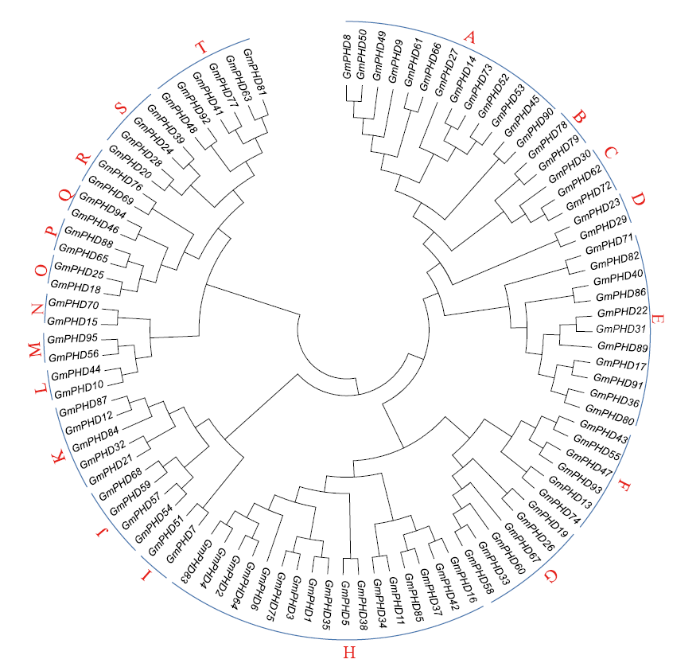
2.3 大豆PHD家族基因进化关系及基因结构分析
图3
图3
大豆PHD家族基因进化关系分析
Fig.3
Phylogenetic relationship of the soybean PHD family genes
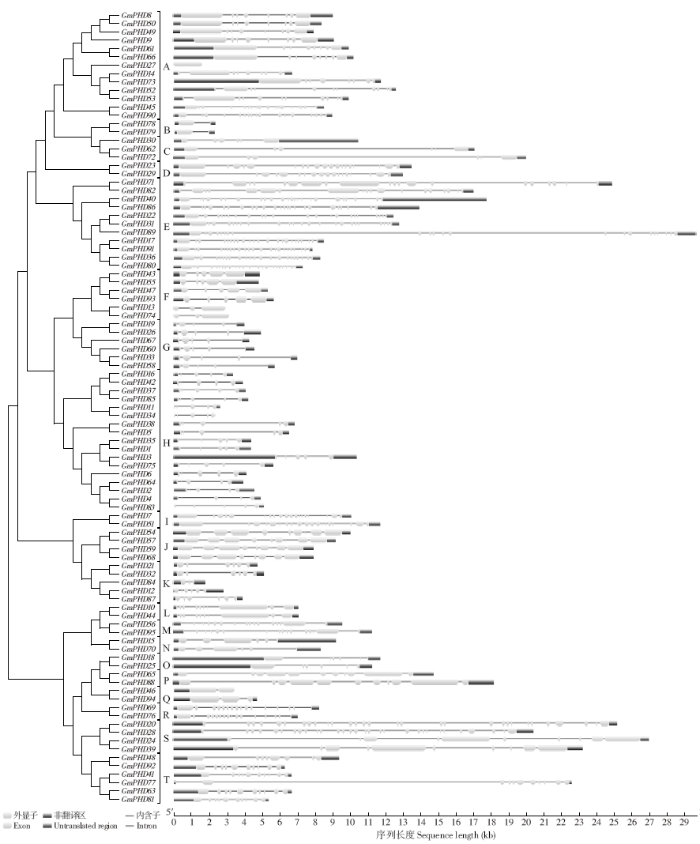
如图4所示,大豆PHD家族基因内含子数目差异较大,从0(GmPHD27)到23(GmPHD40/86)。同一亚家族内含子数目较为保守,且具有相似的内含子-外显子分布模式。不同亚家族间PHD家族基因内含子数和基因结构组成模式差异较大。存在复制关系的PHD家族基因对在结构上也具有较大的相似性。
图4
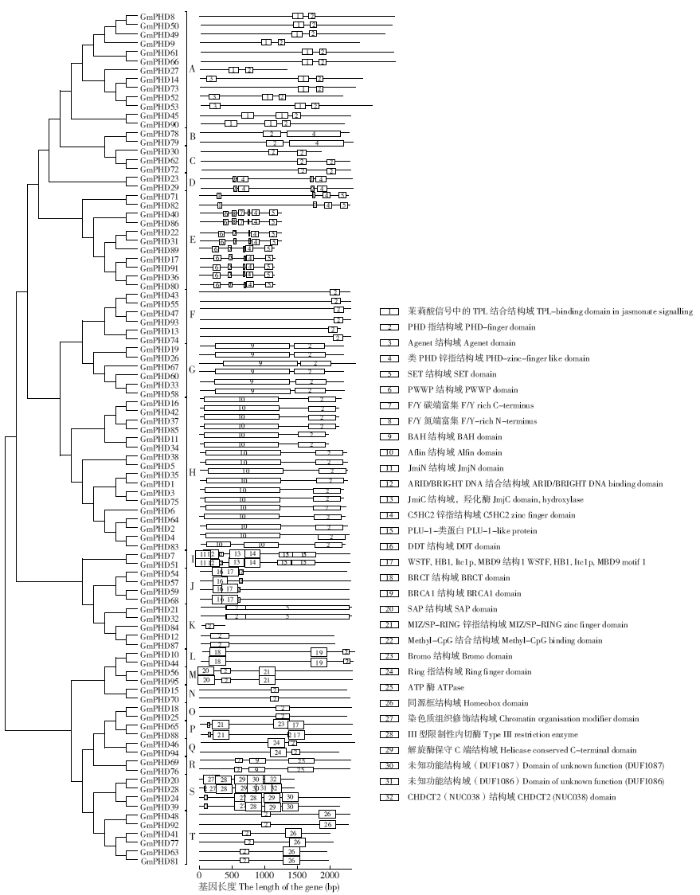
2.4 大豆PHD家族蛋白保守结构域分析
保守功能结构域分析显示,所有大豆PHD家族蛋白均含有至少一个PHD结构域(图5)。此外,亚家族B、D、E还含有1个类PHD结构域,亚家族Q中还含有1个RING结构域。总体来看,同一亚家族中的PHD成员有着相似的结构域,而不同亚家族间的结构域组成差异较大,暗示不同亚家族PHD成员可能存在较大的功能差异。
图5
图5
大豆PHD家族蛋白保守结构域分析
Fig.5
Conserved domains of the soybean PHD family proteins
亚家族H中所有成员均含有1个Alfin结构域(PF12165),该结构域被报道参与盐胁迫应答[25,26]。亚家族A中的所有成员均有1个Jas结构域(PF16135),该结构域参与茉莉酸信号传导,在植物抗病反应中发挥重要作用[27]。亚家族G和R的成员均含有BAH结构域(PF01426),该结构域能够介导蛋白-蛋白相互作用,与DNA甲基化和基因沉默相关[28,29]。亚家族E所有成员及亚家族K中PHD21/32均含有一个SET结构域(PF00856),该结构域可以调控基因转录和染色质结构,并参与植物生长调节[30,31];此外亚家族E还包含一个PWWP结构域(PF00855),其能够介导蛋白-蛋白相互作用[32,33]。这些结果表明,大豆PHD家族基因可能参与基因表达的表观遗传调控、植物生长发育、逆境应答等多种生物学过程,且不同亚家族间功能结构域的多样性暗示PHD家族基因可能存在功能多样性。
2.5 大豆PHD家族蛋白GO功能富集分析
GO功能富集分析(图6)显示,在分子功能类别中,ion binding和DNA binding被明显富集,这与PHD家族蛋白Zn2+结合和DNA结合特性有关;与表观遗传调控相关的分子功能也被富集,说明PHD家族基因可能参与表观遗传调控。细胞成分分类结果显示,多数PHD家族蛋白为细胞核相关组分,如细胞核、染色体、核质等,表明大豆PHD家族蛋白可作为核定位的转录因子发挥作用。生物学过程分类显示大豆PHD家族基因参与28种不同的生物学过程,这与上述功能结构域的多样性相一致,说明大豆PHD家族基因可能存在功能分化。其中,多数PHD成员主要参与生物合成和细胞氮化合物代谢过程。另外,组蛋白甲基化和染色体组装等与表观遗传调控相关的生物过程也得到富集。
图6
2.6 大豆PHD家族基因组织表达模式分析
从组织表达数据中可以看出,所有PHD家族基因至少在一个组织中表达(图7)。根据PHD成员在不同组织中的表达特性,可将其分为7组(Group I-VII)。Group I中的PHD家族基因在不同组织中表达均非常低(最大表达值<2.0,图7A)。Group II PHD成员虽然表达水平较低,但表现出明显的组织特异性,如GmPHD34在豆荚中高表达,GmPHD25在花中高表达,而GmPHD12和GmPHD78在根中高表达(图7B)。Group III PHD成员在不同组织中的最大表达值介于2.0~6.0,且没有组织特异性(图7C)。Group IV PHD成员在叶和花中的表达量较高(图7D);而Group V PHD成员在豆荚中高表达(图7E)。Group VI PHD家族基因表达量较高,无组织特异性(图7F);Group VII PHD成员表达水平最高,且至少在其中1个组织中表达值大于20(图7G)。大豆PHD家族基因的不同组织表达模式暗示其可能参与调控不同的生长发育过程。
图7
图7
大豆PHD家族基因组织表达特性分析
Fig.7
Tissue specific expression of the soybean PHD family genes
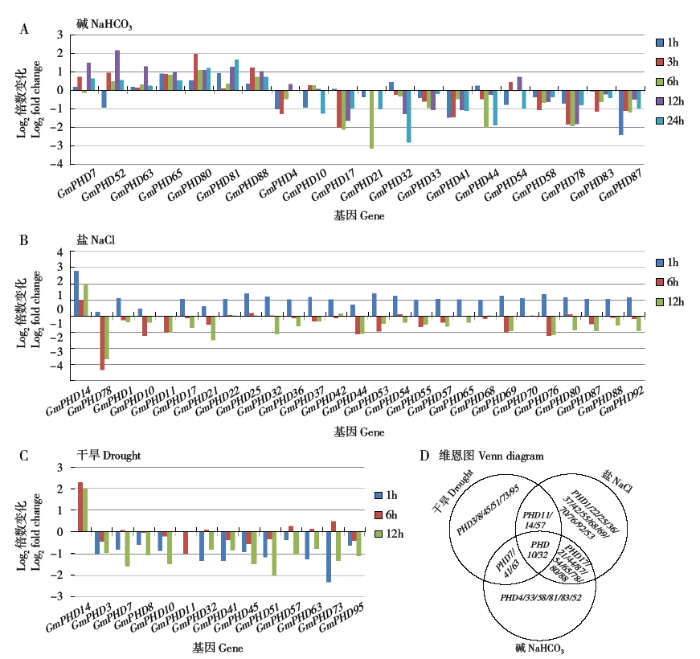
2.7 大豆PHD家族基因逆境胁迫表达模式分析
利用转录组数据分析了大豆PHD家族基因在苏打碱(NaHCO3)、高盐(NaCl)以及干旱(Drought)胁迫下的表达特点。如图8所示,95个大豆PHD家族中有27个基因表达响应高盐胁迫,有20个响应苏打碱胁迫,仅有14个响应干旱胁迫。进一步分析不同胁迫下差异表达PHD家族基因发现,苏打碱胁迫处理有7个基因上调表达,13个下调表达(图8A)。其中GmPHD52上调表达倍数最大(Log2变化倍数>2),GmPHD21下调表达最大(Log2变化倍数<-3)。高盐胁迫下多数差异表达PHD家族基因呈现1h上调、6和12h下调表达的趋势(图8B)。其中GmPHD14基因是唯一一个高盐胁迫处理不同时间点均上调表达的基因(Log2变化倍数>2),GmPHD1则呈现明显的下调表达(Log2变化倍数<-3)。PHD家族基因在苏打碱和高盐胁迫下不同的表达模式再次表明PHD家族基因可能通过不同的途径响应这两种胁迫。此外,干旱胁迫处理,除GmPHD14基因明显上调外,其他PHD家族基因均明显下调(图8C)。
图8
图8
大豆PHD家族基因在逆境胁迫下的表达模式分析
Fig.8
Expression profiles of soybean PHD family genes under different environmental stresses
对3种胁迫处理的共差异表达基因进行分析,发现有17个PHD家族基因在两种胁迫下显示差异表达(图8D),其中GmPHD10/32在3种胁迫下均差异表达。有8个PHD家族基因同时响应苏打碱和高盐胁迫。GmPHD14基因在高盐和干旱胁迫下均表现出显著的上调表达,暗示其可能参与植物对高盐和干旱胁迫的应答。
3 讨论
PHD家族蛋白在植物生长发育和逆境应答中发挥重要作用。虽然近年来被鉴定的PHD家族基因数量在不断增加,但绝大多数PHD家族基因及其生物学功能仍然未知。尽管已有研究工作者报道了6个大豆PHD家族基因(PHD1-6),但迄今尚未对大豆PHD家族成员进行全面、系统地综合分析。本研究利用生物信息学方法对鉴定出的95个大豆PHD家族基因进行了系统的分析。与其他相对保守的基因家族不同,植物PHD家族成员的进化相对多样化[9,10]。首先,系统进化树显示大豆PHD家族成员被分为了20个亚家族。前期研究也显示拟南芥、玉米、杨树PHD家族成员分成了17、10、11个亚族[10-11,15]。其次,不同PHD亚家族在蛋白序列长度、基因结构、功能结构域等方面也存在较大差异,与前人的研究[10,11]相一致,进一步表明植物PHD家族成员的多样性。
植物PHD家族进化及功能结构域的多样性表明,不同亚家族的PHD可能存在功能分化。本研究进一步通过GO功能分类、组织及逆境胁迫表达特征分析,证实了大豆PHD家族蛋白参与不同的分子和生物学功能。GO分子功能及细胞过程结果表明,大豆PHD家族蛋白可能通过2种方式发挥功能:和其他锌指蛋白一样,能够结合DNA,作为转录因子发挥功能;或通过修饰组蛋白或染色质结构,参与表观遗传调控。
此外,本研究还发现大豆PHD家族基因也参与植物对苏打碱、高盐和干旱胁迫的应答过程。苏打碱胁迫处理PHD家族基因多呈现下调表达,且胁迫处理后期差异表达基因数较多,而高盐胁迫处理1h有22个基因表现出上调表达。这一差异说明PHD家族基因可能通过不同的途径响应苏打碱和高盐胁迫。本研究也发现有17个PHD家族基因在两种胁迫下显示差异表达,其中GmPHD10/32在3种胁迫下均差异表达,GmPHD14在高盐和干旱胁迫下均表现出明显的上调表达。下一步将着重研究这几个PHD家族基因是否能够调控大豆对逆境胁迫的耐性和应答过程。
综上所述,大豆PHD家族蛋白结构的多样性表明其可能具有功能分化。目前植物PHD家族蛋白的调控机制仍知之甚少。本研究对大豆PHD家族基因的系统分析可为进一步研究大豆PHD家族成员的蛋白结构,在Zn2+结合、DNA结合和表观遗传调控中的分子功能,以及在植物生长发育和逆境应答中的生物学功能提供参考。
参考文献
大豆C2H2型锌指蛋白基因SCTF-1转化及功能分析
DOI:10.7505/j.issn.1007-9084.2016.03.006
Magsci
[本文引用: 1]

<p>为探讨锌指蛋白在大豆对非生物逆境胁迫耐受过程中的作用,本研究构建了锌指蛋白基因SCTF-1的植物表达载体pCPB-SCTF-1,利用花粉管通道法将其导入大豆中,通过抗性筛选及PCR检测,共获得了6株转SCTF-1基因植株,Southern杂交鉴定表明功能元件以单拷贝形式整合于受体基因组中。荧光定量PCR检测证明转化植株在根、茎、叶部位的表达与未转化植株相比显著提高。在低温胁迫下,转SCTF-1基因植株相对电导率明显低于非转化植株,降低了21.10%~23.09%;丙二醛含量明显低于非转化植株,降低了10.56%~11.74%;过氧化物酶活性明显高于非转化植株,增加了25.02%~30.38%;转基因植株叶片未呈现明显的萎缩、萎蔫、打卷现象。因此,转SCTF-1基因的过量表达提高了转基因大豆的耐冷能力。</p>
Genome-wide identification of C2H2 zinc-finger gene family in rice and their phylogeny and expression analysis
DOI:10.1007/s11103-007-9199-y URL [本文引用: 1]
Multiple modes of RNA recognition by zinc finger proteins
DOI:10.1016/j.sbi.2005.04.004 URL [本文引用: 1]
Genome-wide analysis of LIM gene family in Populus trichocarpa,Arabidopsis thaliana,and Oryza sativa
DOI:10.1093/dnares/dsm013 URL [本文引用: 1]
Zinc-finger transcription factors in plants
DOI:10.1007/s000180050186 URL [本文引用: 1]
The PHD finger:Implications for chromatin-mediated transcriptional regulation
DOI:10.1016/S0968-0004(00)88957-4 URL [本文引用: 1]
The ring finger domain:A recent example of a sequence-structure family
DOI:10.1016/S0959-440X(96)80060-1 URL [本文引用: 1]
The polybasic region that follows the plant homeodomain zinc finger 1 of Pf1 is necessary and sufficient for specific phosphoinositide binding
DOI:10.1074/jbc.M605624200 URL [本文引用: 1]
水稻与拟南芥PHD-finger蛋白的系统分析
PHD-finger (plant homeodomain finger)是具Cys<SUB>4</SUB>-His-Cys<SUB>3</SUB>特征结构的锌指蛋白结构域,它广泛存在于植物或动物转录调节蛋白中。此类蛋白在植物或动物蛋白质组中具有多个功能各异的家族成员,并在发育中扮演着重要的角色。鉴定了水稻44个推定的非冗余PHD-finger蛋白,并与拟南芥蛋白质组中45个该家族成员进行了比较;所构建的2棵进化树涵盖了这89个蛋白质,暗示水稻、拟南芥与人的PHD-finger蛋白中存在共同祖先。
Systematic analysis of the maize PHD-finger gene family reveals a subfamily involved in abiotic stress response
DOI:10.3390/ijms161023517 URL [本文引用: 8]
Genome-wide identification,classification and expression analysis of the PHD-finger protein family in Populus trichocarpa
DOI:10.1016/j.gene.2015.08.042 URL [本文引用: 7]
Genome-wide identification of the PHD-finger family genes and their responses to environmental stresses in Oryza sativa L
DOI:10.3390/ijms18092005 URL [本文引用: 2]
Genome-wide identification and analysis of genes encoding PHD-finger protein in tomato
Genome-wide analysis of PHD family transcription factors in carrot (Daucus carota L.) reveals evolution and response to abiotic stress
Germostatin resistance locus 1 encodes a PHD finger protein involved in auxin-mediated seed dormancy and germination
DOI:10.1111/tpj.13086 URL [本文引用: 2]
The binding specificity of the PHD-finger domain of VIN3 moderates vernalization response
DOI:10.1104/pp.16.01320 URL [本文引用: 1]
A barley PHD finger transcription factor that confers male sterility by affecting tapetal development
DOI:10.1111/pbi.2014.12.issue-6 URL [本文引用: 1]
Chromatin-dependent repression of the Arabidopsis floral integrator genes involves plant specific PHD-containing proteins
DOI:10.1105/tpc.114.130781 URL [本文引用: 1]
Soybean GmPHD-type transcription regulators improve stress tolerance in transgenic Arabidopsis plants
DOI:10.1371/journal.pone.0007209 URL [本文引用: 5]
GmPHD5 acts as an important regulator for crosstalk between histone H3K4 di-methylation and H3K14 acetylation in response to salinity stress in soybean
DOI:10.1186/1471-2229-11-178 URL [本文引用: 1]
RCircos:an R package for Circos 2D track plots
DOI:10.1186/1471-2105-14-244 URL [本文引用: 1]
Genome sequence of the palaeopolyploid soybean
DOI:10.1038/nature08670 [本文引用: 2]
Alfin1 transcription factor overexpression enhances plant root growth under normal and saline conditions and improves salt tolerance in alfalfa
DOI:10.1007/PL00008150 URL [本文引用: 1]
Transgenic overexpression of the transcription factor alfin1 enhances expression of the endogenous MsPRP2 gene in alfalfa and improves salinity tolerance of the plants
DOI:10.1104/pp.120.2.473 URL [本文引用: 1]
Hormone crosstalk in plant disease and defense:more than just jasmonate-salicylate antagonism
DOI:10.1146/annurev-phyto-073009-114447 URL [本文引用: 1]
The BAH (bromo-adjacent homology) domain:a link between DNA methylation,replication and transcriptional regulation
DOI:10.1016/S0014-5793(99)00132-5 URL [本文引用: 1]
Crystal structure of the proximal BAH domain of the polybromo protein
DOI:10.1042/BJ20050310 URL [本文引用: 1]
The Arabidopsis thaliana genome contains at least 29 active genes encoding SET domain proteins that can be assigned to four evolutionarily conserved classes
DOI:10.1093/nar/29.21.4319 URL [本文引用: 1]
Association of SET domain and myotubularin-related proteins modulates growth control
DOI:10.1038/ng0498-331 [本文引用: 1]
The PWWP domain:a potential protein-protein interaction domain in nuclear proteins influencing differentiation
DOI:10.1016/S0014-5793(00)01449-6 URL [本文引用: 1]
Structure and function of the nucleosome-binding PWWP domain
DOI:10.1016/j.tibs.2014.09.001 URL [本文引用: 1]
Arabidopsis AL PHD-PRC1 complexes promote seed germination through H3K4me3-to-H3K27me3 chromatin state switch in repression of seed developmental genes
DOI:10.1371/journal.pgen.1004091 URL [本文引用: 1]