稻瘟病菌(Magnaporthe oryzea B. Couch)引起的稻瘟病是世界水稻生产中最具毁灭性的病害之一[1,2],目前,控制该病害最为经济有效并且对环境安全的策略是种植抗病品种。黑龙江省是中国北方粳稻主产区,种植面积占东北地区总面积的58.1%[3],由于抗病品种的缺失,使稻瘟病的危害越来越重。稻瘟病系统是一个经典的基因对基因系统[4],其中致病菌中的无毒基因(AVR)在功能上与水稻中的特定抗病基因(R)相对应。只有在稻瘟病菌的无毒基因与寄主的抗性基因相互作用时,水稻品种才能表现出抗瘟性[5,6]。近年来,在水稻抗瘟基因与稻瘟病菌无毒基因研究方面已经取得了重要进展,据不完全统计,目前已鉴定的无毒基因有40多个[7],其中PWL2、PWL1、AVR-Pita、ACE1、AVR1-CO39、AvrPiz-t、AVR-Pia、AVR-Pii、AVR-Pik/kp/km、AVR-Pi9、PWL2D和AVR-Pib等12个基因被克隆[8,9,10,11,12,13,14,15,16,17]。张琦峰[18]、李祥晓[19]和张科等[20]先后分析了黑龙江省水稻部分无毒基因的组成。
本研究通过PCR扩增分析了黑龙江省2017年稻瘟病菌无毒基因存在与分布特点,为水稻抗病种质资源筛选提供参考依据。
1 材料与方法
1.1 供试菌株
2017年在黑龙江省7个市16个县水稻种植区采集稻瘟病穗颈瘟标样,经单孢分离获得单孢菌株208个。
1.2 稻瘟病菌基因组DNA的提取
将分离纯化的稻瘟病菌单孢菌株在PDA固体培养基上活化培养,挑取适量菌丝块接种到酵母液体培养基中,于28℃、120r/min摇床振荡培养3~5d,收集菌丝体,分装于1.5mL离心管中。使用真菌DNA提取试剂盒(D3390-01 Omega Bio-Tek公司)提取稻瘟病菌基因组DNA,使用微量分光光度计测定DNA浓度,并将DNA原液稀释成60ng/µL的工作液备用。原液于-20℃冰箱保存。
1.3 引物设计
根据NCBI中公布的无毒基因ACE1、AVR-Pi9、AVR-Pia、AVR-Pii和AVR1-CO39序列,使用Primer Premier 5.0设计5对特异性引物,所有引物均委托生工生物工程(上海)股份有限公司合成,引物序列见表1。
表1 用于扩增稻瘟病菌无毒基因的引物
Table 1
| 无毒基因 Avirulence gene | 引物序列 (5′-3′) Primer sequence (5′-3′) | 目的片段长度 Length of targeted fragment (bp) |
|---|---|---|
| ACE1 | F: GTGGAATCTTGTGGTGCTACGG R: ACGGGAACATCGACGGAAAG | 1 057 |
| AVR-Pi9 | F: CGGCAGGTGGGTTTATCATC R: CCAAAGCAGACAGCGACTCTA | 1 070 |
| AVR-Pia | F: TAGCCATTTCGGTCTCAT R: CGGGTAAACCCTGTCATA | 1 597 |
| AVR-Pii | F: TATGCAGGCCCAAATCCGTAG R: CCCGCAATAGTCCATAACTGTCC | 526 |
| AVR1-CO39 | F: CCTTGCCGCTACTGAAAC R: CCCTATCGCAGAGGAACA | 419 |
1.4 PCR扩增及电泳检测
PCR反应体系(20µL): rTaq酶0.1µL、10×Buffer(Mg2+)2.0µL、dNTP Mixture 1.6µL、正反向引物各0.3µL和DNA模板1µL,加ddH2O补足20µL。扩增程序: 94℃预变性4min;94℃变性45s,55℃~60℃退火45s(具体温度根据引物的GC含量设定),72℃延伸30~90s(具体时间根据扩增片段大小确定),30个循环;72℃延伸10min,4℃保存。扩增产物在1%琼脂糖凝胶中150V电泳20min,电泳液为0.5×TBE,在凝胶电泳成像系统下观察并拍照,统计无毒基因出现频率。
2 结果与分析
2.1 不同无毒基因的出现频率
表2 2017年黑龙江省稻瘟病菌无毒基因的扩增频率
Table 2
| 无毒基因 Avirulent gene | 出现个数 Number of occurrences | 出现频率 Occurrence frequency (%) |
|---|---|---|
| ACE1 | 183 | 87.98 |
| AVR-Pi9 | 166 | 79.81 |
| AVR-Pia | 34 | 16.35 |
| AVR-Pii | 1 | 0.48 |
| AVR1-CO39 | 0 | 0.00 |
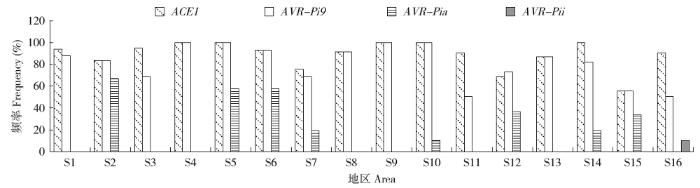
2.2 不同稻区各无毒基因的出现频率比较
由表3可知,4个无毒基因在黑龙江省各稻区存在频率具有明显的地理差别。在哈尔滨通河,无毒基因ACE1的出现频率为75.00%、AVR-Pi9为68.75%、AVR-Pia为18.75%、AVR-Pii和AVR1-CO39的出现频率均为0;而在鸡西密山,ACE1的出现频率为90.00%,AVR-Pi9为50.00%,AVR-Pii为10.00%,AVR-Pia和AVR1-CO39的出现频率均为0。以上结果显示,各稻区不同无毒基因的出现频率有一定的差异,且有些稻区差异较明显。
表3 黑龙江省不同地区无毒基因分布情况
Table 3
| 序号 No. | 地区Area | 总数 Total | ACE1 | AVR-Pi9 | AVR-Pia | AVR-Pii | AVR1-CO39 | |||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 数目 Number | 频率(%) Frequency | 数目 Number | 频率(%) Frequency | 数目 Number | 频率(%) Frequency | 数目 Number | 频率(%) Frequency | 数目 Number | 频率(%) Frequency | |||
| S1 | 齐齐哈尔甘南 | 16 | 15 | 93.75 | 14 | 87.50 | 0 | 0.00 | 0 | 0.00 | 0 | 0 |
| S2 | 齐齐哈尔哈拉海农场 | 6 | 5 | 83.33 | 5 | 83.33 | 4 | 66.67 | 0 | 0.00 | 0 | 0 |
| S3 | 大庆杜蒙 | 19 | 18 | 94.74 | 13 | 68.42 | 0 | 0.00 | 0 | 0.00 | 0 | 0 |
| S4 | 大庆肇源 | 11 | 11 | 100.00 | 11 | 100.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0 |
| S5 | 绥化望奎 | 7 | 7 | 100.00 | 7 | 100.00 | 4 | 57.14 | 0 | 0.00 | 0 | 0 |
| S6 | 绥化庆安 | 14 | 13 | 92.86 | 13 | 92.86 | 8 | 57.14 | 0 | 0.00 | 0 | 0 |
| S7 | 哈尔滨通河 | 16 | 12 | 75.00 | 11 | 68.75 | 3 | 18.75 | 0 | 0.00 | 0 | 0 |
| S8 | 哈尔滨依兰 | 23 | 21 | 91.30 | 21 | 91.30 | 0 | 0.00 | 0 | 0.00 | 0 | 0 |
| S9 | 哈尔滨方正 | 4 | 4 | 100.00 | 4 | 100.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0 |
| S10 | 哈尔滨尚志 | 10 | 10 | 100.00 | 10 | 100.00 | 1 | 10.00 | 0 | 0.00 | 0 | 0 |
| S11 | 佳木斯桦川 | 10 | 9 | 90.00 | 5 | 50.00 | 0 | 0.00 | 0 | 0.00 | 0 | 0 |
| S12 | 佳木斯汤原 | 22 | 15 | 68.18 | 16 | 72.73 | 8 | 36.36 | 0 | 0.00 | 0 | 0 |
| S13 | 佳木斯桦南 | 15 | 13 | 86.67 | 13 | 86.67 | 0 | 0.00 | 0 | 0.00 | 0 | 0 |
| S14 | 鹤岗绥滨 | 16 | 16 | 100.00 | 13 | 81.25 | 3 | 18.75 | 0 | 0.00 | 0 | 0 |
| S15 | 鸡西虎林 | 9 | 5 | 55.56 | 5 | 55.56 | 3 | 33.33 | 0 | 0.00 | 0 | 0 |
| S16 | 鸡西密山 | 10 | 9 | 90.00 | 5 | 50.00 | 0 | 0.00 | 1 | 10.00 | 0 | 0 |
2.3 同一无毒基因在不同稻区的出现频率比较
图1
图1
各无毒基因在黑龙江省不同地区出现频率比较
Fig.1
The occurrence frequency of each avirulence gene in different areas of Heilongjiang Province
3 讨论
探索无毒基因在自然菌株群体中的分布,为抗病品种的布局及病情的分子预警提供参考依据。本试验根据无毒基因ACE1、AVR-Pi9、AVR-Pia、AVR-Pii和AVR1-CO39设计的5对特异性引物对采自黑龙江省2017年208个菌株DNA进行PCR扩增检测。结果显示,与抗性基因Pi-33和Pi-9对应的无毒基因ACE1与AVR-Pi9出现频率均在70.00%以上,且在各稻区均有分布,根据基因对基因假说[4],在品种选育时可以选择携带Pi-33与Pi-9的水稻品种。
李祥晓[19]报道2009年黑龙江省稻瘟病菌菌株中无毒基因AVR1-CO39、AVR-Pita、ACE1、AVR-Pik、AvrPiz-t、AVR-Pia和AVR-Pit均有分布且各地区差异显著,ACE1引物的扩增频率最高, ACE1出现频率为61.6%,AVR-Pia出现频率为42.9%,AVR1-CO39出现频率为31.6%;张科等[20]在2015年采用11对稻瘟病菌的特异性引物扩增黑龙江省124个稻瘟病菌的菌株,结果显示,在黑龙江省无毒基因ACE1、AVR-Pia和AVR1-CO39的出现频率分别为64.5%、43.4%和35.4%。本试验检测到2017年黑龙江省无毒基因ACE1、AVR-Pia和AVR1-CO39的出现频率分别为87.98%、16.35%和0。可以看出,在黑龙江省随着年份的增长,基因ACE1出现频率逐渐升高,而AVR-Pia和AVR1-CO39出现频率呈下降趋势。王世维等[21]报道2012年辽宁省稻瘟病菌无毒基因AVR1-CO39和AVR-Pii的出现频率都为0,在吉林省也未检测到这2个无毒基因,推测在东北地区无毒基因AVR1-CO39和AVR-Pii的出现频率较低。
4 结论
黑龙江省稻瘟病菌群体组成错综复杂,5个无毒基因的存在和完全缺失与地理分布密切相关,所以应及时检测田间稻瘟病菌无毒基因的组成与变化,来指导田间抗性基因的合理布局和抗病品种的定期轮换。由黑龙江省稻瘟病菌无菌基因检测结果可知,抗性基因Pi-33与Pi-9在当地抗病育种中利用价值最大。
参考文献
A multilocus gene genealogy concordant with host preference indicates segregation of a new species,Magnaporthe oryzae,from M. grisea
DOI:10.1080/15572536.2003.11833196 URL [本文引用: 1]
Current status of the gene-for-gene concept
DOI:10.1146/annurev.py.09.090171.001423 URL [本文引用: 2]
Tissue-adapted invasion strategies of the rice blast fungus Magnaporthe oryzae
DOI:10.1105/tpc.110.078048 URL [本文引用: 1]
Multiple plant surface signals are sensed by different mechanisms in the rice blast fungus for appressorium formation
DOI:10.1371/journal.ppat.1001261 URL [本文引用: 1]
Identification and fine mapping of AvrPi15,a novel avirulence gene of Magnaporthe grisea
DOI:10.1007/s00122-006-0347-6
URL
[本文引用: 1]

Avirulence of Magnaporthe grisea isolate CHL346 on rice cultivar GA25 was studied with 242 ascospore progenies derived from the cross CHL346 × CHL42. Segregation analysis of the avirulence in the progeny population was in agreement with the existence of a single avirulence (Avr) gene, designated as AvrPi15. For mapping the Avr gene, we developed a total of 121 microsatellite DNA markers [simple sequence repeat (SSR)], which evenly distributed in the whole-genome of M. grisea through bioinformatics analysis (BIA) using the publicly available sequence. Linkage analysis of the AvrPi15 gene with these SSR markers showed that six markers on chromosome 6, MS6-1, MS6-2, MS6-3, MS6-7, MS6-8 and MS6-10, were linked to the AvrPi15 locus. To further define the chromosomal location of the AvrPi15 locus, two additional markers, MS6-17 and STS6-6, which were developed based on the sequences of telomeric region 11 (TEL11), were subjected to linkage analysis. The results showed that MS6-17 and STS6-6 were associated with the locus by 3.3 and 0.8 cM, respectively. To finely map the Avr gene, two additional candidate avirulence gene (CAG) markers, CAG6-1 and CAG6-2, were developed based on the gene annotation of the sequence of TEL 11. Linkage analysis of the Avr gene with these two markers revealed that both of them completely cosegregated with the AvrPi15 locus. Finally, this locus was physically mapped into ∼ 7.2-kb interval of the TEL11 by BIA using these sequence-ready markers. This is the key step toward positional cloning of the AvrPi15 gene.]]>
Identification,cloning,and characterization of PWL2,a gene for host species specificity in the rice blast fungus
The PWL host specificity gene family in the blast fungus Magnaporthe grisea
DOI:10.1094/MPMI-8-0939 URL [本文引用: 1]
A telomeric avirulence gene determines efficacy for the rice blast resistance gene Pi-ta
DOI:10.1105/tpc.12.11.2019 URL [本文引用: 1]
Magnaporthe grisea avirulence gene ACE1 belongs to an infection-specific gene cluster involved in secondary metabolism
DOI:10.1111/nph.2008.179.issue-1 URL [本文引用: 1]
Chromosome walking to the AVR1-CO39 avirulence gene of Magnaporthe grisea: Discrepancy between the physical and genetic maps
The Magnaporthe oryzae avirulence gene AvrPiz-t encodes a predicted secreted protein that triggers the immunity in rice mediated by the blast resistance gene Piz-t
DOI:10.1094/MPMI-22-4-0411 URL [本文引用: 1]
Association genetics reveals three novel avirulence genes from the rice blast fungal pathogen Magnaporthe oryzae
DOI:10.1105/tpc.109.066324 URL [本文引用: 1]
Comparative genomics identifies the Magnaporthe oryzae avirulence effector AVR-Pi9 that triggers Pi9-mediated blast resistance in rice
DOI:10.1111/nph.13310 URL [本文引用: 1]
Overexpression and purification of PWL2D,a mutant of the effector protein PWL2 from Magnaporthe grisea
DOI:10.1016/j.pep.2010.04.020 URL [本文引用: 1]
Function and evolution of Magnaporthe oryzae avirulence gene AVR-Pib responding to the rice blast resistance gene Pib
DOI:10.1038/srep11642 URL [本文引用: 1]
辽宁省稻瘟病菌无毒基因型鉴定及分析
DOI:10.3864/j.issn.0578-1752.2014.03.006
URL
[本文引用: 1]

【目的】鉴定稻瘟病菌(Magnaporthe oryzae)的无毒基因型,了解无毒基因在不同地区流行菌株中的分布情况,为品种布局提供参考。【方法】根据已经克隆且与稻瘟病菌致病性相关的6个无毒基因序列设计引物,选取辽宁省稻瘟病常发区的26株稻瘟病菌单孢菌株,提取各菌株DNA样本作为模板,进行PCR扩增。通过琼脂糖凝胶电泳及PCR产物测序,对6个无毒基因PCR产物进行碱基和氨基酸序列的分析比较。对琼脂糖凝胶电泳未出条带的,设计不同引物进行验证性试验。【结果】在PCR产物电泳检测中,Avr1-CO39、Avr-pia和Avr-pii没有产物条带,对这3个无毒基因设计验证引物,其PCR产物电泳检测仍没有条带出现,其结果证明辽宁省各水稻主产区流行稻瘟病菌中多不携带Avr1-CO39、Avr-pia和Avr-pii;对于其他3个无毒基因 AvrPiz-t、Avr-pik和Avr-pita则有特异性扩增产物,说明这3个无毒基因在各稻区稻瘟病菌中以不同突变类型及不同频率出现。其中,与Pi2、Pi9和Piz-t对应的AvrPiz-t,分别在22个菌株的中被检测到,且有21个菌株的序列与其序列一致,说明该基因遗传相对稳定,也间接证明携带Pi2、Pi9和Piz-t 的水稻品种在辽宁地区的广谱抗性;与基因组序列不同的16号菌株,在DNA序列192 bp处发生一个单碱基C的缺失,从而导致移码突变,且碱基突变导致氨基酸序列至72位氨基酸时提前终止,而使该基因编码的蛋白质失去无毒基因的功能。与Pik、Pik-p、Pik-m和pik-s对应的Avr-pik,电泳检测结果表明各菌株均有特异性条带出现,经测序验证等位基因序列分为4种类型(B、D、F、G),其中12个菌株携带可为Pik或其等位基因Pik-m和pik-p所识别的D类型;9个菌株携带B类型,该等位基因曾被报道,但是否具有无毒基因的功能仍未验证;另有2个菌株携带F类型等位基因,该基因为首次发现,并分别出现在丹东和盘锦地区。其特点在于与D类型基因间存在143(A/G)的碱基差异,氨基酸序列翻译结果显示其为错义突变,即48(G/D);而其余3个菌株携带G类型等位基因,该基因亦属首次发现,且仅出现在抚顺新宾地区,碱基序列与D类型存在168(G/A)的差异,导致翻译提前终止,基因功能丧失。对于Avr-pita,26个菌株特异性扩增产物一致,但测序检测到5种等位基因类型,且均与Avr-pita有差异,碱基序列的变化多导致错义突变。这5种等位基因的氨基酸序列之间差异由3个氨基酸位点的差异所致,分别为83(D/N)、192(Y/C)和207(K/R),3处突变均在基因结构域范围内,几种等位基因均已见报道。【结论】辽宁稻区流行稻瘟病菌中Avr-pik、AvrPiz-t和Avr-pita分布较为广泛,选育及推广携带相应抗病基因的水稻品种可减轻稻瘟病的危害。