燕麦属(Avena L.)属于禾本科(Poaceae Barn.)早熟禾亚科(Pooideae Benth.)燕麦族(Aveneae Dumort.),主要分布在欧洲、非洲、亚洲、澳大利亚、美洲的温带和寒带地区。我国燕麦主要分布在华北、西北和西南高海拔等干旱、半干旱地区[1];其抗逆性强,对土壤的要求低,且耐盐、耐酸、耐冷、耐旱、耐瘠薄[2,3]。燕麦属植物具有重要经济价值,主要是作为粮食作物、保健食品、高级饲料和工业原料。在世界八大谷类作物中,燕麦属栽培种(简称栽培燕麦)总产量居第6位,仅次于小麦(Triticum aestivum)、水稻(Oryza sativa)、玉米(Zea mays)、大麦(Hordeum vulgare)和高粱(Sorghum bicolor)。栽培燕麦富含蛋白质、脂肪、碳水化合物、维生素和微量元素等多种营养成分,与人们日常食用的小麦、水稻、玉米、大麦和高粱等作物相比,其籽粒中蛋白质、脂肪、矿物元素、纤维素和维生素五大指标含量均居首位。此外,燕麦粉蛋白质的氨基酸组成比较平衡,其中赖氨酸的含量比小麦等粮食作物高1.5~3.0倍。带稃型栽培燕麦籽粒蛋白质、粗纤维、粗脂肪、钙、磷含量高,是马属动物的标准饲料。燕麦茎秆是高档纸的优质原料,稃壳可以提取糠醛,是重要的有机化工原料。
燕麦属是相对古老的作物属(x=7),关于其分类问题,最早由林奈于1753年按形态学特征命名了燕麦属的4个种:普通栽培燕麦(A. sativa)、野红燕麦(A. sterilis)、普通野燕麦(A. fatua)和裸燕麦(A. nuda);Malzev于1930年对燕麦进行了详尽的分类。Kihara(1924)依据细胞学对燕麦分类进行研究,发现3个不同的染色体数14、28和42,依此将研究的10个种分为3个种群:二倍体(2n=14)、四倍体(2n=28)和六倍体(2n=42)种群。董玉琛等[4]总结前人的结果,将燕麦属27个种按倍性水平分成3个种群,其中二倍体种12个,四倍体种8个,六倍体种7个;按野生种和栽培种分为22个野生种和5个栽培种。其中二倍体燕麦(A、C基因组)主要包括小粒裸燕麦(A. nudibrevis Vav.)、砂燕麦(A. strigosa Schreb.)、威氏燕麦或沙漠燕麦(A. wiestii Steud.)和短燕麦(A. brevis Roth.);四倍体燕麦(AB、AC基因组)主要包括细燕麦(A. barbata Pott.)和阿比西尼亚燕麦(A. abyssinica Hochst.);六倍体燕麦(ACD基因组)主要包括地中海燕麦(A. byzantina C. koch)、普通野燕麦(A. fatua L)、普通栽培燕麦(A. sativa L.)和野红燕麦(A. sterilis L.)[5]。通过染色体核型、RAPD、醇溶蛋白图谱研究证实,二倍体物种的A、C基因组是燕麦属中差异最显著的2个基因组类型[6]:A基因组包括Ap、Al、Ad、Ac和As 5个亚型,各亚型间的进化关系最复杂[7];C基因组包括Cm、Cp和Cv 3个亚型,是燕麦属进化速度最慢的基因组[8,9]。大量研究[10]表明B、D基因组和A基因组有较高的同源性,由于自然界尚未发现B、D基因组二倍体物种,而且B基因组只存在于四倍体物种,D基因组只存在于六倍体物种,导致有关燕麦的起源演化问题至今还未得出一致的结论。
核糖体DNA基因(ribosomal DNAs,rDNA)是真核生物中一类高度保守的基因家族,具有重要的生物学功能,成簇分布于1对或多对染色体上[11]。高等植物细胞核45S rDNA是串联重复序列,由18S rDNA-5.8S rDNA-28S rDNA和基因间的2个内转录间隔区(ITS1和ITS2)组成1个重复单元,其中ITS1-5.8S rDNA-ITS2称为ITS区。利用荧光原位杂交(FISH)的方法将45S rDNA在染色体上进行物理定位,不仅可以作为一种染色体标记对植物染色体进行分析和识别,而且可以分析染色体的结构变异和研究种属之间的进化关系等遗传学问题[12,13,14]。同时,在植物分子系统学研究中,由于ITS区较编码区进化速率更快,因此常用于较低分类阶元的系统学研究[15]。
1 材料和方法
1.1 供试材料
材料为偏凸燕麦(A. ventricosa,CvCv)、砂燕麦(A. strigosa,AsAs)、裂稃燕麦(A. barbata,AABB)、葡萄牙野燕麦(A. fatua var. portugal,AACCDD)、光稃野燕麦(A. fatua var. glabrata,AACCDD)和栽培燕麦16Co1、16Co2(A. sativa,AACCDD),由贵州大学农学院/国家小麦改良中心贵州分中心保存提供。普通小麦5S rDNA和45S rDNA由山东农业大学农学院提供。
1.2 试验方法
1.2.1 细胞学鉴定 种子在室温条件下浸泡吸胀后腹沟朝下均匀置于湿润的滤纸上,25℃培养32h以上,待根长2~3cm时取下根尖,经高纯度笑气(N2O)处理2h左右,90%乙酸固定5~10min后,转移到70%乙醇溶液中保存。将固定好的根尖转移到纤维素果胶酶中,37℃水浴55~60min后,进行压片,镜检,照相。
1.2.2 质粒DNA的提取 挑取单菌落于新鲜的LB液体培养基中,加抗生素(氨苄,50mg/L),在37℃、200转/min培养8~12h后,取部分菌液加甘油于-80℃保存备用,余下菌液用于提取质粒。从测序正确的大肠杆菌中提取质粒,方法参照全式金(北京,全式金生物技术有限公司)Easy PureTM Plasmid MiniPrep Kit试剂盒说明书进行。
1.2.3 FISH分析 将Kato等[13]的方法略作修改。采用缺口平移法,提取的5S rDNA和45S rDNA分别利用Texas red-5-dUTP和Fluorescein-12-dUCP进行标记。每张染色体制片加20μL杂交液,盖上盖玻片,55℃保湿下杂交过夜。杂交完成后,揭去盖玻片,室温下依次置于2×SSC、2×SSC和ddH2O中各5min,晾干,复染,盖上盖玻片,最后通过荧光显微镜(OLYMPUS BX-61,日本)进行观察分析,利用显微镜照相机(Nikon DS-Ri1,日本)进行图像采集。
1.2.4 45S rDNA序列的获取 在NCBI(
1.3 数据处理
2 结果与分析
2.1 燕麦属植物染色体细胞学鉴定
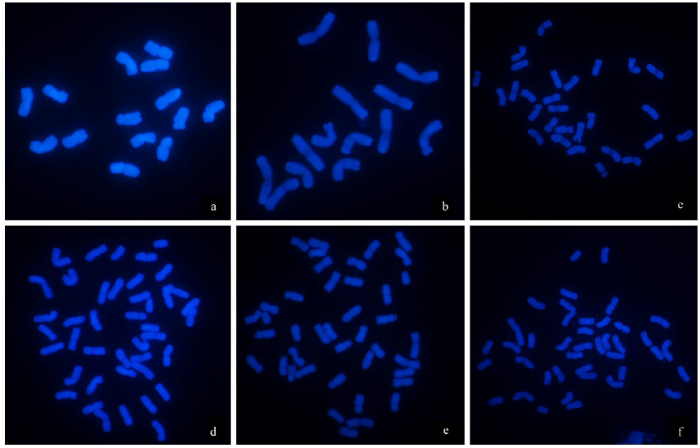
为获得FISH适宜的染色体分裂时期,对供试材料的有丝分裂染色体进行了普通细胞学鉴定。结果(图1)表明,所选根尖细胞染色体均处于有丝分裂中期,且偏凸燕麦、砂燕麦、裂稃燕麦、葡萄牙野燕麦、光稃野燕麦、栽培燕麦16Co1和16Co2的染色体条数分别为14、14、28、42、42、42、42条,符合各供试材料的倍性水平。
图1
图1
燕麦有丝分裂染色体
a:偏凸燕麦;b:砂燕麦;c:裂稃燕麦;d:葡萄牙野燕麦;e:光稃野燕麦;f:栽培燕麦16Co2
Fig.1
Mitotic chromosome in oat species
a: A. ventricosa; b: A. strigosa; c: A. barbata; d: A. fatua var. portugal; e: A. fatua var. glabrata; f: A. sativa 16Co2
2.2 5S rDNA和45S rDNA在燕麦属植物染色体的分布
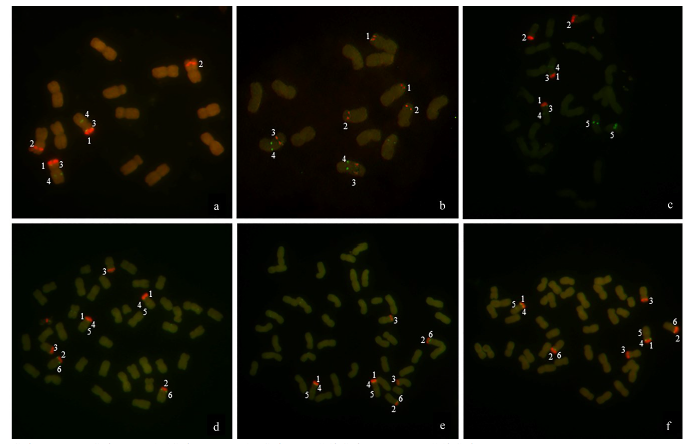
利用Texas red-5-dUTP标记的45S rDNA(红色)和Fluorescein-12-dUCP标记的5S rDNA(绿色)为探针对7份燕麦有丝分裂染色体进行FISH分析。
结果(表1和图2)表明,在砂燕麦和偏凸燕麦有丝分裂中期染色体上45S rDNA分别有2对和3对杂交信号,5S rDNA各有2对和1对杂交信号,这些5S rDNA杂交信号分别和1对45S rDNA杂交信号位于同一条染色体上(图2a~b)。5S rDNA和45S rDNA在裂稃燕麦分别有3对和2对杂交信号,其中2对5S rDNA位点和1对45S rDNA位点位于同一条染色体(图2c)。光稃野燕麦(图2d)和栽培燕麦(16Co1、16Co2)(图2e~f)有丝分裂中期染色体5S rDNA和45S rDNA杂交信号没有明显差异,各呈现3对杂交信号,其中2对染色体上含有5S rDNA和45S rDNA位点,1对染色体上只有45S rDNA位点。
表1 供试燕麦45S rDNA和5S rDNA信号信息
Table 1
| 物种 Species | 基因组 Genome | 染色体数目 Chromosome number | 位点 Signal | 位点数目 Number of signals | 信号分布区域 Signal region | 信号强弱 Signal lightness |
|---|---|---|---|---|---|---|
| 砂燕麦 A. strigosa | AsAs | 2n=2x=14 | 45S | 4 | 染色体端部 | 较强 |
| 5S | 4 | 1对位于染色体端部,1对位于染色体中部 | 较弱 | |||
| 偏凸燕麦 A. ventricosa | CvCv | 2n=2x=14 | 45S | 6 | 2对位于染色体端部,1对位于染色体中部 | 较弱 |
| 5S | 2 | 染色体中部 | 较弱 | |||
| 裂稃燕麦 A. barbata | AABB | 2n=4x=28 | 45S | 4 | 染色体端部 | 1强1弱 |
| 5S | 6 | 1对位于染色体端部,2对位于染色体中部 | 较弱 | |||
| 葡萄牙野燕麦 A. fatua var. portugal | AACCDD | 2n=6x=42 | 45S | 6 | 染色体端部 | 较强 |
| 5S | 6 | 2对位于染色体端部,1对位于染色体中部 | 较弱 | |||
| 光稃野燕麦 A. fatua var. glabrata | AACCDD | 2n=6x=42 | 45S | 6 | 染色体端部 | 较强 |
| 5S | 6 | 2对位于染色体端部,1对位于染色体中部 | 较弱 | |||
| 栽培燕麦16Co1 A. sativa 16Co1 | AACCDD | 2n=6x=42 | 45S | 6 | 染色体端部 | 较强 |
| 5S | 6 | 2对位于染色体端部,1对位于染色体中部 | 较弱 | |||
| 栽培燕麦16Co2 A. sativa 16Co2 | AACCDD | 2n=6x=42 | 45S | 6 | 染色体端部 | 较强 |
| 5S | 6 | 2对位于染色体端部,1对位于染色体中部 | 较弱 |
图2
图2
燕麦有丝分裂染色体45S rDNA和5S rDNA FISH鉴定
a:砂燕麦;b:偏凸燕麦;c:裂稃燕麦;d:光稃野燕麦;e:栽培燕麦16Co1;f:栽培燕麦16Co2
Fig.2
45S rDNA and 5S rDNA FISH identification of mitotic chromosomes in oat species
a: A. strigosa; b: A. ventricosa; c A. barbata; d: A. fatua var. glabrata; e: A. sativa (16Co1); f: A. sativa (16Co2)
2.3 45S rDNA ITS区DNA序列分析
以“internal transcribed spacer AVENA”为检索条件,在NCBI中检索获得341条燕麦属植物45S rDNA序列。删除小于400bp的序列,手动筛选含ITS1、5.8S rDNA和ITS2区域全长的DNA序列,共获得214条燕麦属植物45S rDNA ITS全长序列,涵盖燕麦属二倍体(AcAc、AdAd、AlAl、AsAs、CpCp、CvCv)、四倍体(CmCmCmCm、AABB、AACC)和六倍体(AACCDD)10种基因组类型。为后续分析方便,依据所在基因组和物种的不同,对每条序列进行重命名(表2)。所有DNA序列经CLUSTAL_X比对后,通过GeneDoc软件手工修订,保留保守序列,最终每条序列长度约600bp。
表2 公开的含45S rDNA ITS区DNA序列全长的燕麦属物种
Table 2
| 物种Species | 基因组Genome | 染色体数目Chromosome number | 序列数Sequence number | 序列名Sequence name |
|---|---|---|---|---|
| 加那利燕麦A. canariensis | AcAc | 2n=2x=14 | 5 | can1 Ac-can5 Ac |
| 大马士革燕麦A. damascena | AdAd | 2n=2x=14 | 9 | dam1 Ad-dam9 Ad |
| 长颖燕麦A. longiglumis | AlAl | 2n=2x=14 | 6 | lon1 Al-lon6 Al |
| 砂燕麦A. strigosa | AsAs | 2n=2x=14 | 3 | str1 As-str3 As |
| 沙漠燕麦A. wiestii | AsAs | 2n=2x=14 | 9 | wie1 As-wie9 As |
| 大西洋燕麦A. atlantica | AsAs | 2n=2x=14 | 6 | atl1 As-atl6 As |
| A. hirtula | AsAs | 2n=2x=14 | 6 | hir1 As-hir6 As |
| A. lusitanica | AsAs | 2n=2x=14 | 4 | lus1 As-lus4 As |
| 短燕麦A. brevis | AsAs | 2n=2x=14 | 3 | bre1 nA-sbre3 As |
| A. hispanica | AsAs | 2n=2x=14 | 4 | his1 As-his4 As |
| 异颖燕麦A. eriantha | CpCp | 2n=2x=14 | 9 | eri1 Cp-eri9 Cp |
| 物种Species | 基因组Genome | 染色体数目Chromosome number | 序列数Sequence number | 序列名Sequence name |
| 不完全燕麦A. clauda | CpCp | 2n=2x=14 | 9 | cla1 Cp-cla9 Cp |
| A. pilosa | CpCp | 2n=2x=14 | 1 | pil1 Cp |
| 偏凸燕麦A. ventricosa | CvCv | 2n=2x=14 | 5 | ven1 Cv-ven5 Cv |
| 裂稃燕麦A. barbata | AABB | 2n=4x=28 | 16 | bar1 AB-bar16 AB |
| 阿比西尼亚燕麦A. abyssinica | AABB | 2n=4x=28 | 9 | aby1 AB-aby9 AB |
| A. agadiriana | AABB | 2n=4x=28 | 7 | aga1 AB-aga7 AB |
| 瓦维洛夫燕麦A. vaviloviana | AABB | 2n=4x=28 | 7 | vav1 AB-vav7 AB |
| 岛屿燕麦A. insularis | AACC | 2n=4x=28 | 4 | ins1 AC-ins4 AC |
| 大燕麦A. maroccana | AACC | 2n=4x=28 | 9 | mar1 AC-mar9 AC |
| 墨菲燕麦A. murphyi | AACC | 2n=4x=28 | 6 | mur1 AC-mur6 AC |
| 大穗燕麦A. macrostachya | CmCmCmCm | 2n=4x=28 | 10 | mac1 CmCm-mac10 CmCm |
| 野燕麦A. fatua | AACCDD | 2n=6x=42 | 21 | fat1 ACD-fat21 ACD |
| 普通栽培燕麦A. sativa | AACCDD | 2n=6x=42 | 16 | sat1 ACD-sat16 ACD |
| 野红燕麦A. sterilis | AACCDD | 2n=6x=42 | 11 | ste1 ACD-ste11 ACD |
| A. occidentalis | AACCDD | 2n=6x=42 | 12 | occ1 ACD-occ12 ACD |
| 大粒裸燕麦A. nuda | AACCDD | 2n=6x=42 | 6 | nud1 ACD-nud6 ACD |
| 地中海燕麦A. byzantina | AACCDD | 2n=6x=42 | 1 | byz1 ACD |
2.4 系统进化分析
利用最大似然法构建小麦为外群的系统进化树(图3),这些序列依据基因组的差异,可以分为明显的4支。As基因组沙漠燕麦(A. wiestii)和大西洋燕麦(A. atlantica)的ITS DNA序列的聚在一起(蓝色),表明这些序列的“祖先”基因发生分化的时间较早。其余3个分支,C基因组(绿色)、AB基因组(橙色)和ACD基因组(红色)的大多数ITS DNA序列由于基因组的差异能够分别聚到一起,但各物种不同类型的ITS DNA序列在3个分支中均有分布,且存在于不同的进化支中,不存在单独分支,说明5个物种的进化模式不存在单独的特点,而是有同样的进化模式。
图3
图3
燕麦属植物与小麦45S rDNA序列系统进化树
Fig.3
Construction of 45S rDNA sequence phylogenetic tree of oat species and wheat
3 讨论
rDNA在染色体上的位点数目和分布特点可以反映出近缘种属物种之间的系统进化关系,也能有效地反映种属间的分化程度。本试验所用二倍体砂燕麦和偏凸燕麦基因组分别为AsAs和CvCv,裂稃燕麦基因组为AABB,葡萄牙野燕麦、光稃野燕麦和栽培燕麦基因组均为AACCDD。尽管自然界中尚未发现燕麦属物种B和D基因组的二倍体供体种,通过对7份燕麦的45S rDNA和5S rDNA位点在染色体上的定位和分布统计,可以推测AABB基因组中45S rDNA位点均来源于A基因组,含有5号杂交信号的染色体来源于B基因组(图2c);AACCDD基因组中,含1+4+5号杂交信号的染色体可能来源于A基因组,含2+6号杂交信号的染色体可能来源于D基因组,含有3号杂交信号的染色体可能来源于A基因组或C基因组(图2d~f)。虽然在物种的进化过程中,rDNA在染色体上的位点可能发生变化,但这依然可以为查找燕麦属B和D基因组的二倍体供体种提供参考。同时由于本试验分析的品种还较少,还需进一步的研究以提供更为详细的信息。
在植物进化过程中,多倍化是普遍的生物学现象。本研究中燕麦属物种含有二倍体、四倍体和六倍体3种不同的类型。在多倍化进程中45S rDNA位点数会随着染色体的加倍而相应加倍,二倍体、四倍体和六倍体燕麦属物种的45S rDNA位点数目应随着倍性的增加而增多。但本试验中,二倍体的砂燕麦和四倍体的裂稃燕麦均含有2对45S rDNA位点;六倍体的野燕麦和栽培燕麦含有3对45S rDNA位点。其45S rDNA位点数并没有随着染色体的加倍而等比例加倍,出现了拷贝数减少信号削弱或者位点数目丢失等情况,可能是由于植物多倍化之后的二倍化过程中,相对强势的45S rDNA会抑制较为弱势物种的45S rDNA基因的表达,导致非表达45S rDNA拷贝数减少,或同源配对和随后重组导致的染色体结构变异(如缺失、易位等)造成原始物种45S rDNA位点的丢失。在偃麦属[21,22]、冰草属[23]和人参属[24]中均发现类似的生物学现象。这些类似基因组冲击(genomic shock)的适应性变化对新形成多倍体物种基因组的进化和稳定发挥了重要的作用[25]。
45S rDNA ITS在核基因组中的重复单位通过染色体的不等交换和高频率的基因转换进行位点内和位点间的致同进化,使不同拷贝的序列趋于基本相近或完全一致。这种特性使ITS序列被广泛应用于被子植物系统发育研究[26]。燕麦属含有29个种,涉及二倍体、四倍体和六倍体种,包括A、B、C和D 4种基因组。二倍体燕麦为A和C基因组2种,四倍体为AB或AC基因组,六倍体燕麦则都为ACD基因组类型。其中B染色体组只存在于四倍体中,而D染色体组只存在于六倍体中。由于燕麦属植物基因组构成的复杂性,以及多倍体物种整个基因组的重复,致使燕麦属植物ITS序列致同进化更为复杂。有研究[27,28]表明,由于致同进化不能在不同亲本提供的重复单元之间发生,导致异源多倍体双亲ITS序列都得以保留,或由于定向致同进化导致只有1个亲本类型的ITS被固定。本研究中,利用以公开的45S rDNA ITS区DNA序列构建进化树,结果显示来源于相同基因组的大部分序列很好地聚集到一起,说明燕麦属多倍体45S rDNA ITS受到致同进化的影响,也说明燕麦属同一倍性的不同物种可能起源于几种相同的远源材料。
由于“近乎完美”的致同进化特点,使ITS区DNA序列为物种的进化提供了非常有用的系统发育信息。本研究中,ITS基因树分为明显的A基因组和C基因组分支。其中,除沙漠燕麦(A. wiestii)和大西洋燕麦(A. atlantica)的As基因组单独聚在一起外,含A基因组的不同多倍体燕麦属物种都以较高的支持率和A基因组二倍体聚在一起;而C基因组分支除了含有C基因组的二倍体外,还有六倍体野燕麦(A. fatua)的部分ITS区DNA序列拷贝类型。说明燕麦属物种的A基因组和C基因组具有明显差异,而且同一倍性的不同燕麦属物种其二倍体亲本来源可能不同。另外,所有六倍体ACD基因组和四倍体AC、AB基因组都聚合在A基因组分支内,没有发现B和D基因组的特异分支。这一结果虽然一定程度上反映了A、B和D基因组之间的一致性,但也可能是由于A、B和D基因组间较快的致同进化导致在含B和D基因组的燕麦属物种中找不到B和D基因组的分支。
4 结论
二倍体的砂燕麦和四倍体的裂稃燕麦均含有2对45S rDNA位点,二倍体的偏凸燕麦和六倍体的野燕麦和栽培燕麦含有3对45S rDNA位点。以“internal transcribed spacer AVENA”为检索条件,手动筛选获得214条燕麦属植物45S rDNA ITS全长序列。涵盖燕麦属二倍体(AcAc、AdAd、AlAl、AsAs、CpCp、CvCv)、四倍体(CmCmCmCm、AABB、AACC)和六倍体(AACCDD)10种基因组类型。利用最大似然法构建的ITS基因树分为明显的A和C基因组分支,没有发现B和D基因组的特异分支。
参考文献
Genome and species relationships in genus Avena based on RAPD and AFLP molecular markers
Cytogenetic analysis of diploid Avena L. species containing the as genome
Genomic configuration of the autotetraploid oat species avena macrostachya inferred from comparative analysis of ITS1 and ITS2 sequences:on the oat karyotype evolution during the early events of the Avena species divergence
On evolutionary pathways of Avena species
Evolutionary insights inferred by molecular analysis of the ITS1-5.8S-ITS2 and IGS Avena sp. sequences
Physical mapping,expression patterns and interphase organisation of rDNA loci in Portuguese endemic Silene cintrana and Silene rothmaleri
Chromosome painting using repetitive DNA sequences as probes for somatic chromosome identification in maize
Contrasting patterns of evolution of 45S and 5S rDNA families uncover new aspects in the genome constitution of the agronomically important grass Thinopyrum intermedium (Triticeae)
Chloroplast DNA and the study of plant phylogeny:present status and future prospects
//Soltis P S,Soltis D E,Doyle J J. Molecular Systematics of Plants.
Molecular diversity of the 5S rRNA gene and genomic relationships in the genus Avena (Poaceae:Aveneae)
Phylogenetic analysis of the genus Avena based on chloroplast intergenic spacer psb A-trn H and single-copy nuclear gene Acc1
Oat evolution revealed in the maternal lineages of 25 Avena species
The CLUSTAL_X Windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools
MEGA X:Molecular evolutionary genetics analysis across computing platforms
Molecular cytogenetic (FISH) and genome analysis of diploid wheatgrasses and their phylogenetic relationship
Contrasting patterns of evolution of 45S and 5S rDNA families uncover new aspects in the genome constitution of the agronomically important grass Thinopyrum intermedium (Triticeae)
FISH and GISH analysis of the genomic relationships among Panax species
Molecular phylogeny of Anagallis (Myrsinaceae) based on ITS, trn L-F,and ndh F sequence data
Phylogenetic relationships in subtribe Poinae (Poaceae,Poeae) based on nuclear ITS and plastid trn T-trn L-trn F sequences
Preliminary molecular phylogeny of Eurasian Scrophularia (Scrophulariaceae) based on DNA sequence data from trnS-trnG and ITS regions