玉米杂种优势类群的划分方法有系谱分析、表型聚类分析、配合力分析、同工酶法以及分子标记法。现在主要用分子标记法进行种质资源类群的划分,分子标记法包括SSR标记、SNP标记及芯片技术[4]。作为第三代分子标记的SNP标记,其优点为遗传稳定性好、分布密度高和可自动化分析等,因此SNP标记常被应用于玉米杂种优势类群划分与遗传多样性分析[5]。卢媛等[6]用34 257个SNP标记把44份糯玉米自交系分为5个类群,分别为P群、塘四平头群、旅大红骨群、Reid群和Lancaster群。高嵩等[7]利用6973个SNP位点将205份玉米自交系划分为7个类群,分别为Lancaster群、塘四平头群、旅大红骨群、Reid、PA、PB群和热带类群。姜思奇等[8]利用46 899个高质量的SNP标记将36份玉米自交系分为4个杂种优势群,分别为Lancaster群、Reid群、塘四平头与旅大红骨混合群和类PH4CV群。师亚琴等[9]利用4550个SNP位点将80份玉米自交系划分为7个类群,包括Reid、黄改群、P群、Lancaster、Iodent、迪卡选系和先锋改良群。Li等[10]和Wang等[11]对不同玉米杂种优势类群的基因组和表型分化的遗传基础进行了研究,从分子层面对玉米优势类群的机理进行解析,这对利用分子标记划分类群提供了理论支撑。
杂种优势类群划分结果的种类数量差异是由类群划分的标准不同造成的,类群划分结果均可在育种工作中作为参考。杂种优势类群划分的种类过多会导致杂种模式过于复杂,对育种工作会有所干扰而降低育种效率。目前在我国北方的生产应用中,育种家主要利用Reid、Non-Reid(以Lancaster为主)和Dom群(塘四平头群和旅大红骨类群)这三大优势类群的材料,对具有国外血缘的自交系可划分为Reid和Non-Reid两大杂种优势群,Dom群为国内种质,包括塘四平头群和旅大红骨类群[12]。本试验利用玉米60K SNP芯片鉴定100份玉米种质资源并进行遗传多样性分析,为品种选育和提高育种效率提供一定的借鉴。
1 材料与方法
1.1 试验材料
供试材料为黑龙江省农业科学院绥化分院提供的100份玉米资源,编号、名称及来源详见表1。其中以6个已知来源的骨干自交系作为测验种,其中SH060、SH075和SH083属于Reid群,SH085属于Non-Reid群,SH084和SH097属于Dom群。
表1 100份玉米种质资源
Table 1
| 编号Number | 名称Name | 来源Source | ||
|---|---|---|---|---|
| SH001 | KWS10/KWS73/ 欧早 | 黑龙江省农业科学院绥化分院 | ||
| SH002 | SX618/欧早 | 黑龙江省农业科学院绥化分院 | ||
| SH003 | S1029 | 黑龙江省农业科学院绥化分院 | ||
| SH004 | SI15412 | 黑龙江省农业科学院绥化分院 | ||
| SH005 | S1035 | 黑龙江省农业科学院绥化分院 | ||
| SH006 | S1037 | 黑龙江省农业科学院绥化分院 | ||
| SH007 | S1040 | 黑龙江省农业科学院绥化分院 | ||
| SH008 | SI15111 | 黑龙江省农业科学院绥化分院 | ||
| SH009 | SI15215 | 黑龙江省农业科学院绥化分院 | ||
| SH010 | LK702 | 黑龙江省龙科种业集团有限公司 | ||
| SH011 | LK708 | 黑龙江省龙科种业集团有限公司 | ||
| SH012 | LK725 | 黑龙江省龙科种业集团有限公司 | ||
| SH013 | LK727 | 黑龙江省龙科种业集团有限公司 | ||
| SH014 | LK729 | 黑龙江省龙科种业集团有限公司 | ||
| SH015 | LK738 | 黑龙江省龙科种业集团有限公司 | ||
| SH016 | SD3♀/欧早 | 黑龙江省农业科学院绥化分院 | ||
| SH017 | N3527 | 黑龙江省农业科学院绥化分院 | ||
| SH018 | N3528 | 黑龙江省农业科学院绥化分院 | ||
| SH019 | N3529-2 | 黑龙江省农业科学院绥化分院 | ||
| SH020 | N3532-1 | 黑龙江省农业科学院绥化分院 | ||
| SH021 | N3533-4 | 黑龙江省农业科学院绥化分院 | ||
| SH022 | N3535-1 | 黑龙江省农业科学院绥化分院 | ||
| SH023 | N3538-3 | 黑龙江省农业科学院绥化分院 | ||
| SH024 | N3540-2 | 黑龙江省农业科学院绥化分院 | ||
| SH025 | N3542-3 | 黑龙江省农业科学院绥化分院 | ||
| SH026 | N3543-2 | 黑龙江省农业科学院绥化分院 | ||
| SH027 | N3545-4 | 黑龙江省农业科学院绥化分院 | ||
| SH028 | N3546-3 | 黑龙江省农业科学院绥化分院 | ||
| SH029 | N3547-2 | 黑龙江省农业科学院绥化分院 | ||
| SH030 | N3547-4 | 黑龙江省农业科学院绥化分院 | ||
| SH031 | N3528-2 | 黑龙江省农业科学院绥化分院 | ||
| SH032 | N3540-3 | 黑龙江省农业科学院绥化分院 | ||
| SH033 | N3542-2 | 黑龙江省农业科学院绥化分院 | ||
| SH034 | N3543-2 | 黑龙江省农业科学院绥化分院 | ||
| SH035 | SYN12 | 黑龙江省农业科学院绥化分院 | ||
| SH036 | K3/郑58 | 黑龙江省农业科学院绥化分院 | ||
| SH037 | B73/郑58 | 黑龙江省农业科学院绥化分院 | ||
| SH038 | 30715-3 | 黑龙江省农业科学院绥化分院 | ||
| SH039 | 30716-4 | 黑龙江省农业科学院绥化分院 | ||
| SH040 | K3/341 | 黑龙江省农业科学院绥化分院 | ||
| SH041 | 8941/340 | 黑龙江省农业科学院绥化分院 | ||
| SH042 | 344/9229 | 黑龙江省农业科学院绥化分院 | ||
| SH043 | SDM7 | 黑龙江省农业科学院绥化分院 | ||
| SH044 | 加群 | 中国农业科学院作物科学研究所 | ||
| SH045 | 98-4/郑58 | 黑龙江省农业科学院绥化分院 | ||
| SH046 | 4207-5/C8605 | 黑龙江省农业科学院绥化分院 | ||
| SH047 | 9176/郑58 | 黑龙江省农业科学院绥化分院 | ||
| SH048 | 扎461/(330/ M67) | 黑龙江省农业科学院绥化分院 | ||
| SH049 | 30716 | 黑龙江省农业科学院绥化分院 | ||
| SH050 | 15D86 | 吉林省宏宇种业有限公司 | ||
| SH051 | 15D90 | 吉林省宏宇种业有限公司 | ||
| SH052 | W08 | 吉林省宏宇种业有限公司 | ||
| SH053 | 93622 | 黑龙江省农业科学院绥化分院 | ||
| SH054 | 15D98 | 吉林省宏宇种业有限公司 | ||
| SH055 | 15D109 | 吉林省宏宇种业有限公司 | ||
| SH056 | 6171/SX711 | 黑龙江省农业科学院绥化分院 | ||
| SH057 | 98-4/扎461 | 黑龙江省农业科学院绥化分院 | ||
| SH058 | 344/SX711 | 黑龙江省农业科学院绥化分院 | ||
| SH059 | SX711/SX709/ 81162 | 黑龙江省农业科学院绥化分院 | ||
| SH060(Reid) | SX718 | 黑龙江省农业科学院绥化分院 | ||
| SH061 | SX718/8941/ C7-2 | 黑龙江省农业科学院绥化分院 | ||
| SH062 | RA1 | 中国农业科学院作物科学研究所 | ||
| SH063 | RA3 | 中国农业科学院作物科学研究所 | ||
| SH064 | RA7 | 中国农业科学院作物科学研究所 | ||
| SH065 | RA8 | 中国农业科学院作物科学研究所 | ||
| SH066 | RA12 | 中国农业科学院作物科学研究所 | ||
| SH067 | RA10 | 中国农业科学院作物科学研究所 | ||
| SH068 | RA13 | 中国农业科学院作物科学研究所 | ||
| SH069 | S1025 | 黑龙江省农业科学院绥化分院 | ||
| SH070 | S007♀ | 黑龙江省农业科学院绥化分院 | ||
| SH071 | SY203♂ | 黑龙江省农业科学院绥化分院 | ||
| SH072 | SD3♀ | 黑龙江省农业科学院绥化分院 | ||
| SH073 | SWM76 | 黑龙江省农业科学院绥化分院 | ||
| SH074 | CA193 | 中国农业科学院作物科学研究所 | ||
| SH075(Reid) | KWS10/KWS73 | 黑龙江省农业科学院绥化分院 | ||
| SH076 | L201 | 国内骨干 | ||
| SH077 | SD3♂ | 黑龙江省农业科学院绥化分院 | ||
| SH078 | L237 | 国内骨干 | ||
| SH079 | SL202 | 黑龙江省农业科学院绥化分院 | ||
| SH080 | S65♀ | 黑龙江省农业科学院绥化分院 | ||
| SH081 | S65♂ | 黑龙江省农业科学院绥化分院 | ||
| SH082 | WC007 | 吉林省中玉农业有限公司 | ||
| SH083(Reid) | 郑58 | PA群对照 | ||
| SH084(Dom) | 444 | 唐四平头群对照 | ||
| SH085(Non-Reid) | 合344 | 兰卡斯特对照 | ||
| SH086 | 9176/C8605 | 黑龙江省农业科学院佳木斯分院 | ||
| SH087 | SX717 | 黑龙江省农业科学院绥化分院 | ||
| SH088 | SX711 | 黑龙江省农业科学院绥化分院 | ||
| SH089 | SX721 | 黑龙江省农业科学院绥化分院 | ||
| SH090 | SX718/30747 | 黑龙江省农业科学院绥化分院 | ||
| SH091 | 4207/C8605/ SX718 | 黑龙江省农业科学院绥化分院 | ||
| SH092 | SX619 | 黑龙江省农业科学院绥化分院 | ||
| SH093 | 20837 | 黑龙江省农业科学院绥化分院 | ||
| SH094 | SD302♂ | 黑龙江省农业科学院绥化分院 | ||
| SH095 | SD302♀ | 黑龙江省农业科学院绥化分院 | ||
| SH096 | SX706 | 黑龙江省农业科学院绥化分院 | ||
| SH097(Dom) | C7-2 | 唐四平头群对照 | ||
| SH098 | SD1♀ | 黑龙江省农业科学院绥化分院 | ||
| SH099 | 531 | 东北农业大学 | ||
| SH100 | WY1M1 | 吉林省中玉农业有限公司 | ||
1.2 DNA提取
大田中幼苗3叶1心时取嫩叶,采用磁珠法提取100份样品DNA。分别通过琼脂糖电泳和紫外分光光度计检测DNA纯度和浓度等质量状况。选择条带单一、明亮、完整无降解且无拖尾现象的DNA检测琼脂糖电泳,紫外分光光度计检测OD260/OD280值介于1.8~2.0,DNA浓度介于30~120 ng/μL。
1.3 基因型鉴定
采用北京市农林科学院玉米研究中心研制的Maize6H-60K芯片进行基因型分析。Maize6H-60K芯片中有61 214个SNP位点在染色体上的分布,染色体1号到10号上SNP位点数分别是9552、7334、7264、6591、6423、5068、4991、4886、4873、4232个。Affymetrix芯片使用DQC和QC call-rate进行试验质控。DQC是使用已知的、非多态性的marker计算AT通道和GC通道的区分度,DQC是对芯片通道信号值的质控。QC call-rate是选取已测试过的分型效果好的约20K探针评估call rate,QC call-rate是对样品的质控。试验要求QC>90,DQC>0.82。使用Axiom Analysis Suite 3.1.51软件分别依据PolyHighResolution、NoMinorHom、MonoHighResolution、OTVCallRateBelowThreshold、Other分类法对CEL文件进行分型[11]。
1.4 数据处理
利用TASSEL软件完成基因型数据描述性分析和玉米种质资源间遗传距离计算,采用UPGMA方法进行聚类分析,利用MEGA 11软件绘制聚类图,利用R软件计算玉米种质资源间遗传相似度。用GCTA 1.94.0进行主成分分析。
2 结果与分析
2.1 基因型数据的描述性分析
采用Maize6H-60K芯片进行扫描,获得100个样本的61 224个SNP位点的原始数据,将这些数据利用Axiom Analysis进行基因型分析,结果(表2)显示,61 224个SNP位点有6种类型,其中分型较好的有PHR类型,为39 381个,MHR为178个,NMH为127个,这些数据可进行各种遗传应用分析;其中分型不好的有OTV类型,为11 216个,CRBT类型1373个,其他类型8939个,这些SNP位点未能获得准确可区分的基因型数据。
表2 61 224个SNP位点的6种类型
Table 2
| 转换型Conversion type | 数量Number | 百分比Percentage (%) |
|---|---|---|
| PHR | 39 381 | 64.333 |
| OTV | 11 216 | 18.323 |
| 其他Other | 8939 | 14.603 |
| CRBT | 1373 | 2.243 |
| MHR | 178 | 0.291 |
| NMH | 127 | 0.207 |
PHR类型的39 381个SNP在100份玉米资源的基因型最小等位变异频率变化范围为0~0.5,平均值为0.3146;缺失数据比例变化范围0~0.1200,平均值为0.01095;杂合度比例变化范围为0~ 0.70213,平均值为0.03846。
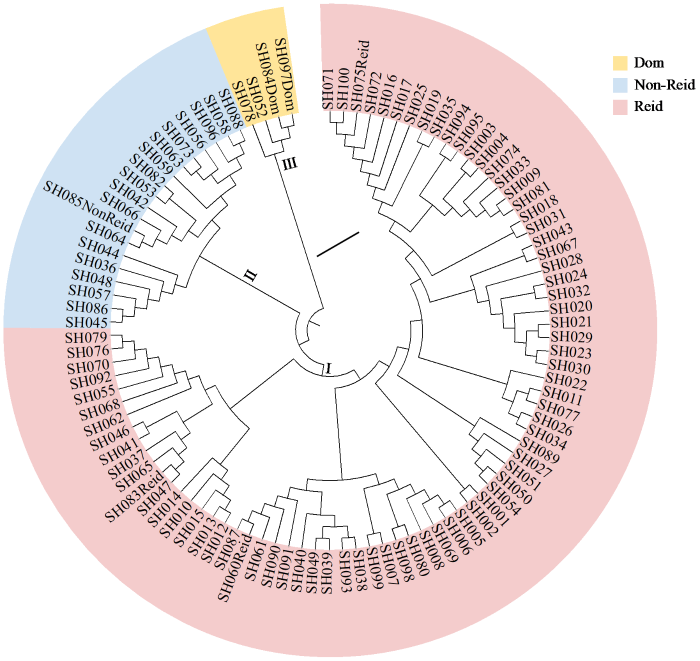
2.2 供试材料聚类分析
聚类分析结果(图1)表明,以6份测验种为参考,100份玉米资源被划分为3大类,其中类群Ⅰ为Reid群,占77%,包含KWS10/KWS73/欧早、SX618/欧早、S1029、SI15412、S1035等77份玉米资源;类群Ⅱ为Non-Reid群,占19%,包含344/9229、98-4/郑58、6171/SX711等19份玉米资源;类群Ⅲ为Dom群,共4份资源,占4%,包含W08、L237、444和C7-2。
图1
图1
基于遗传距离的100份玉米自交系聚类分析图
Fig.1
Cluster analysis diagram of the 100 maize inbred lines based on genetic distance
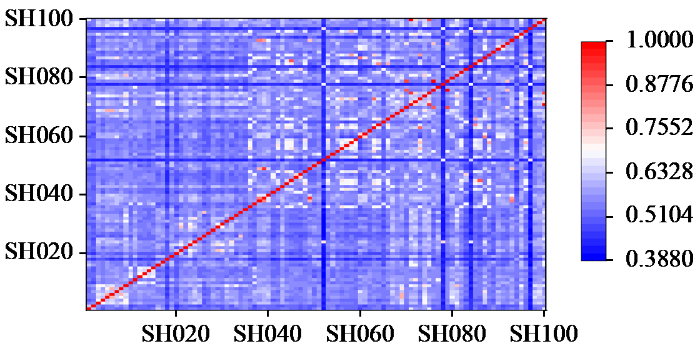
2.3 供试材料遗传相似度分析
图2
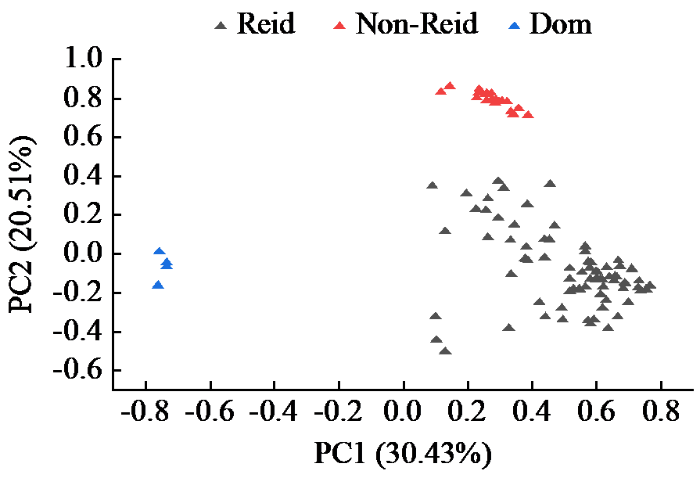
2.4 供试材料主成分分析
根据遗传相似矩阵进行主成分分析(图3),PC1和PC2的贡献率分别为30.43%和20.51%,累计贡献率50.94%。根据100份自交系的集中程度,可清晰地鉴定出3大类群,这与群体遗传距离聚类分析结果基本一致。
图3
图3
根据遗传相似矩阵对100份玉米自交系进行主成分分析
Fig.3
Principal component analysis of the 100 maize inbred lines based on genetic similarity matrix
3 讨论
研究者[16]常根据材料的表型、系谱来源和分子标记等方面对玉米种质资源进行优势类群划分,目前利用分子标记技术划分玉米杂种优势类群更方便可靠[17]。在分子标记技术中,SNP标记技术由于具有高通量检测和高密度分布等优势,常被应用于划分玉米杂种优势类群[18⇓⇓⇓⇓⇓-24]。目前在我国北方的生产应用中,育种家主要利用Reid、Non-Reid(以Lancaster为主)和Dom群(塘四平头群和旅大红骨类群)这三大优势类群的材料,本研究利用Maize6H-60K SNP芯片对100份玉米自交系进行基因型分析,计算出100份玉米自交系之间的遗传距离,根据材料间遗传距离进行聚类分析,将这100份种质资源划分为3个类群(Reid、Non-Reid和Dom)。其中划分为Reid类群的自交系最多,占77%,而Dom类群只有4%,这是因为近些年黑龙江地区的玉米育种目标主要为高产、抗病、抗倒伏、脱水快和适于机械化收获。Reid类群的特点多为植株叶片较上冲、果穗大、籽粒马齿型、抗病、抗倒伏和配合力高,因此该类群材料在育种工作中使用较多,血缘偏向于Reid类群的种质资源占比较大。Dom类群为国内玉米种质资源,包括塘四平头群和旅大红骨类群,在黑龙江地区的玉米育种中的使用相对较少,但是该类群有很大的利用潜力,比如塘四平头群在耐密方面有优势,旅大红骨群的果穗大、配合力强。
遗传相似度分析可以更进一步研究种质资源间的遗传关系。相同杂种优势类群的玉米自交系的遗传相似度可达到60%以上,遗传相似度为50%左右或以下代表血缘关系不明显[25]。当种质间遗传相似度小于0.9时,这2个种质材料可当不同的自交系应用于育种工作。当遗传相似度大于0.9或接近1.0,代表2份材料遗传背景相似度过高,育种上应尽量避免使用[13]。本研究遗传相似度结果表明,100份玉米资源遗传相似度分布范围为0.3380~0.9928。有4对玉米材料之间的遗传相似度超过了0.9,其中SH071和SH100之间的遗传相似度大于0.99。说明SH71和SH100可能为同一来源。通过遗传相似分析,对这100份玉米自交系的血缘关系的了解更加清晰,为以后的育种工作避免了一些无效组合。为了得到更加直观的遗传相似度分析结果,对这100份自交系的遗传相似度进行主成分分析,根据100份自交系的集中程度,可清晰地鉴定出3个优势类群,这与群体遗传距离的聚类分析结果基本一致。通过聚类分析、遗传相似度分析和主成分分析,对这100份种质资源遗传关系的认知变得更加清晰,为利用和改良这些自交系提供参考。
4 结论
通过SNP芯片标记进行100份玉米种质资源遗传多样性分析,根据遗传距离进行聚类分析,划分为3个类群(Reid、Non-Reid和Dom)。利用R软件计算100份玉米资源间遗传相似度,分布范围为0.3380~0.9928,发现有4对玉米材料之间的遗传相似度超过了0.9,这些材料在育种工作中使用时需要注意。对玉米材料间遗传相似度进行主成分分析,与遗传距离的聚类分析结果基本一致。
参考文献
SSR标记在玉米杂种优势类群划分中的应用
Genomic insights into historical improvement of heterotic groups during modern hybrid maize breeding
DOI:10.1038/s41477-022-01190-2
PMID:35851624
[本文引用: 1]

Single-cross maize hybrids display superior heterosis and are produced from crossing two parental inbred lines belonging to genetically different heterotic groups. Here we assembled 1,604 historically utilized maize inbred lines belonging to various female heterotic groups (FHGs) and male heterotic groups (MHGs), and conducted phenotyping and genomic sequencing analyses. We found that the FHGs and MHGs have undergone both convergent and divergent changes for different sets of agronomic traits. Using genome-wide selection scans and association analyses, we identified a large number of candidate genes that contributed to the improvement of agronomic traits of the FHGs and MHGs. Moreover, we observed increased genetic differentiation between the FHGs and MHGs across the breeding eras, and we found a positive correlation between increasing heterozygosity levels in the differentiated genes and heterosis in hybrids. Furthermore, we validated the function of two selected genes and a differentiated gene. This study provides insights into the genomic basis of modern hybrid maize breeding.© 2022. The Author(s), under exclusive licence to Springer Nature Limited.
De novo genome assembly and analyses of 12 founder inbred lines provide insights into maize heterosis
DOI:10.1038/s41588-022-01283-w
PMID:36646891
[本文引用: 2]

Hybrid maize displays superior heterosis and contributes over 30% of total worldwide cereal production. However, the molecular mechanisms of heterosis remain obscure. Here we show that structural variants (SVs) between the parental lines have a predominant role underpinning maize heterosis. De novo assembly and analyses of 12 maize founder inbred lines (FILs) reveal abundant genetic variations among these FILs and, through expression quantitative trait loci and association analyses, we identify several SVs contributing to genomic and phenotypic differentiations of various heterotic groups. Using a set of 91 diallel-cross F hybrids, we found strong positive correlations between better-parent heterosis of the F hybrids and the numbers of SVs between the parental lines, providing concrete genomic support for a prevalent role of genetic complementation underlying heterosis. Further, we document evidence that SVs in both ZAR1 and ZmACO2 contribute to yield heterosis in an overdominance fashion. Our results should promote genomics-based breeding of hybrid maize.© 2023. The Author(s), under exclusive licence to Springer Nature America, Inc.
基于SNP标记揭示中国鲜食玉米品种的遗传多样性
DOI:10.3724/SP.J.1006.2022.13031
[本文引用: 1]

中国已经成为全球鲜食玉米最主要生产和消费国家之一, 解析我国鲜食玉米品种之间的遗传多样性和亲缘关系, 对品种鉴定和品种培育具有重要的指导意义。本研究利用Illumina Maize 6K芯片对全国范围内的385个鲜食玉米品种进行全基因组扫描, 了解群体结构, 划分种质类群, 估算品种间的遗传距离, 揭示其遗传多样性。结果表明, 5067个SNP标记在385个鲜食玉米品种中基因多样性平均0.406, 变幅为0.097~0.500; 多态信息含量(polymorphism information content, PIC)平均0.319, 变幅为0.092~0.375。通过PCA分析和群体遗传结构分析一致表明, 本研究所收集的品种主要划分为3个类群, 分别为糯玉米类群(糯玉米和甜糯玉米, 共185个品种)、温带甜玉米类群(123个品种)和热带甜玉米类群(77个品种)。两两品种间的遗传距离在0.132~0.472之间, 平均值为0.37。通过F<sub>ST</sub>分析检测到不同类群间有160个区域受到强烈选择, 其中包括4个玉米籽粒淀粉合成途径的关键基因(sh2、su1、su2和wx1), 进一步利用分子标记验证了sh2和DGAT1-2两个位点在鲜食玉米群体不同选择模式。本研究为我国鲜食玉米品种选育和改良提供了重要的理论指导。
SNP分子标记在作物品种鉴定中的应用和展望
DOI:10.13430/j.cnki.jpgr.20200309002
[本文引用: 1]

单核苷酸多态性(SNP)作为最新一代遗传分子标记,已广泛应用于生物各研究领域。本文围绕SNP 分子标记的开发和检测以及在作物品种鉴定中的应用展开阐述。目前SNP 的开发主要有基于公共数据库和测序两种途径,检测方法从以凝胶电泳为基础的传统检测向高通量自动化的新检测技术发展。在品种鉴定中,对于大样本群体和多SNP 检测位点的作物,建议使用基因芯片或基因测序分型(GBS)技术;对于小样本群体和少SNP 检测1 位点的作物,则通过竞争性等位基因特异性PCR(KASP)和高分辨率熔解曲线(HRM)分型技术实现更加灵活高效低成本的检测。
The wheat 660K SNP array demonstrates great potential for marker-assisted selection in polyploid wheat
DOI:10.1111/pbi.13361
PMID:32065714
[本文引用: 1]

The rapid development and application of molecular marker assays have facilitated genomic selection and genome-wide linkage and association studies in wheat breeding. Although PCR-based markers (e.g. simple sequence repeats and functional markers) and genotyping by sequencing have contributed greatly to gene discovery and marker-assisted selection, the release of a more accurate and complete bread wheat reference genome has resulted in the design of single-nucleotide polymorphism (SNP) arrays based on different densities or application targets. Here, we evaluated seven types of wheat SNP arrays in terms of their SNP number, distribution, density, associated genes, heterozygosity and application. The results suggested that the Wheat 660K SNP array contained the highest percentage (99.05%) of genome-specific SNPs with reliable physical positions. SNP density analysis indicated that the SNPs were almost evenly distributed across the whole genome. In addition, 229 266 SNPs in the Wheat 660K SNP array were located in 66 834 annotated gene or promoter intervals. The annotated genes revealed by the Wheat 660K SNP array almost covered all genes revealed by the Wheat 35K (97.44%), 55K (99.73%), 90K (86.9%) and 820K (85.3%) SNP arrays. Therefore, the Wheat 660K SNP array could act as a substitute for other 6 arrays and shows promise for a wide range of possible applications. In summary, the Wheat 660K SNP array is reliable and cost-effective and may be the best choice for targeted genotyping and marker-assisted selection in wheat genetic improvement.© 2020 The Authors. Plant Biotechnology Journal published by Society for Experimental Biology and The Association of Applied Biologists and John Wiley & Sons Ltd.
Development of a maize 55K SNP array with improved genome coverage for molecular breeding
Potential to improve oilseed rape and canola breeding in the genomics era.
基于SNP标记技术的糯玉米种质遗传多样性分析
DOI:10.7668/hbnxb.2015.03.015
[本文引用: 1]

为揭示糯玉米种质的遗传多样性,利用新一代分子标记技术-SNPs(Single nucleotide polymorphisms)对39份不同基因型的糯玉米自交系进行了基因型分析。结果表明,1 059个SNP标记在39份自交系中的多态性信息含量(PIC)为0.05~0.38,平均含量为0.31;最小等位基因频率(MAF)为0.03~0.50,平均值为0.29;期望杂合度变化范围为0.05~0.50,平均期望杂合度为0.39。39份自交系之间的亲缘关系(Kinship)系数为0.00~0.91,京6与京糯6之间亲缘关系最近。通过Neighbor-joining(NJ)聚类分析,将39份自交系划分为5大类群,其中系谱来源不清晰的糯玉米自交系均被划分至不同类群中,明确了其亲缘关系。因此,SNP标记适用于糯玉米自交系的遗传多样性分析及亲缘关系研究,可为糯玉米种质资源利用及品种选育提供参考。
Crop breeding chips and genotyping platforms: progress, challenges, and perspectives
DOI:S1674-2052(17)30174-0
PMID:28669791
[本文引用: 1]

There is a rapidly rising trend in the development and application of molecular marker assays for gene mapping and discovery in field crops and trees. Thus far, more than 50 SNP arrays and 15 different types of genotyping-by-sequencing (GBS) platforms have been developed in over 25 crop species and perennial trees. However, much less effort has been made on developing ultra-high-throughput and cost-effective genotyping platforms for applied breeding programs. In this review, we discuss the scientific bottlenecks in existing SNP arrays and GBS technologies and the strategies to develop targeted platforms for crop molecular breeding. We propose that future practical breeding platforms should adopt automated genotyping technologies, either array or sequencing based, target functional polymorphisms underpinning economic traits, and provide desirable prediction accuracy for quantitative traits, with universal applications under wide genetic backgrounds in crops. The development of such platforms faces serious challenges at both the technological level due to cost ineffectiveness, and the knowledge level due to large genotype-phenotype gaps in crop plants. It is expected that such genotyping platforms will be achieved in the next ten years in major crops in consideration of (a) rapid development in gene discovery of important traits, (b) deepened understanding of quantitative traits through new analytical models and population designs, (c) integration of multi-layer -omics data leading to identification of genes and pathways responsible for important breeding traits, and (d) improvement in cost effectiveness of large-scale genotyping. Crop breeding chips and genotyping platforms will provide unprecedented opportunities to accelerate the development of cultivars with desired yield potential, quality, and enhanced adaptation to mitigate the effects of climate change.Copyright © 2017 The Authors. Published by Elsevier Inc. All rights reserved.