玉米是重要的粮食及饲料作物,是饲料和畜牧行业生产的主要投入品,也是深加工等行业的重要原料,其次还关联到化工和医药等领域,长期以来在生产中占据重要地位[1⇓-3],对保障我国粮食及饲料安全有着重要作用[4]。了解玉米的种群结构和群体遗传多样性等相关信息,对玉米种质的开发利用具有重要意义[5]。种质资源是品种改良工作的基础,种质资源的数量及质量是实现育种目标的关键,因此对优良种质的广泛搜集、鉴定评价工作极为重要[6]。遗传多样性分析是作物种质资源研究的一种重要手段,可以从形态学标记与分子标记2个方面进行,了解不同品种之间的遗传差异,为推进植物育种与遗传改良奠定基础[7]。形态标记等传统的评价方法很难对种质资源进行差异比较,无法精准评估种质特性[8],而不受环境影响的DNA分子标记为遗传育种和种质鉴定研究提供了一种准确有效的方法[9]。SSR标记具有易扩增、共显性及多态性高等优点,被广泛应用于遗传连锁图谱的绘制、群体遗传学、种质鉴定与亲缘分析等方面[10]。本研究利用SSR分子标记技术,以河北省的自交系及各地区收集的玉米品种为研究对象,对其进行遗传多样性分析,为玉米种质收集、评价及合理利用提供理论依据。
1 材料与方法
1.1 参试材料
参试材料具体见表1,共69份,其中材料1~27和58~69为自交系,其余皆为品种(50~57为早熟品种)。
表1 参试材料基本信息
Table 1
| 编号 Number | 名称 Name | 育成地 Breeding area | 编号 Number | 名称 Name | 育成地 Breeding area | 编号 Number | 名称 Name | 育成地 Breeding area | ||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | WL134 | 河北 | 24 | X324 | 河北 | 47 | 联创825 | 北京 | ||
| 2 | 冀42 | 河北 | 25 | Z598 | 河北 | 48 | 中科玉505 | 北京 | ||
| 3 | A87 | 河北 | 26 | L91158 | 河北 | 49 | 吉单27 | 东北三省 | ||
| 4 | X372 | 河北 | 27 | H86B | 河北 | 50 | 利合228 | 山西 | ||
| 5 | X302 | 河北 | 28 | 承玉33 | 河北 | 51 | 先玉1802 | 东北三省 | ||
| 6 | 石359 | 河北 | 29 | 锦华105 | 北京 | 52 | C7367 | 甘肃 | ||
| 7 | 外杂11 | 河北 | 30 | 巡天1102 | 河北 | 53 | 义超863 | 东北三省 | ||
| 8 | 冀H521 | 河北 | 31 | 飞天358 | 湖北 | 54 | 科沃8904 | 东北三省 | ||
| 9 | H1710 | 河北 | 32 | 农华101 | 北京 | 55 | G1938 | 甘肃 | ||
| 10 | H74 | 河北 | 33 | 京科968 | 北京 | 56 | C2235 | 甘肃 | ||
| 11 | L117 | 河北 | 34 | 先玉335 | 东北三省 | 57 | 685 | 山东 | ||
| 12 | HF138-1 | 河北 | 35 | 杂交农大108 | 北京 | 58 | 604母 | 河北 | ||
| 13 | 衡H14 | 河北 | 36 | 隆平206 | 安徽 | 59 | 冀18FF | 河北 | ||
| 14 | JN1028 | 河北 | 37 | 农大372 | 北京 | 60 | 903母 | 河北 | ||
| 15 | W25 | 河北 | 38 | 京农科728 | 北京 | 61 | 掖476 | 河北 | ||
| 16 | L5895 | 河北 | 39 | 裕丰303 | 北京 | 62 | H92 | 河北 | ||
| 17 | 石1447 | 河北 | 40 | 纪元196 | 河北 | 63 | WL134 | 河北 | ||
| 18 | B132 | 河北 | 41 | 纪元168 | 河北 | 64 | 冀42 | 河北 | ||
| 19 | 农系3435 | 河北 | 42 | DM1 | 东北三省 | 65 | H521W | 河北 | ||
| 20 | H59 | 河北 | 43 | 鹏玉1号 | 东北三省 | 66 | 冀5F | 河北 | ||
| 21 | X522 | 河北 | 44 | 纪元155 | 河北 | 67 | 冀75F父 | 河北 | ||
| 22 | 冀57 | 河北 | 45 | 纪元128 | 河北 | 68 | K36 | 河北 | ||
| 23 | JN1118 | 河北 | 46 | 纪元1号 | 河北 | 69 | S221 | 河北 |
1.2 DNA提取及PCR扩增
DNA提取:使用北京庄盟国际生物基因科技有限公司的植物基因组DNA提取试剂盒提取供试种质DNA,用超微量分光光度计检测DNA浓度和纯度。SSR标记:采用了40对SSR引物,均为玉米品种鉴定行业标准中发布的引物,具有多态性好、均匀分布在玉米的10条染色体上的特征,具体引物名称、序列及片段长度等信息见参考文献[11]。
PCR-SSR反应体系为20 μL,包括10× PCR buffer 2 μL、2.5 mmol/L dNTP 1 μL、模板DNA1 μL、正反向引物各1 μL、DMSO稳定剂1 μL、10× Loading buffer 2 μL,补充ddH2O至20 μL。进行PCR反应时覆盖一滴矿物油。PCR反应程序为95 ℃预变性3 min;95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸3 min,4 ℃保存。采用8%的非变性聚丙烯酰胺凝胶进行反应产物电泳检测,银染后拍照记录保存。
1.3 数据处理
2 结果与分析
2.1 SSR标记的遗传多样性
通过GenALEx得到供试玉米种质在引物上的遗传多样性指数(表2),利用40对引物对玉米种质DNA进行扩增,最终筛选出32对多态性好、条带清晰的引物,共检测出97个等位基因,每个SSR位点的等位基因变异范围为2~5,平均每对引物扩增3个等位基因,多态性位点百分率为68.75%。32对引物的多态信息量(PIC)变化范围为0.08~0.51,平均为0.27,引物216的PIC值最低,引物222的PIC值最高;其中16对SSR引物为中高度多态性引物(PIC>0.25);32对SSR引物的平均Ne、I和He分别为1.40、0.31、0.20。结果表明选取的SSR标记有较丰富的多态性,能较好地检测参试材料的遗传多样性。引物203和205的固定指数(F)均小于0。
表2 32个位点在供试玉米种质资源的遗传多样性指数
Table 2
| 位点Locus | Na | Ne | I | Ho | He | F | PIC |
|---|---|---|---|---|---|---|---|
| 201 | 1.56 | 1.23 | 0.23 | 0.02 | 0.14 | 0.71 | 0.20 |
| 202 | 1.56 | 1.36 | 0.29 | 0.01 | 0.19 | 0.93 | 0.28 |
| 203 | 1.89 | 1.75 | 0.54 | 0.45 | 0.38 | -0.18 | 0.49 |
| 204 | 1.78 | 1.40 | 0.38 | 0.28 | 0.24 | 0.05 | 0.24 |
| 205 | 1.56 | 1.29 | 0.27 | 0.21 | 0.17 | -0.11 | 0.15 |
| 209 | 1.78 | 1.55 | 0.39 | 0.20 | 0.25 | 0.14 | 0.29 |
| 210 | 1.89 | 1.72 | 0.47 | 0.12 | 0.30 | 0.55 | 0.36 |
| 213 | 1.89 | 1.51 | 0.43 | 0.14 | 0.28 | 0.53 | 0.33 |
| 214 | 1.44 | 1.18 | 0.17 | 0.00 | 0.11 | 1.00 | 0.16 |
| 215 | 1.56 | 1.20 | 0.22 | 0.00 | 0.13 | 1.00 | 0.20 |
| 216 | 1.33 | 1.07 | 0.10 | 0.03 | 0.06 | 0.51 | 0.08 |
| 217 | 1.44 | 1.17 | 0.19 | 0.09 | 0.12 | 0.14 | 0.15 |
| 218 | 1.44 | 1.32 | 0.24 | 0.00 | 0.16 | 1.00 | 0.25 |
| 219 | 2.00 | 1.66 | 0.43 | 0.01 | 0.25 | 0.96 | 0.38 |
| 220 | 1.44 | 1.34 | 0.27 | 0.00 | 0.19 | 1.00 | 0.22 |
| 221 | 1.56 | 1.29 | 0.29 | 0.00 | 0.19 | 1.00 | 0.23 |
| 222 | 2.22 | 2.05 | 0.58 | 0.01 | 0.34 | 0.96 | 0.51 |
| 223 | 1.22 | 1.19 | 0.14 | 0.00 | 0.10 | 1.00 | 0.12 |
| 224 | 1.78 | 1.53 | 0.40 | 0.00 | 0.25 | 1.00 | 0.38 |
| 225 | 1.67 | 1.49 | 0.30 | 0.00 | 0.18 | 1.00 | 0.28 |
| 226 | 1.56 | 1.26 | 0.24 | 0.06 | 0.16 | 0.54 | 0.23 |
| 227 | 1.67 | 1.41 | 0.35 | 0.00 | 0.22 | 1.00 | 0.33 |
| 228 | 1.44 | 1.25 | 0.22 | 0.00 | 0.14 | 1.00 | 0.21 |
| 229 | 1.44 | 1.28 | 0.25 | 0.00 | 0.17 | 1.00 | 0.20 |
| 230 | 1.56 | 1.37 | 0.30 | 0.03 | 0.20 | 0.84 | 0.30 |
| 232 | 1.56 | 1.33 | 0.27 | 0.00 | 0.17 | 1.00 | 0.26 |
| 234 | 2.00 | 1.57 | 0.47 | 0.15 | 0.29 | 0.53 | 0.36 |
| 235 | 1.44 | 1.19 | 0.17 | 0.01 | 0.10 | 0.88 | 0.15 |
| 237 | 1.44 | 1.28 | 0.25 | 0.00 | 0.17 | 1.00 | 0.20 |
| 238 | 2.00 | 1.59 | 0.44 | 0.00 | 0.27 | 1.00 | 0.40 |
| 240 | 1.89 | 1.54 | 0.43 | 0.14 | 0.27 | 0.49 | 0.41 |
| 603 | 1.44 | 1.31 | 0.26 | 0.00 | 0.18 | 1.00 | 0.21 |
| 平均Mean | 1.64 | 1.40 | 0.31 | 0.06 | 0.20 | 0.73 | 0.27 |
2.2 群体遗传多样性分析
分析群体间遗传分化系数和基因流,发现玉米种群有3个位点的近交系数(Fis)小于0,其余位点的近交系数均大于种群间无遗传分化的标准(Fst=0~0.05)[15],具体见表3。群体间遗传分化系数Fst为0.14~0.77,表明群体间存在不同程度的遗传分化;其中自交系和河北省品种之间的分化系数最小,山东省和安徽省品种的分化系数最大。群体遗传分化系数在不同位点上的平均值为0.286,表明总遗传变异有28.6%来自群体之间,71.4%来自群体之内,说明样本群体遗传变异主要发生在群体内。群体间Nm在0.034~2.883之间,说明群体间存在不同程度的基因交流,其中又以自交系与河北省间的Nm值最大,基因交流最为密切,具体见表4。
表3 玉米群体内各位点遗传分析
Table 3
| 位点Locus | 近交系数Fis | 分化系数Fst | 基因流Nm |
|---|---|---|---|
| 201 | 0.84 | 0.76 | 0.08 |
| 202 | 0.93 | 0.51 | 0.24 |
| 203 | -0.18 | 0.17 | 1.27 |
| 204 | -0.16 | 0.21 | 0.96 |
| 205 | -0.19 | 0.24 | 0.79 |
| 209 | 0.22 | 0.35 | 0.46 |
| 210 | 0.59 | 0.55 | 0.21 |
| 213 | 0.49 | 0.39 | 0.39 |
| 214 | 1.00 | 0.25 | 0.74 |
| 215 | 1.00 | 0.60 | 0.17 |
| 216 | 0.50 | 0.77 | 0.08 |
| 217 | 0.27 | 0.14 | 1.61 |
| 218 | 1.00 | 0.68 | 0.12 |
| 219 | 0.97 | 0.63 | 0.15 |
| 220 | 1.00 | 0.53 | 0.22 |
| 221 | 1.00 | 0.59 | 0.17 |
| 222 | 0.96 | 0.44 | 0.31 |
| 223 | 1.00 | 0.68 | 0.12 |
| 224 | 1.00 | 0.51 | 0.24 |
| 225 | 1.00 | 0.56 | 0.20 |
| 226 | 0.64 | 0.31 | 0.56 |
| 227 | 1.00 | 0.58 | 0.18 |
| 228 | 1.00 | 0.72 | 0.10 |
| 229 | 1.00 | 0.53 | 0.22 |
| 230 | 0.88 | 0.53 | 0.23 |
| 232 | 1.00 | 0.52 | 0.23 |
| 234 | 0.48 | 0.38 | 0.41 |
| 235 | 0.88 | 0.21 | 0.92 |
| 237 | 1.00 | 0.55 | 0.20 |
| 238 | 1.00 | 0.56 | 0.20 |
| 240 | 0.50 | 0.50 | 0.25 |
| 603 | 1.00 | 0.52 | 0.23 |
表4 玉米群体间遗传分化系数和基因流
Table 4
| 地区 Area | 自交系 Inbred line | 河北 Hebei | 北京 Beijing | 东北三省 Three provinces in the northeast of China | 甘肃 Gansu | 湖北 Hubei | 安徽 Anhui | 山西 Shanxi | 山东 Shandong |
|---|---|---|---|---|---|---|---|---|---|
| 自交系Inbred line | *** | 2.883 | 2.492 | 2.007 | 0.957 | 0.673 | 0.686 | 0.542 | 0.517 |
| 河北Hebei | 0.080 | *** | 2.712 | 2.289 | 1.064 | 0.437 | 0.840 | 0.610 | 0.520 |
| 北京Beijing | 0.091 | 0.084 | *** | 2.607 | 0.942 | 0.490 | 0.880 | 0.531 | 0.443 |
| 东北三省 Three provinces in the northeast of China | 0.111 | 0.098 | 0.087 | *** | 1.833 | 0.348 | 0.955 | 0.899 | 0.537 |
| 甘肃Gansu | 0.207 | 0.190 | 0.210 | 0.120 | *** | 0.160 | 0.354 | 0.383 | 0.433 |
| 湖北Hubei | 0.271 | 0.364 | 0.338 | 0.418 | 0.610 | *** | 0.053 | 0.053 | 0.063 |
| 安徽Anhui | 0.267 | 0.229 | 0.221 | 0.207 | 0.414 | 0.825 | *** | 0.043 | 0.034 |
| 山西Shanxi | 0.316 | 0.291 | 0.320 | 0.218 | 0.395 | 0.824 | 0.852 | *** | 0.050 |
| 山东Shandong | 0.326 | 0.325 | 0.361 | 0.318 | 0.366 | 0.800 | 0.879 | 0.833 | *** |
“***”左下为群体间Fst,右上为群体间Nm。
The lower left of“***”is the intergroup Fst, and the upper right is the intergroup Nm.
对不同玉米群体进行遗传多样性分析,结果(表5)显示,不同玉米群体观测到的平均Ne在1.00~2.18,I在0.00~0.83,He在0.00~0.50,PIC值在0.00~0.48之间,以上指标说明不同来源玉米群体属于中度多态性,遗传多样性水平适中;参试材料在总体水平上平均检测到1.64个Na,且有效等位基因数为1.40,表明87%的等位基因真实可靠;期望杂合度为0.20,Ho仅为0.06,表明参试材料遗传稳定、纯合度高;多态性条带百分率为98.9%,I为0.31,表明能真实反映32个位点的多态性;自交系及河北、北京和东北三省的材料Na、Ne均比总体水平高,自交系的Na、Ne、I、He和PIC皆最大,表明其遗传多样性较高、遗传基因更丰富,遗传基础比较广泛;而安徽、山西、山东的材料遗传多样性较低,遗传基础较窄。因本试验中上述省份材料比较少,仅有1个,这可能是造成遗传多样性低的原因。
表5 不同来源玉米群体的遗传多样性信息
Table 5
| 地区Area | 样本数量Sample number | Na | Ne | I | Ho | He | PIC |
|---|---|---|---|---|---|---|---|
| 自交系Inbred line | 39 | 2.94 | 2.18 | 0.83 | 0.01 | 0.50 | 0.48 |
| 河北Hebei | 7 | 2.03 | 1.64 | 0.53 | 0.06 | 0.35 | 0.33 |
| 北京Beijing | 9 | 2.09 | 1.64 | 0.54 | 0.08 | 0.34 | 0.33 |
| 东北三省Three provinces in the northeast of China | 6 | 2.00 | 1.55 | 0.48 | 0.06 | 0.30 | 0.29 |
| 甘肃Gansu | 4 | 1.41 | 1.27 | 0.23 | 0.06 | 0.16 | 0.15 |
| 湖北Hubei | 1 | 1.16 | 1.16 | 0.11 | 0.16 | 0.08 | 0.07 |
| 安徽Anhui | 1 | 1.00 | 1.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| 山西Shanxi | 1 | 1.06 | 1.06 | 0.04 | 0.06 | 0.03 | 0.03 |
| 山东Shandong | 1 | 1.06 | 1.06 | 0.04 | 0.06 | 0.03 | 0.03 |
| 平均Mean | 1.64 | 1.40 | 0.31 | 0.06 | 0.20 | 0.19 |
2.3 群体间相似度、聚类分析和主成分分析
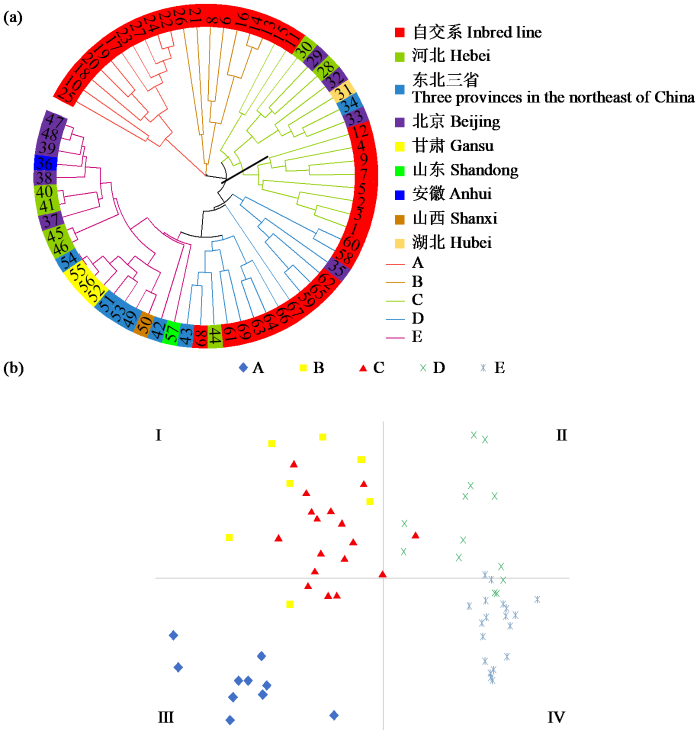
由遗传距离和遗传相似度矩阵(表6)可以看出,各群体的遗传距离介于0.103~0.844之间,自交系材料与河北的玉米品种遗传距离最近,其次是北京;与山东的玉米品种遗传距离最远。基于供试玉米材料分子标记数据进行聚类分析,从图1a可以看出,参试材料被分为5类,自交系材料分布相对集中但也被分成不同的亚群,所有自交系材料都集中分布在A、B、C、D 4个组中,且与自交系夹杂为一组的材料基本都为北京和河北的品种。东北三省的参试品种则与部分北京和河北以及甘肃的材料聚在一组,值得注意的是,E组中除了部分来自北京的材料外,其余材料的育种单位所在地农作物基本为一年一熟,可能由于当地气候环境等因素,育种单位进行种质选育时有着相似的目标选择。品种与自交系之间存在较为明显的分组,说明自交系与大部分品种之间存在较大的位点差异。对聚类组群进行主成分分析,可以进一步判断组群间的关系,将坐标图划为4个区域,主成分分析图显示虽然存在极少数的离散材料,但整体上各组群分布相对集中(图1b),基本上各自占据相应的位置。B、C组主要分布在I区,可以说明自交系与来自北京、河北的品种关系更为接近,D组分布在II区,A组集中在III区,E组主要分布在IV区。各组群坐标分布结果与聚类分组结果一致,且二者分析结果可以相互
佐证。
表6 不同地区、类型材料的遗传距离和遗传相似度矩阵
Table 6
| 地区 Area | 自交系 Inbred line | 河北 Hebei | 北京 Beijing | 东北三省 Three provinces in the northeast of China | 甘肃 Gansu | 湖北 Hubei | 安徽 Anhui | 山西 Shanxi | 山东 Shandong |
|---|---|---|---|---|---|---|---|---|---|
| 自交系Inbred line | *** | 0.869 | 0.856 | 0.829 | 0.732 | 0.703 | 0.735 | 0.628 | 0.606 |
| 河北Hebei | 0.140 | *** | 0.898 | 0.876 | 0.800 | 0.617 | 0.833 | 0.714 | 0.667 |
| 北京Beijing | 0.155 | 0.107 | *** | 0.899 | 0.767 | 0.662 | 0.853 | 0.691 | 0.619 |
| 东北三省 Three provinces in the northeast of China | 0.188 | 0.133 | 0.106 | *** | 0.902 | 0.563 | 0.854 | 0.833 | 0.713 |
| 甘肃Gansu | 0.312 | 0.224 | 0.265 | 0.103 | *** | 0.448 | 0.809 | 0.818 | 0.861 |
| 湖北Hubei | 0.353 | 0.483 | 0.412 | 0.575 | 0.803 | *** | 0.504 | 0.331 | 0.430 |
| 安徽Anhui | 0.307 | 0.182 | 0.159 | 0.158 | 0.212 | 0.684 | *** | 0.762 | 0.699 |
| 山西Shanxi | 0.465 | 0.336 | 0.370 | 0.183 | 0.201 | 1.107 | 0.272 | *** | 0.677 |
| 山东Shandong | 0.501 | 0.406 | 0.480 | 0.338 | 0.150 | 0.844 | 0.359 | 0.389 | *** |
“***”左下为遗传距离矩阵,右上为遗传相似度矩阵。
The lower left corner of“***”is the genetic distance matrix, and the upper right corner is the genetic similarity matrix.
图1
图1
参试玉米材料的聚类图(a)和聚类组群主成分分析图(b)
Fig.1
Cluster diagram (a) and cluster group principal component analysis diagram (b) of the tested maize materials
3 讨论
3.1 玉米种质资源遗传多样性
遗传多样性指生物群体携带的遗传信息的总和,是评价物种资源状况的重要依据,也是物种适应多变的环境条件、维持长期生存和进化的前提[16]。Ne、Ho、He等都是反映群体遗传多样性的指标,其数值越大,说明遗传多样性越高,适应能力和进化潜力越高,本试验中,以自交系的遗传多样性最为丰富。F统计量系数是表示种群基因分化程度和近交情况的重要指标[17],主要包括群体内近交系数Fis、总群体近交系数Fit、群体间分化系数Fst[18]。Fis越趋近于0,表示基因分布越平衡,群体中的遗传多样性越高,在本试验中,Fis和Fit的平均值分别为0.738和0.824,表明参试群体内存在较高程度的近交现象。玉米群体的观测杂合度(0.06)小于期望杂合度(0.20),近交系数高达0.738,表明整个群体中存在高度的杂合子缺失现象。这可能与玉米的繁殖特性有关,玉米是风媒花,花粉主要靠风力传播,这在一定程度上加剧了近亲交配的几率,且本试验中自交系多为二环系,在玉米育种上将从杂交后代中选到的自交系称为二环系,一般利用单交种或三交种选育二环系时,一个很重要的问题是,由于优系较少,在组配杂交时选育的二环系很容易造成亲源关系接近,导致遗传基础狭窄。
3.2 遗传距离、相似度及聚类结果分析
聚类分析可以比较直观地表示种质间的遗传背景和相似性,SSR聚类分析基本将同一来源的材料划分为一组,同时材料50~57皆为早熟品种,在聚类图中紧密接近,说明SSR标记也可以在一定程度上区分出种质类型。聚类图中将自交系与河北、北京的多数品种聚为一类,说明种质间亲缘关系的远近程度与育种单位和地理距离有一定的关系。在今后的育种实践中,对玉米种质的挖掘应结合其表型性状、生物学性状、分子标记等手段综合解析玉米资源的遗传背景。
4 结论
本研究揭示了69份玉米种质资源的遗传多样性,初步分析了参试玉米种质资源的亲缘关系和遗传差异,并将所用材料划分为5个类群,对69份玉米种质进行地理来源、SSR标记等不同角度的遗传多样性分析,结果发现地理来源与分子标记结果基本吻合,参试自交系的遗传多样性较高。
参考文献
308个糯玉米审定品种SSR标记遗传多样性分析
DOI:10.13430/j.cnki.jpgr.20210629001
[本文引用: 1]

随着中国糯玉米产业的蓬勃发展,每年通过审定的糯玉米品种逐渐增多,为了解其育成品种的遗传多样性和亲缘关系,本研究以308个糯玉米审定品种为材料,利用SSR标记从审定年份和适宜种植区2个角度分析近年来糯玉米审定品种遗传多样性特点和发展趋势。结果表明,308份糯玉米审定品种在40个SSR标记上共检测出529个等位基因、1480种基因型,PIC变幅为0.226~0.890,平均为0.684。从审定时间看,各年份间审定品种的遗传多样性指数波动不大,其中2015年审定品种的遗传多样性最高,为0.720。从适宜种植区看,西南区审定品种的PIC值最高,遗传距离分析表明西南区和另外3个适宜种植区有着最远的遗传距离,平均为0.0217;聚类分析结果显示,西南区部分品种聚集为一组,表现出一定的地域特色。聚类分组中以京科糯2000为代表的第Ⅲ组品种数目最多,且在主坐标图中独立分布,表明优良品种的出现及其杂优模式的推广能够丰富和拓宽品种的遗传多样性,为糯玉米新品种改良提供导向作用的同时推动糯玉米产业的发展。
基于SSR分子标记的41份茶树种质资源遗传多样性及亲缘关系分析
DOI:10.11924/j.issn.1000-6850.casb2022-0862
[本文引用: 1]

为了对豫南茶树种质资源进行初步鉴定和评价,以豫南茶树种质资源圃内41份无性系品种为材料,利用SSR分子标记技术进行遗传多样性及亲缘关系分析。结果表明:从33对引物中筛选出26对扩增较好的引物,26对引物共扩增出102条谱带,平均每对引物扩增条带4条,最少为2条,最多为7条,检测的平均观测等位基因数2个,平均有效等位基因数1.8664个,平均Nei’s遗传多样性0.4626,平均Shannon信息指数0.6550,引物多态信息量最大值0.80,最小值0.23,平均值0.62。聚类分析结果显示,在相似系数为0.5处,将41份材料分为4大类群,地理来源和遗传背景相近的大都聚在一起,供试品种的遗传距离为0.08~1.87,说明资源圃内品种间遗传基础较宽。
西藏石榴野生群体的SSR遗传多样性分析
DOI:10.12302/j.issn.1000-2006.202105011
[本文引用: 1]

【目的】调查西藏地区野生石榴种质资源,分析西藏野生石榴群体的遗传多样性和遗传结构,为野生石榴资源的保护和利用提供理论依据。【方法】使用13对SSR引物对3个自然群体共42份西藏地区野生石榴种质资源材料DNA进行PCR扩增,毛细管电泳检测扩增片段长度,使用GenAlEx和Arlequin等软件对SSR数据进行分析。【结果】13对引物共检测到44个等位基因,平均3.385个,引物的平均有效等位基因数(N<sub>e</sub>)、香农信息指数(I)、期望杂合度(H<sub>e</sub>)和多态信息量(PIC)分别为1.971、0.771、0.481和0.393。3个野生群体的平均有效等位基因数(N<sub>e</sub>)、平均香农信息指数(I)和平均期望杂合度(H<sub>e</sub>)分别为1.867、0.646和0.421,林芝b(LZb)群体的遗传多样性水平高于其他2个群体。AMOVA分析表明,群体内遗传变异高达88.43%,3个群体间的遗传分化系数(F<sub>st</sub>)为0.116。种质聚类分析将供试种质划分为3个亚群,结果与种质地理来源具有一定关联性。遗传结构分析显示西藏野生石榴有4个可能的基因库来源。【结论】13对SSR引物可用于西藏野生石榴种质的遗传多样性等研究。西藏野生石榴种质的遗传变异主要存在于群体内;林芝b(LZb)群体遗传多样性最高,遗传结构复杂,且含有最多的野生种质采样点,可予以优先保护。
黄颡鱼(Pelteobagrus eupogon)不同生态地理分布群体遗传多样性的微卫星分析
7个地方山羊品种遗传多样性及遗传结构分析
DOI:10.13560/j.cnki.biotech.bull.1985.2019-1069
[本文引用: 1]

检测了7个地方山羊群体在15个微卫星位点的遗传多样性和遗传结构,旨为地方山羊群体的保护利用奠定基础。采集燕山绒山羊、辽宁绒山羊、承德无角山羊、济宁青山羊、太行山羊、武安山羊血液样品及内蒙古绒山羊的耳组织,利用微卫星方法分析遗传多样性及遗传结构。结果表明,7个山羊群体的平均有效等位基因数为4.235 8、平均期望杂合度为0.718 8、平均多态信息含量为0.706 8,均具有高度的遗传多样性;总群体平均近交系数为0.088 3,平均遗传分化系数为0.544 2,平均基因流为0.209 4。武安山羊和承德无角山羊的遗传距离最近,太行山羊和燕山绒山羊的遗传距离最远。系统进化树聚类分析表明,燕山绒山羊与辽宁绒山羊聚为一类,济宁青山羊、承德无角山羊、武安山羊、内蒙古绒山羊和太行山羊聚为一类。综上,7个地方山羊群体遗传多样性丰富,群体间遗传分化程度大,基因交流少,受近交程度影响小,具有较高的利用价值和潜力。
玉米杂种优势类群划分高多态SSR引物筛选
DOI:10.6048/j.issn.1001-4330.2022.06.008
[本文引用: 1]

【目的】 研究玉米杂种优势类群划分高多态SSR引物筛选。【方法】 遴选均匀分布在玉米10个连锁群上160对SSR引物,在104份玉米自交系DNA中扩增,根据引物的染色体分布和PIC值,分别取40、30、20和10对高多态引物建立4套新引物体系,检测104份自交系的分类效果。【结果】 (1)160对引物中的63对引物带型稳定,多态性高,最高PIC值0.762 3,高于过去常用核心SSR引物,63对引物中过去常用核心SSR引物仅保留40%左右;(2)4套引物体系中,40和30对引物体系的分类结果与已知自交系类群高度吻合,20对体系与40对体系比较具90.5%一致性,10对体系与40对体系比较的一致性81.0%;(3)40对体系分类104份材料为5大类群,分别是瑞德、改良瑞德、兰卡斯特、黄改和和旅大红骨,与目前国内利用的优势群划分结果一致,也能鉴别出每个群中亲缘关系最远的亚群是相近群间二环系来源的混合基因型;(4) 各群的代表自交系是单一遗传结构成分,亚群是多遗传结构成分。【结论】 部分SSR引物的多态性在新自交系中会出现下降,建立的新的40对引物体系有精确的分类功能,可应用于类群划分,20对体系的分类精确性稍低,但检测工作量减半,在批量材料分类中有利用价值。
基于SNP标记的小麦品种遗传相似度及其检测准确度分析
DOI:10.3724/SP.J.1006.2024.31044
[本文引用: 1]

遗传相似度检测的准确度估计是对SNP标记法在农作物品种检测体系中应用的必要补充和完善。本研究基于2021年小麦品种SNP标记法跨实验室协同验证实验数据, 分析了该方法的检测准确度及在品种间的遗传相似度。分析结果表明: (1) 10个实验室对55组小麦品种组合的标记位点相似度检测的总体准确度约为98%。(2) GGE双标图的品种遗传关系功能图显示, 7组小麦品种的组内遗传相似度在95%以上, 其余组合的遗传相似度较低。(3) 依据GGE双标图的“正确度-精确度”功能图和“准确度排序”功能图, 发现洛旱7号/洛旱11等品种组合的相似度检测准确度较高, 晋麦47/临抗11的检测准确度一般, 而济麦22/婴泊700的检测准确度较差。(4) 10个实验室的检测准确度存在显著差异, 其中2个实验室检测的正确度、精确度和准确度表现显著差于其余实验室。(5) 各实验室检测正确度的容许误差分布于1.3%~1.9%之间, 平均为1.5%; 准确度的容许误差分布于1.5%~2.0%之间, 平均为1.7%。其中, Lab2和Lab3的检测正确度和准确度的容许误差显著差于其余实验室。本研究构建了SNP标记法对品种相似性检测的准确度统计模型, 分析了品种组合和实验室的检测准确度及其容许误差, 采用GGE双标图方法对检测正确度、精确度和准确度进行可视化分析, 验证了各实验室对品种位点相似性检测的准确度和可靠性, 为SNP标记法在农作物品种遗传相似性检测中的准确度评价提供了理论支持和应用范例。