开放科学(资源服务)标识码(OSID): 
普通豇豆(Vigna unguiculata)属于豆科(Leguminosae)蝶形花亚科(Papilionoideae)菜豆族(Phaseoleae)豇豆属(Vigna)[1],是世界上主要的食用豆类作物之一。普通豇豆广泛种植于非洲、拉丁美洲等贫困及生态贫瘠地区[2,3],因其富含人体所需的植物性蛋白及膳食纤维,在人们的日常饮食和营养保健中发挥着重要作用,也是亚洲、非洲和拉丁美洲等地区人们植物蛋白质的重要来源。普通豇豆具有高效的固氮能力,可提高土壤肥力,常用于禾谷类作物的轮作或间作套种等[3,4],在我国栽培历史悠久,全国各地均有种植。我国共收集豇豆种质资源4 700多份[5],但由于育种研究相对落后,主栽品种多为地方品种,资源利用率较低。
1 材料与方法
1.1 试验材料
88份普通豇豆应用核心种质中78份来自国内各省(市、区),9份来自国际干旱农业研究所,1份来源未知,这些种质的种子均由中国农业科学院作物科学研究所提供(表1)。
表1 88份普通豇豆核心种质资源信息
Table 1
| 序号 Number | 统一编号 Uniform number | 品种 Variety | 原产地 Origin | 序号 Number | 统一编号 Uniform number | 品种 Variety | 原产地 Origin | |
|---|---|---|---|---|---|---|---|---|
| BJ-1 | I00003 | 白黑眼 | 中国北京 | JL-3 | - | JD6 | 中国吉林 | |
| BJ-2 | I00030 | 红豇豆 | 中国北京 | JL-4 | - | 黑豇豆9801 | 中国吉林 | |
| BJ-3 | I01338 | 中豇1号 | 中国北京 | HL-1 | I00344 | 花豇豆 | 中国黑龙江 | |
| BJ-4 | I01343 | 豇豆 | 中国北京 | HL-2 | I02828 | 豇豆 | 中国黑龙江 | |
| BJ-5 | I01921 | 豇豆 | 中国北京 | TJ-1 | I00502 | 豇豆 | 中国天津 | |
| BJ-6 | I01922 | 豇豆 | 中国北京 | TJ-2 | I00503 | 豇豆 | 中国天津 | |
| BJ-7 | I01925 | 豇豆 | 中国北京 | SH | I00511 | 豇豆 | 中国上海 | |
| BJ-8 | I01927 | 豇豆 | 中国北京 | AH-1 | I00764 | 白豇豆 | 中国安徽 | |
| BJ-9 | I02620 | 豇豆 | 中国北京 | AH-2 | I00769 | 白豇豆 | 中国安徽 | |
| BJ-10 | I02631 | 夏宝 | 中国北京 | AH-3 | I00774 | 白豇豆 | 中国安徽 | |
| BJ-11 | I02632 | 春燕 | 中国北京 | HB-1 | I00970 | 黑饭豆 | 中国湖北 | |
| BJ-12 | I03099 | 豇豆 | 中国北京 | HB-2 | I02740 | 坡豇豆 | 中国湖北 | |
| BJ-13 | I03112 | 豇豆 | 中国北京 | GX-1 | I01117 | 红饭豆 | 中国广西 | |
| BJ-14 | I03113 | 豇豆 | 中国北京 | GX-2 | I01187 | 狗面豆 | 中国广西 | |
| BJ-15 | I03114 | 豇豆 | 中国北京 | SN-1 | I01230 | 打豇豆 | 中国陕西 | |
| BJ-16 | I03118 | 豇豆 | 中国北京 | SN-2 | - | JD4 | 中国陕西 | |
| BJ-17 | I03120 | 豇豆 | 中国北京 | SN-3 | - | JD5 | 中国陕西 | |
| BJ-18 | I03126 | 豇豆 | 中国北京 | IITA-1 | I01273 | IT82D-703 | IITA | |
| BJ-19 | I03132 | 豇豆 | 中国北京 | IITA-2 | I01274 | IT82D-716 | IITA | |
| BJ-20 | I03133 | 豇豆 | 中国北京 | IITA-3 | I01278 | IT82D-952 | IITA | |
| BJ-21 | I03137 | 豇豆 | 中国北京 | IITA-4 | I01281 | TVX3236 | IITA | |
| BJ-22 | I03138 | 豇豆 | 中国北京 | IITA-5 | I01293 | 中豇2号(IT82D-789) | IITA | |
| BJ-23 | I03139 | 豇豆 | 中国北京 | IITA-6 | I01298 | IT84E-124 | IITA | |
| BJ-24 | I03140 | 豇豆 | 中国北京 | IITA-7 | I01299 | IT82D-453-2 | IITA | |
| BJ-25 | I03141 | 豇豆 | 中国北京 | IITA-8 | I01302 | IT83S-852 | IITA | |
| BJ-26 | I03171 | 豇豆 | 中国北京 | IITA-9 | I01321 | IT82D-889 | IITA | |
| BJ-27 | I03181 | 豇豆 | 中国北京 | HA-1 | I01398 | 白豇豆 | 中国河南 | |
| BJ-28 | I03182 | 豇豆 | 中国北京 | HA-2 | I01407 | 白豇豆 | 中国河南 | |
| BJ-29 | I03282 | 豇豆 | 中国北京 | HA-3 | I02818 | 豫豇1号 | 中国河南 | |
| HE-1 | I00058 | 豇豆 | 中国河北 | NX-1 | I01831 | 豇豆 | 中国宁夏 | |
| HE-2 | I00081 | 串蔓花豇豆 | 中国河北 | NX-2 | I01836 | 豇豆 | 中国宁夏 | |
| HE-3 | I00563 | 花豇豆 | 中国河北 | SD | I02372 | 胡豇豆 | 中国山东 | |
| HE-4 | - | JD1 | 中国河北 | HN-1 | I02496 | 豇豆 | 中国湖南 | |
| SX-1 | I00093 | 爬豇豆 | 中国山西 | HN-2 | I02497 | 豇豆 | 中国湖南 | |
| SX-2 | - | JD3 | 中国山西 | HN-3 | I02504 | 枫树饭豆 | 中国湖南 | |
| IM-1 | I00199 | 红豇豆 | 中国内蒙古 | HN-4 | I02510 | 豇豆 | 中国湖南 | |
| IM-2 | I00200 | 豇豆 | 中国内蒙古 | HN-5 | I03101 | 早生王 | 中国湖南 | |
| IM-3 | I02130 | 长豇豆 | 中国内蒙古 | HN-6 | I03102 | 耐热王 | 中国湖南 | |
| IM-4 | - | JD9 | 中国内蒙古 | GD-1 | I03144 | 长豆角 | 中国广东 | |
| LN-1 | I00258 | 豇豆 | 中国辽宁 | GD-2 | I03145 | 早豆角 | 中国广东 | |
| LN-2 | I00272 | 花豇豆 | 中国辽宁 | JS-2 | - | 苏豇2号 | 中国江苏 | |
| LN-3 | I00277 | 花豇豆 | 中国辽宁 | JS-2 | - | 早豇1号 | 中国江苏 | |
| JL-1 | I00321 | 花豇豆 | 中国吉林 | JS-3 | - | 海门短肉豇 | 中国江苏 | |
| JL-2 | I00336 | 大花豇豆 | 中国吉林 | Unknow | I01314 | - | 不明 |
Note: IITA represent The International Institute of Tropical Agriculture
注:IITA指国际干旱农业研究所
1.2 基因组DNA的提取
每份材料选取2~3粒健壮种子播于营养钵内,采集三出复叶的幼嫩叶片,用改良的CTAB法[15]提取基因组DNA,用紫外分光光度计测定其浓度,用ddH2O稀释至20ng/μL,于4℃冰箱保存备用。
1.3 PCR扩增反应
1.4 凝胶电泳检测
PCR 反应结束后,用8%的聚丙烯酰胺非变性凝胶电泳分离扩增产物,200V恒压下电泳。凝胶用蒸馏水洗涤2次后,用0.2% AgNO3溶液染色10min,1.5% NaOH加0.5%甲醛溶液显影至条带清晰,用蒸馏水洗涤2次。
1.5 数据统计与分析
根据PCR扩增片段即等位变异迁移率的不同,对电泳图谱上清晰的DNA条带进行统计,样品间同一迁移位置上的电泳条带记为“1”,无则记为“0”。SSR标记多态性指标以多态性息含量(polymorphism information content,PIC)表示,PICk=1-∑(Pi)2,式中Pi表示第k个位点上第i个等位基因的频率。利用NTSYSpc 2.1计算材料间遗传相似系数,用非加权平均法(unweight pair group method with arithmetic mean,UPGMA)进行遗传相似性聚类分析,并绘制材料间亲缘关系聚类树状图。利用Microsoft Excel进行其他统计分析。
2 结果与分析
2.1 SSR标记的多态性分析
由表2可知,14对SSR引物在88份普通豇豆材料中共检测到39个等位变异,每对引物的等位变异数为2~5个,平均每对2.79个,其中引物AG1检测到的等位变异最多,其次是引物VM31和12M1。PIC值变化范围为0.25~0.72,平均为0.58。除引物VM30、VM36和VM34外,其他引物的PIC值均大于等于0.50。简单相关性分析显示,等位变异数与PIC值无显著相关。
表2 基于14个SSR分子标记的88份普通豇豆应用核心种质多态性信息
Table 2
| 引物编号 Primer number | 等位基因数 Number of alleles | 有效等位基因数 Number of effective alleles | 多态性信息含量 Polymorphism information content |
|---|---|---|---|
| VM30 | 3 | 1.34 | 0.25 |
| VM36 | 3 | 1.95 | 0.49 |
| VM31 | 4 | 2.95 | 0.66 |
| VM35 | 2 | 3.40 | 0.71 |
| VM34 | 2 | 1.73 | 0.42 |
| VM39 | 3 | 2.91 | 0.66 |
| VM28 | 2 | 2.97 | 0.66 |
| PVBR185 | 3 | 2.98 | 0.66 |
| BMd-2 | 2 | 2.25 | 0.56 |
| BMd-17 | 2 | 2.29 | 0.56 |
| AG1 | 5 | 2.08 | 0.52 |
| PVBR4 | 2 | 3.62 | 0.72 |
| PVBR12 | 2 | 3.25 | 0.69 |
| 12M1 | 4 | 2.01 | 0.50 |
| 平均 Average | 2.79 | 2.55 | 0.58 |
2.2 聚类分析
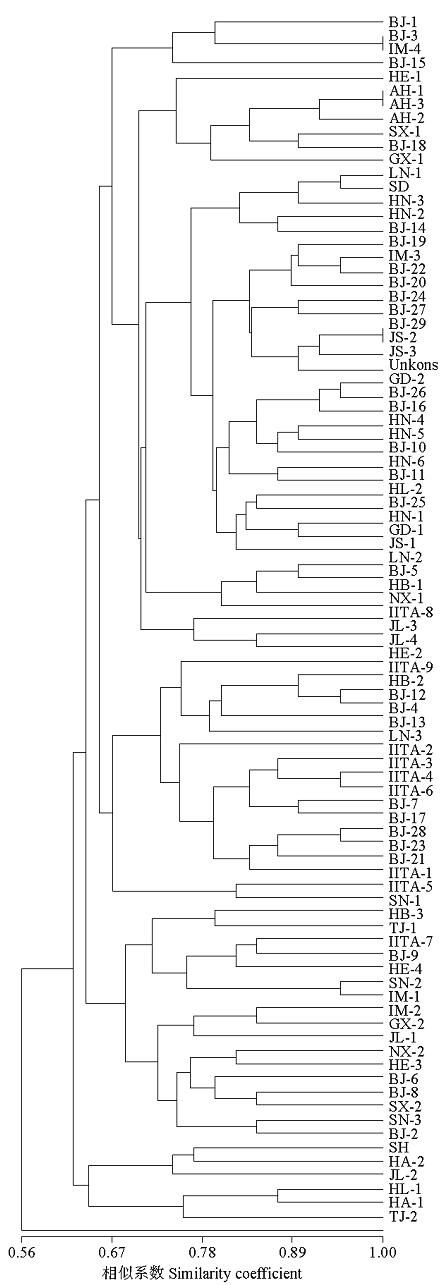
UPGMA结果显示,除AH-1与AH-3、BJ-4与IM-4新品系、及BJ-11与HN-4 3对种质外,其余种质均能被有效区分(图1)。
图1
按照遗传相似系数,可将88份普通豇豆应用核心种质分为4个类群。其中第一类群含46份种质,包括来自中国江苏、安徽、山东、湖南和广东的所有种质,及来自中国北京、吉林、辽宁和内蒙古等的部分种质;第二类群包括18份种质,主要来自中国北京(8份)和IITA(7份),另外3份分别来自中国湖北、河北和辽宁;第三类群包括17份种质,除中国广西、湖北和IITA各1份外,其余均来自我国北方各地的种质;第四类群仅包括7份种质,来自中国北京、河南、黑龙江、上海和天津,其中来自中国天津种质TJ-2的遗传背景与其他种质差异最大。从聚类图(图1)可以看出,大部分来源一致的种质有聚在一起的趋势。
3 讨论
SSR分子标记因其共显性、可重复性、精确性和多态性丰富等特点,已被广泛应用于动植物种内[17]、种间[18]的多样性及品种特征鉴定[19]等分析中。普通豇豆是我国主要的栽培豆种之一,保存有3 000多份种质资源,但其育种及基础研究相对薄弱,资源利用率低。因此,开展普通豇豆种质资源的SSR分析,将有效提高种质资源在遗传研究和育种利用中的效率。国内外豇豆种质资源均有丰富的表型变异类型[20,21],然而本研究中平均SSR等位变异数仅为2.79个,甚至有3对种质不能被有效区分开,远低于长豇豆[2]和大豆[22]等豆科作物,但与徐雁鸿等[14]对316份国内外普通豇豆及其近缘作物绿豆[23]和小豆[24]等的研究结果相对一致。豇豆属作物较低的SSR变异水平不利于图谱构建及基因定位[25,26]。随着普通豇豆全基因组序列的释放[27],DNA分子标记开发更加便利,而SSR作为操作简单、基因组分布广泛的DNA分子标记之一,在多样性分析及图谱构建中具有独特优势。不同SSR位点的变异水平差异较大[28],因此,有必要进一步开发并筛选多态性高的SSR标记,以满足遗传连锁图谱的加密及基因定位等研究需要。
本研究构建了88份核心豇豆种质资源的SSR指纹图谱,为这些种质的杂交组配及标记辅助选择提供可靠信息,遗传多样性分析结果可作为资源考察、收集和引进等的参考。本研究发现有3对种质在14个SSR位点的指纹一致,而且大部分SSR标记对种质的区分能力有限。因此,需要加强多态性高的SSR引物的筛选,建立一套更有效的核心SSR标记,提高标记辅助选择的效率。
4 结论
除3对种质外,其余85份普通豇豆种质均可以被14对SSR引物有效区分,且聚类结果显示来源相同的种质大多聚在一起,但是14对SSR引物的平均等位变异数相对较低。不同SSR位点的变异水平差异较大,需进一步开发并筛选多态性高的SSR标记来满足基因遗传定位研究等要求。
参考文献
Determining genetic similarities and relationships among cowpea breeding lines and cultivars by microsatellite markers
RAPD variation in wild:weedy and cultivated azuki beans in Asia
Genetic mapping of a new set of microsatellite markers in a reference common bean (Phaseolus vulgaris) population BAT93×Jalo EEP558
Genetic diversity of Chinese cultivated soybean revealed by SSR markers
Genetic diversity assessment of a set of introduced mung bean accessions (Vigna radiata L.)
A genetic linkage map for azuki bean [Vigna angularis (Willd.) Ohwi & Ohashi]
The genome of cowpea (Vigna unguiculata [L.] Walp.)
Genetic relationship of cowpea (Vigna unguiculata) varieties from Senegal based on SSR markers