基因表达分析是阐明植物生命周期中遗传、信号传导及代谢途径等复杂调控过程的重要工具[4]。目前,转录组测序技术已成为分析生物体基因转录表达水平的普遍方法之一[5],且已广泛应用于忍冬[6]、木兰[7]和款冬花[8]等植物花香物质代谢通路的分子机理研究。近年来,桔梗的研究主要集中在栽培应用、药用成分检测及功能分析等方面[9]。因此,对桔梗花发育过程中的转录组学研究尤为重要。本研究以桔梗白花为对照,研究不同发育时期的桔梗紫花呈色差异表达基因。采用高通量测序平台对紫花桔梗花瓣进行转录组测序分析,比较研究紫花花瓣不同时期的基因差异表达情况,经基因功能注释和代谢通路富集,筛选出差异表达基因,研究结果可为后期开展影响桔梗花色的关键基因深入挖掘和功能研究提供科学参考。
1 材料与方法
1.1 试验材料
试验材料为多年生桔梗白花(B)和紫花(Z),由延边大学作物育种实验室保存。于2023年8月晴朗天气选取无病虫害、生长旺盛的鲜花,擦除花上灰尘和水珠后,按花苞期(A)、转色期(B)、开放期(C)进行采样,各设3次重复(图1)。采取的样品经液氮速冻固定,研磨混匀,分装入10 mL离心管并编号,随后转移至-80 ℃冰箱保存,用于后续转录组测序及RT-qPCR验证试验。
图1
图1
不同发育时期的桔梗紫花和白花
Fig.1
Purple and white flowers of P.grandiflorus at different development stages
1.2 RNA的提取与质检
根据Genstar公司提供的操作方案分离和纯化总样品的RNA。用NanoDrop ND-2000对总RNA质控。再通过Agient2100/LabChip GX检测RNA的完整性,同时通过琼脂糖电泳的方法进行验证。
1.3 转录组文库的构建及测序
样品检测合格后,进行文库构建,主要流程如下:
(1)用带有Oligo(dT)的磁珠富集真核生物mRNA;(2)加入Fragmentation Buffer将mRNA进行随机打断;(3)以mRNA为模板,合成第一条cDNA链及二链,并进行cDNA纯化;(4)纯化的双链cDNA再进行末端修复、加A尾并连接测序接头,然后用AMPure XP beads进行片段大小选择;(5)最后通过PCR富集得到cDNA文库。
文库质检合格后,使用Illumina NovaSeq6000测序平台进行PE150模式测序。
1.4 分析流程
测序数据下机后,使用百迈客云平台BMKCloud(www.biocloud.net)提供的生物信息学分析流程进行数据分析。将Clean Data进行序列组装,获得该物种的Unigene库。基于此,进行文库质量评估、结构水平分析、差异表达分析、GO、KEGG富集分析等。
1.5 测序样品的相关性分析
采用Spearman相关性分析来评估各个样品的3次重复之间结果的可靠性。
1.6 实时定量聚合酶链式反应(quantitative real- time polymerase chain reaction,qRT-PCR)验证
选取11个明显富集到苯丙酸代谢、类黄酮生物合成、苯丙氨酸代谢、花青素生物合成途径的基因,进行qRT-PCR分析,验证转录组数据。利用Primer 3 plus设计引物(表1),参照GenStar公司的2×RealStar Green Fast Mixture试剂盒说明书进行qRT-PCR检测。扩增体系为20 μL(1 μL cDNA,上、下游引物各0.5 μL,10 μL 2×RealStar Green Fast Mixture,8.0 μL ddH2O)。反应程序:95℃ 60 s;95℃ 10s,60℃ 30 s,40个循环。以Actin为内参基因,每个取样时间点设3次生物学重复,每个样品设置3次技术重复。使用2-ΔΔCt方法进行表达量的计算,用SPSS 20.0进行统计分析,用Graph Pad Prism 8软件进行图像绘制。
表1 qRT-PCR引物序列
Table 1
| 基因ID Gene ID | 正向引物Forward primer (5'-3') | 反向引物Reverse primer (5'-3') |
|---|---|---|
| Actin | CATCCACGAGACCACCTACAA | CGAGCCACCAATCCAAACA |
| TRINITY_DN23720_c0_g2 | AATGCTCAATCAGTCCCTG | CTAACCGTATTCCCGTCTT |
| TRINITY_DN2567_c0_g1 | TAGTAGCGGCAGCAGAAGC | TTCATCAACAACGGCATCA |
| TRINITY_DN967_c1_g1 | AGTATAGGTTATGCCTGTGG | TTATGGGATTGATGCTCTT |
| TRINITY_DN2406_c1_g1 | GTTTGCCAATTATGGACTC | AACCCAATTCGTTCTGTTA |
| TRINITY_DN3035_c0_g1 | CCAAGAGTGAGGTCTGGCTGAG | GGGCGGTGACGGAGAAGTA |
| TRINITY_DN37472c0_g1 | CATTGGCTCGTGGTTAGTC | AAGTTGGTGTCAGCTTTCG |
| TRINITY_DN5772_c0_g1 | GGAATCGGAATGGTTGAGT | TTGGCTAGGCAGTGTAAGC |
| TRINITY_DN6749_c0_g1 | ATGTGGAGACGGAACGACG | TGACAGCCCTGGGAAAGAA |
| TRINITY_DN8143_c2_g1 | TGCCCAATCCAATTATACCA | ACCGACGAAGTTGCTGACTA |
| TRINITY_DN8697_c0_g1 | TGAGCCTTCAGTTTCTTCC | GGGTGCCTTTGATTGTTAG |
| TRINITY_DN3637_c0_g1 | TTCGGCAACAAGTATGAAA | TCCAGTCCTCCTCTTAGCA |
2 结果与分析
2.1 转录组测序质量分析
使用百迈克云平台对桔梗白花和紫花的花苞期、转色期、开放期进行高通量转录组测序。结果见表2,经过测序质量控制,共得到187.22 Gb质量控制后的数据,每个cDNA文库有效序列占比均在90%以上;每组样本碱基质量值≥20(Q20)均在99%以上;碱基质量值≥30(Q30)均在97%以上;GC碱基含量在43%~45%,说明样品测序质量较好,碱基组成平衡。以上结果表明测序数据质量和数量满足后续分析的要求。
表2 测序数据分析汇总表
Table 2
| 样本ID Sample ID | 读长总数Read sum | 碱基总数Base sum | GC碱基含量GC base content (%) | Q20 (%) | Q30 (%) |
|---|---|---|---|---|---|
| BA1 | 22673594 | 6791955690 | 43.69 | 99.64 | 97.86 |
| BA2 | 23522001 | 7046338862 | 44.34 | 99.62 | 97.75 |
| BA3 | 20665494 | 6190548036 | 43.94 | 99.68 | 98.12 |
| BB1 | 19419289 | 5817024270 | 43.96 | 99.64 | 97.86 |
| BB2 | 21363691 | 6399577508 | 44.15 | 99.62 | 97.73 |
| BB3 | 20202020 | 6051810874 | 44.07 | 99.66 | 98.03 |
| BC1 | 21679804 | 6492693228 | 42.67 | 99.60 | 97.60 |
| BC2 | 22115453 | 6624717198 | 43.30 | 99.63 | 97.80 |
| BC3 | 25825530 | 7734409312 | 42.96 | 99.65 | 97.95 |
| ZA1 | 23120475 | 6924725334 | 43.28 | 99.68 | 98.17 |
| ZA2 | 24396786 | 7306725610 | 43.76 | 99.69 | 98.22 |
| ZA3 | 24180417 | 7241221932 | 43.22 | 99.66 | 98.03 |
| ZB1 | 24876074 | 7451447804 | 43.91 | 99.66 | 98.02 |
| ZB2 | 21070788 | 6312223716 | 44.02 | 99.65 | 97.93 |
| ZB3 | 22153286 | 6636874790 | 44.23 | 99.61 | 97.71 |
| ZC1 | 22804663 | 6831635192 | 43.71 | 99.66 | 97.97 |
| ZC2 | 22750568 | 6815684020 | 43.69 | 99.61 | 97.64 |
| ZC3 | 25896801 | 7757837316 | 43.87 | 99.69 | 98.20 |
2.2 转录组测序数据组装
如表3所示,N50长度的非冗余序列为2542,组装完整性较高,1000 bp以下的基因数占比49.12%,1000~2000 bp的基因数占比23.75%,2000 bp以上的基因数占比27.13%。通过Trinity 测序,共拼接得到201 809条转录本,平均长度为1503 bp,上述结果表明,组装完整性与测序数据质量均较高,可充分满足后续分析要求。
表3 组装结果统计
Table 3
| 所有组合转录本长度 All combination transcripts length (bp) | 总数 Total number | 百分比 Percentage (%) |
|---|---|---|
| 100~300 | 25 306 | 12.54 |
| 300~500 | 31 668 | 15.69 |
| 500~1000 | 42 152 | 20.89 |
| 1000~2000 | 47 928 | 23.75 |
| >2000 | 54 755 | 27.13 |
| 总数Total number | 201 809 | |
| 总长度Total length | 303 409 855 | |
| N50长度N50 length | 2452 | |
| 平均长度Mean length | 1503 |
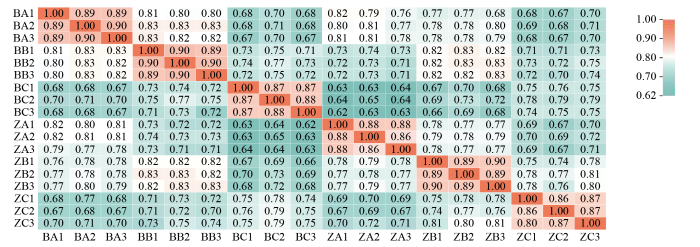
2.3 转录组测序样品的相关性分析
对样本进行Spearman相关性分析,相关系数(R2)越接近1,表明样品之间表达模式的相似度越高,R2≥0.80说明高度相关,0.60<R2<0.80说明中度相关,结果见图2。每个样品间的R2基本都接近0.80,表明样品间表达模式相似度高,可用于后续试验。
图2
2.4 差异表达基因数目统计
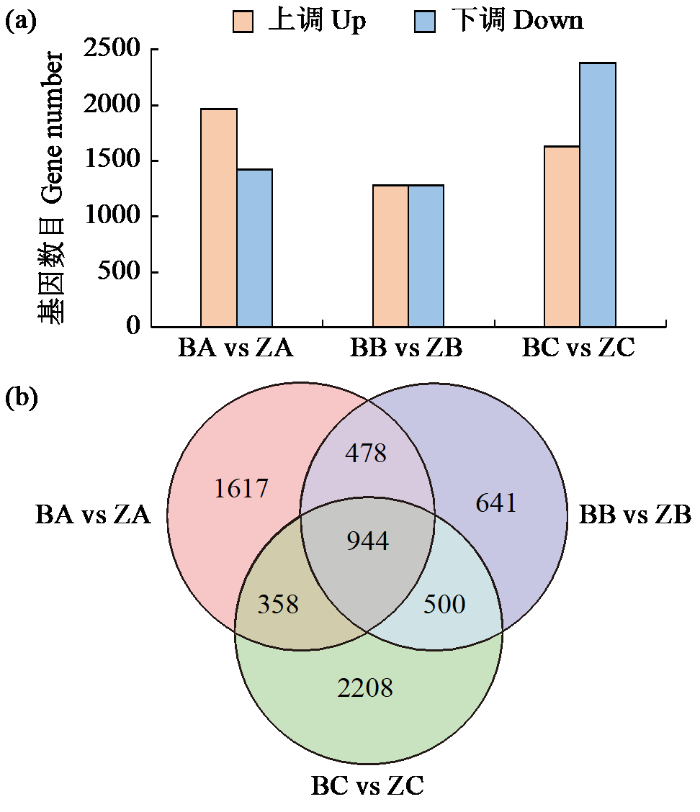
基于基因在各样本中的Count值,使用差异分析软件进行差异表达基因筛选,对于有生物学重复的差异分组,用DESeq2软件进行差异分析;对于没有生物学重复的差异分组,使用EBSeq软件进行差异分析。在差异表达基因检测过程中,将差异倍数(fold change)≥2且发现错误率(FDR)<0.01作为筛选标准。并用柱状图直观地展示整体的上下调数量,结果如图3a所示,BA vs ZA中,有3397个差异表达基因(DEGs)响应差异表达,其中,上调1970个,下调1427个;BB vs ZB中,有2563个DEGs响应差异表达,其中,上调1282个,下调1281个;BC vs ZC中,有4010个DEGs响应差异表达,其中,上调1630个,下调2380个。
图3
图3
差异表达基因数目统计
(a) 各比较组差异表达基因数目统计柱状图;(b) 各比较组差异表达基因韦恩图。
Fig.3
Statistics on the number of differentially expressed genes
(a) Histogram of the number of differentially expressed genes in each comparison group; (b) Venn diagram of differentially expressed genes for each comparison group.
通过图3b可以看出,获得至少在一次比较中为差异表达基因共9970个,其中944个DEGs为3个时期共有,在花朵开放过程中持续表达,花苞期(A)特有DEGs为1617个、转色期(B)特有DEGs为641个、开放期(C)特有DEGs为2208个。
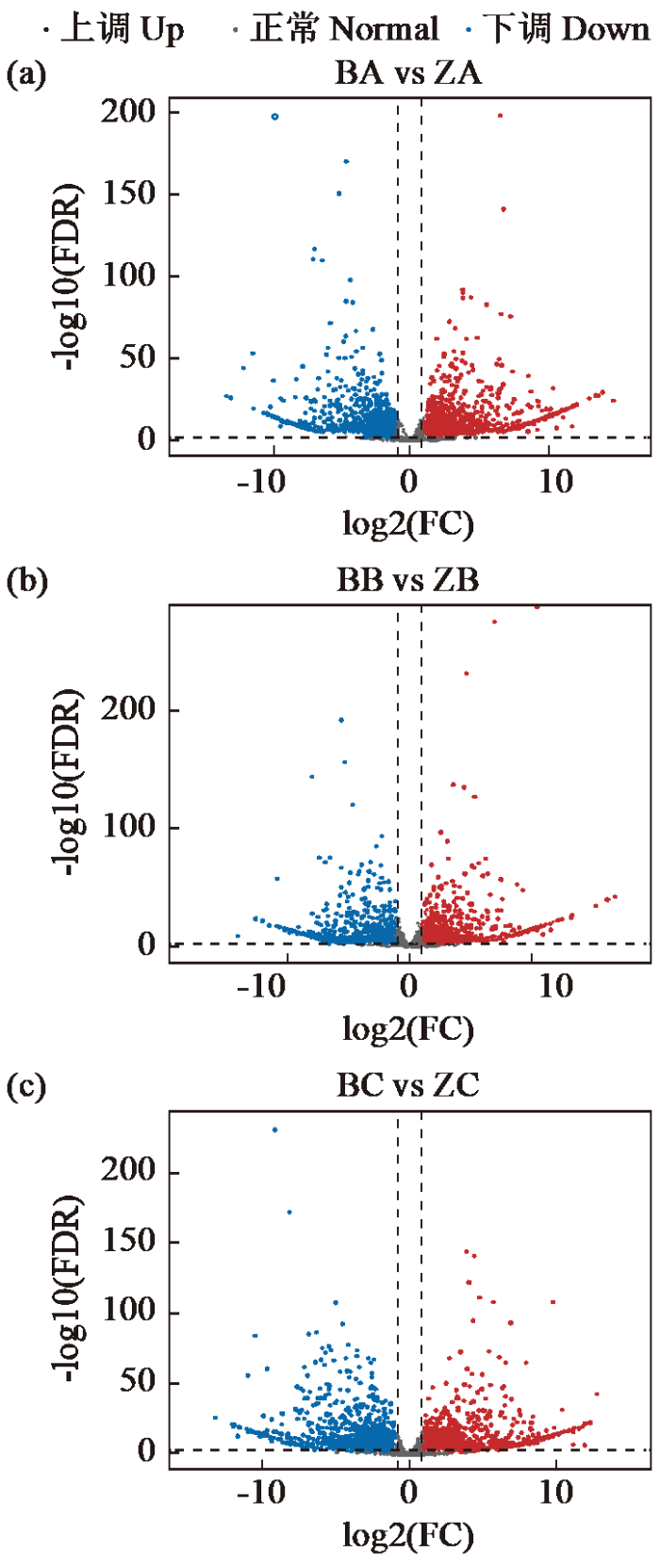
2.5 差异基因表达水平火山图
利用火山图可以了解不同表达基因的总体分布,如图4所示,横轴表示各样品间基因的差异表达倍数,纵轴表示各基因表达量的变化统计学显著性。3个比较组中红点代表显著上调表达的基因,蓝点代表显著下调表达的基因,由此可见,本研究选取的转录组数据包含大量具备统计学意义的显著差异基因,且上调和下调数量差异较小。同时花朵生长发育过程中DEGs数量先降低再升高,在开放期(C)DEGs数量最多。
图4
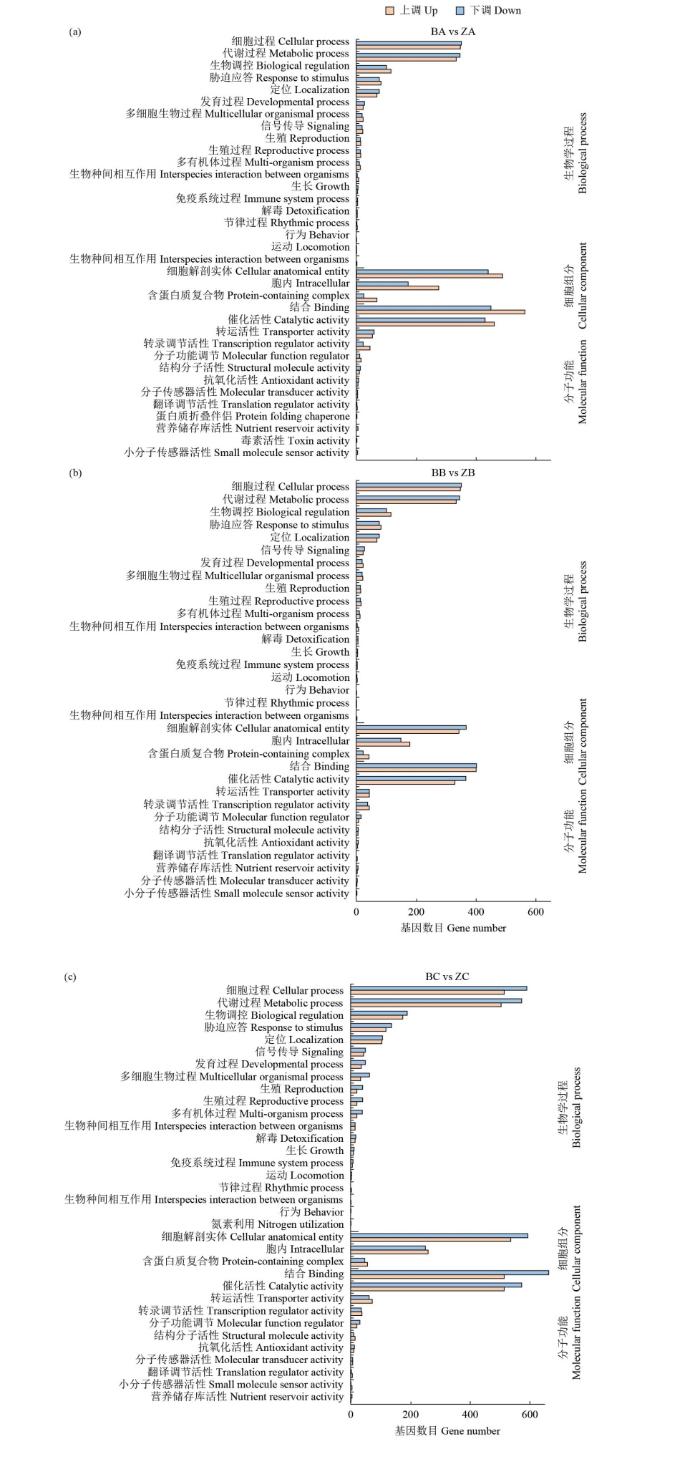
2.6 DEGs的GO富集分析
为了探究桔梗生长发育过程中花色形成相关差异表达基因的生物学功能,对3个时期鉴定到的特有DEGs进行GO功能注释。GO数据库是GO组织(gene ontology consortium)在2000年构建的一个结构化的标准生物学注释系统,旨在建立基因及其产物知识的标准词汇体系,对于各物种都适用。GO注释系统是一个有向无环图,包含3个主要分支,即生物学过程(biological process)、分子功能(molecular function)和细胞组分(cellular component)。
如图5所示,进一步对BA vs ZA、BB vs ZB、BC vs ZC特有DEGs进行GO富集程度分析,以发P<0.05为条件对GO条目进行筛选,共获得45个显著富集的GO Term,其中生物学过程功能23个、细胞组分6个、分子功能16个。在生物学过程(biological process)中细胞过程途径的差异基因最多,3个时期上调基因分别为507、347、516个;下调基因分别为416、351、593个。分子功能中结合途径的差异基因最多,3个时期上调基因分别为562、399、515个;下调基因分别为448、401、664个。细胞组分中细胞途径的差异基因最多,3个时期上调基因分别为489、343、536个,下调基因分别为438、367、597个。
图5
特有的DEGs的GO富集数据表明,在细胞组分类别中,主要富集在细胞、含蛋白质复合物、胞内等GO注释中;在生物学过程类别中,主要富集在细胞过程、代谢过程、生物调控等GO注释中,表明花色形成起始于相关基因在细胞内开始催化、转运的功能,为生物学功能提供物质基础;在分子功能类别中,主要富集在结合、催化活性、转运活性等GO注释中,细胞代谢旺盛,并在细胞内形成花色形成所需物质,保证花朵呈色。在花色相关细胞的形成过程中,花青素是植物次生代谢产物,糖苷衍生物。因此推测,归类于代谢过程和催化活性的DEGs,可能为参与桔梗花青素生物合成差异相关的良好候选基因。
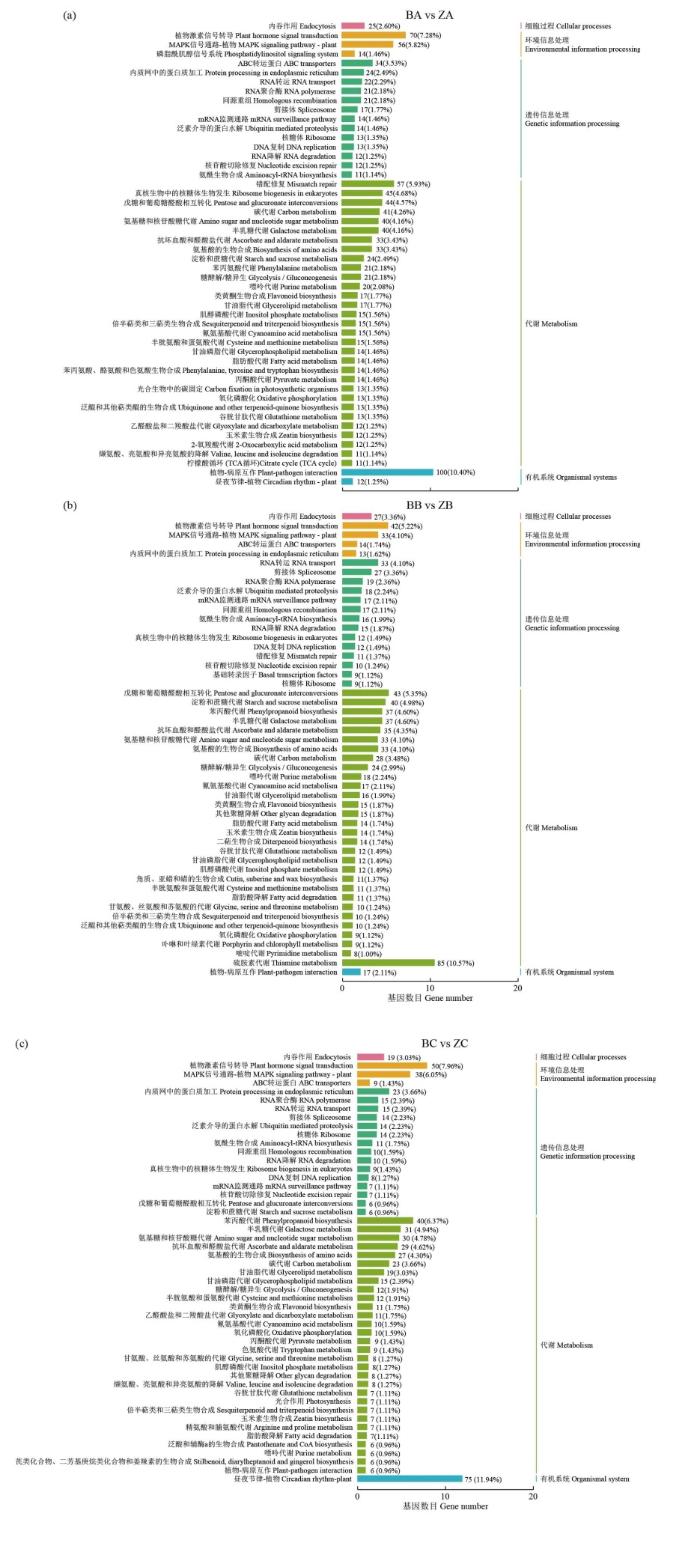
2.7 DEGs的KEGG富集分析
如图6所示,在KEGG数据库中BA vs ZA特有DEGs共有804个注释到125个通路上,BB vs ZB特有DEGs共有628个注释到120个通路上。BC vs ZC特有DEGs共有1580个注释到127个通路上。DEGs被注释到以下5大类通路中,分别是细胞过程、环境信息处理、遗传信息处理、代谢过程、有机系统。
图6
以P<0.05为条件对KEGG通路进行筛选,特有DEGs显著富集的通路包括植物―病原互作(ko04626)、植物激素信号转导(ko04075)、戊糖和葡萄糖醛酸相互转化(ko00040)、碳代谢(ko01200)、淀粉和蔗糖代谢(ko00500)等。是植物体内最重要的基础代谢,为类黄酮代谢途径和花青素合成提供大量能量,同时也为桔梗花朵各生长发育时期生理生化进程做准备。
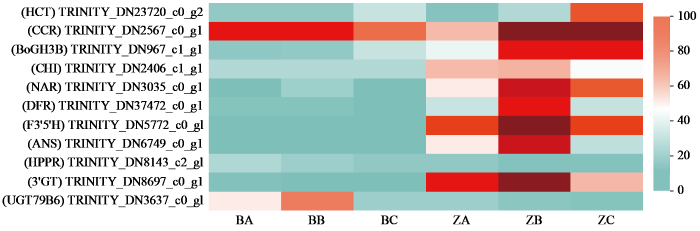
通过KEGG富集分析,筛选出各时期比较组与花色有关通路及持续表达基因,推测以下基因与桔梗紫花花色形成有关(表4)。
表4 各时期比较组花色关键结构基因差异表达
Table 4
| 代谢通路 Pathway ID | 通路名称 Name of pathway | 基因ID Gene ID | 基因名称 Gene name | 详细描述 Detailed description | 表达 Expression |
|---|---|---|---|---|---|
| ko00940 | 苯丙酸代谢 | TRINITY_DN23720_c0_g2 | HCT | 羟基肉桂酰辅酶A莽草酸酯 | 上调 |
| TRINITY_DN2567_c0_g1 | CCR | 肉桂酰辅酶A还原酶 | 上调 | ||
| ko00941 | 类黄酮生物合成 | TRINITY_DN967_c1_g1 | β-葡萄糖苷酶BoGH3B | β-葡萄糖苷酶BoGH3B | 上调 |
| TRINITY_DN2406_c1_g1 | CHI | 查尔酮―黄酮异构酶 | 上调 | ||
| TRINITY_DN3035_c0_g1 | NAR | 柚皮素,2-氧代戊二酸3-双加氧酶 | 上调 | ||
| TRINITY_DN37472_c0_g1 | DFR | 二氢黄酮醇4-还原酶 | 上调 | ||
| TRINITY_DN5772_c0_g1 | F3′5′H | 类黄酮-3',5'-羟化酶 | 上调 | ||
| TRINITY_DN6749_c0_g1 | ANS | 花青素合酶 | 上调 | ||
| ko00360 | 苯丙氨酸代谢 | TRINITY_DN8143_c2_g1 | HPPR | 羟苯基丙酮酸还原酶 | 下调 |
| ko00942 | 花青素生物合成 | TRINITY_DN8697_c0_g1 | 3'GT | 花青素3-O-葡萄糖基转移酶 | 上调 |
| TRINITY_DN3637_c0_g1 | UGT79B6 | UDP-糖基转移酶79B6 | 下调 |
如图7,通过筛选出的各时期关键基因的表达量,可以看出TRINITY_DN5772_c0_g1(F3′5′H基因)在桔梗紫花转色期表达量最高,为紫花呈色提供大量前体物质。
图7
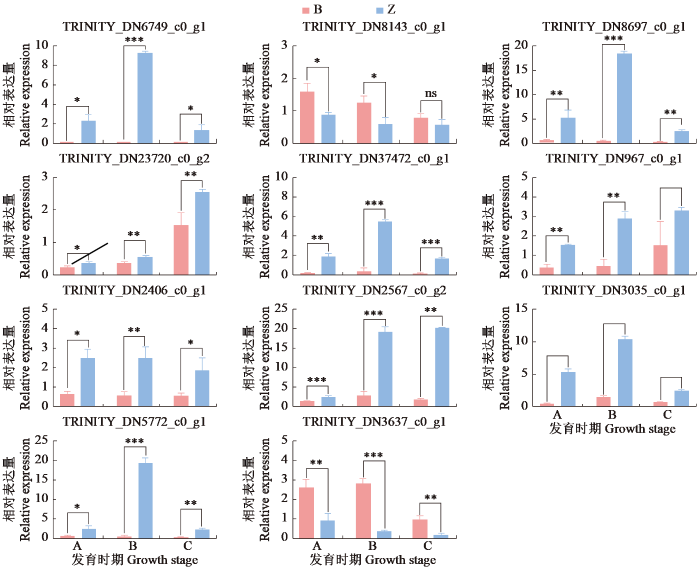
2.8 qRT-PCR结果分析
为了确认测序分析中获得的转录物,进一步验证转录组测序数据的准确性,选取了上述总结的11个差异基因进行qRT-PCR验证(图8)。将11组数据的实时荧光定量鉴定结果与转录组数据(RNA-seq)进行比较,结果表明,这些基因的表达趋势与转录组测序结果基本一致,说明qRT-PCR数据和转录组测序数据之间具有良好的一致性,本研究中用于花青素合成相关基因分析的转录组数据是可靠的。
图8
图8
差异基因实时荧光定量结果
“*”、“**”和“***”分别代表同一比较组中白花和紫花在P < 0.05、P < 0.01和P < 0.001水平上差异显著。
Fig.8
qRT-PCR results of differential genes
“*”,“**”and“***”indicate that white flowers and purple flowers in the same comparison group were significantly different at P < 0.05, P < 0.01, and P < 0.001 levels, respectively.
3 讨论
3.1 花青素生物合成途径
第二阶段由查尔酮合成酶(chalconesynthase,CHS)催化丙二酰辅酶A和香豆酰辅酶A生成查尔酮,随后,查尔酮异构酶(chalcone isomerase,CHI)将查尔酮异构成三羟基黄烷酮,黄烷酮-3-羟化酶(flavanone-3-hydroxylase,F3H)将三羟基黄烷酮进一步羟基化形成二氢黄酮醇。二氢黄酮醇在不同酶的催化作用下生成不同的物质,其中一部分通过类黄酮-3′-羟化酶(flavonoid-3′- hydroxylase,F3′H)将B环3′位羟基化生成矢车菊素的前体物质二氢槲皮素,还有一部分通过类黄酮-3′,5′-羟化酶(flavonoid-3′,5′-hydroxylase,F3′5′H)将B环3′,5′位置羟基化,生成飞燕草素的前体物质二氢杨梅黄酮[14-15]。
第三阶段二氢黄酮醇、二氢槲皮素和二氢杨梅黄酮在二氢黄酮醇还原酶(dihydroflavonol-4- reductase,DFR)和花青素合成酶(anthocyanidin synthase,ANS)作用下形成有色花青素,在植物细胞中花青素通常不能以单体的形式存在,因此他们往往会与糖苷结合转化成稳定的花青素苷[13]。
本试验也探究出与花色形成有关的通路,如苯丙酸代谢、类黄酮生物合成、苯丙氨酸、酪氨酸和色氨酸的生物合成、苯丙氨酸代谢及花青素生物合成等。还有甜菜色素生物合成、类胡萝卜素生物合成、异黄酮生物合成等通路被发现,但只有少量差异表达基因富集。
3.2 花青素生物合成关键酶
类黄酮类、类胡萝卜素类和生物碱类是自然界中主要的三大色素类群,类黄酮物质中的花青素苷是绝大多数观赏植物重要的呈色物质之一[16]。而PAL、CHS、CHI、DFR、F3H和4CL均是植物花青素生物合成路径中的关键酶,在花青素生物合成方面发挥重要作用,影响着植物花色的变化[17]。本研究发现CHI、DFR、F3′5′H、ANS等关键酶基因影响桔梗紫花呈色。王冲等[18]在解析大花君子兰花瓣花青素代谢差异中发现,主要富集与花青素合成相关的通路,包括苯丙素生物合成途径、类黄酮生物合成途径、异黄酮生物合成途径、花青素生物合成途径和苯丙氨酸代谢生物合成途径。陈家龙等[19]研究表明,在花青苷生物合成途径中,与白花相比,粉花中C4H、CHS、CHI和DFR的表达量显著增加,F3H和LDOX的表达量显著降低,而HCT和F3'H的同源基因有的表达上调,有的下调。这一结果表明C4H、CHS、CHI以及DFR可能是导致粉花品种花色呈色的关键基因,而DFR基因专门用于合成花青苷[20]。
3.3 蓝紫色花形成的关键酶与代谢产物
3.4 内参基因选择
4 结论
综上,通过比较分析并挖掘桔梗紫花的差异基因表达,为后续研究桔梗紫花花色特异基因表达调控及分析相关基因功能提供一定的参考价值。通过筛选紫花特异且高表达的花青素生物合成基因。在桔梗白花与紫花对比中,KEGG分析结果表明,苯丙酸代谢、类黄酮生物合成、花青素生物合成途径中差异基因高度表达与桔梗紫花花色形成有关;共筛选出9条关键酶基因(HCT、CHI、DFR、F3′5′H、ANS、3'GT、CCR、NAR、HPPR)以及2个糖基酶基因(β-葡萄糖苷酶BoGH3B和UGT79B6),持续表达影响桔梗紫花花色形成,为桔梗花青素生物合成途径关键酶基因进一步研究提供了依据,也为将来花青素生物合成途径的解析及调控机制的研究奠定了基础。
参考文献
Comprehensive development and utilization of wild Platycodon
Reserach progress in the use diversity and directive breeding of Platycodon grandiflorum
Identification of reliable reference genes for quantitative real-time PCR normalization in pitaya
De novo transcriptome analysis of Osmanthus serrulatus Rehd. flowers and leaves by Illumina sequencing
Integrated metabolic profiling and transcriptome analysis of pigment accumulation in Lonicera japonica flower petal during colour-transition
Integrated metabolome and transcriptome analysis of Magnolia champaca identifies biosynthetic pathways for floral volatile organic compounds
DOI:10.1186/s12864-017-3846-8
PMID:28615048
[本文引用: 1]

Background: Magnolia champaca, commonly known as champak is a well-known tree due to its highly fragrant flowers. Champak floral scent is attributed to a complex mix of volatile organic compounds (VOCs). These aromatic flowers are widely used in flavors and fragrances industry. Despite its commercial importance, the VOC biosynthesis pathways in these flowers are largely unknown. Here, we combine metabolite and RNA sequencing (RNA-seq) analyses of fully opened champak flowers to discover the active VOC biosynthesis pathways as well as floral scent-related genes.Results: Volatile collection by headspace method and analysis by gas chromatography-mass spectrometry (GC-MS) identified a total of 43 VOCs from fully opened champak flowers, of which 46.9% were terpenoids, 38.9% were volatile esters and 5.2% belonged to phenylpropanoids/benzenoids. Sequencing and de novo assembly of champak flower transcriptome yielded 47,688 non-redundant unigenes. Transcriptome assembly was validated using standard polymerase chain reaction (PCR) based approach for randomly selected unigenes. The detailed profiles of VOCs led to the discovery of pathways and genes involved in floral scent biosynthesis from RNA-seq data. Analysis of expression levels of many floral-scent biosynthesis-related unigenes in flowers and leaves showed that most of them were expressed higher in flowers than in leaf tissues. Moreover, our metabolite-guided transcriptomics, in vitro and in vivo enzyme assays and transgenic studies identified (R)-linalool synthase that is essential for the production of major VOCs of champak flowers, (R)-linalool and linalool oxides.Conclusion: As our study is the first report on transcriptome analysis of Magnolia champaca, this transcriptome dataset that serves as an important public information for functional genomics will not only facilitate better understanding of ecological functions of champak floral VOCs, but also provide biotechnological targets for sustainable production of champak floral scent.
款冬花不同发育阶段的代谢组学和比较转录组学分析
DOI:10.13865/j.cnki.cjbmb.2017.06.12
[本文引用: 1]

以不同发育阶段款冬花(发育初期、中期、中后期、解封期及花朵期)为对象,基于核磁共振的代谢组学技术,分析其次生代谢物种类及含量的合成累积规律,并进行高通量转录组学测序,从差异表达的基因中寻找次生代谢物生物合成的关联酶基因。代谢组学分析发现,款冬花不同发育阶段的次生代谢物代谢组成明显不同,苯丙素类成分在发育初期至中后期含量较高,之后逐渐降低;黄酮类成分(芦丁、山奈酚)在发育的各个阶段含量均有波动,但总体变化不大。转录组学分析结果显示,与苯丙素类成分生物合成相关的酶基因(COMT、HCT),随着花蕾的发育表达量逐渐降低;与黄酮类成分合成相关的酶基因(FLS、F3H、DFR),其表达量在不同阶段变化不大。转录组测序中的相关酶基因表达量同次生代谢物成分的变化趋势基本一致。本文采用的代谢组学和转录组学结合的方式,对款冬花的次生代谢物累积规律进行分析,为今后款冬花次生代谢物的生物合成调控研究奠定了基础。
中草药桔梗人工栽培研究进展
DOI:10.11923/j.issn.2095-4050.cjas16030021
[本文引用: 1]

对目前国内桔梗种植中种质资源分布、种质资源创新、桔梗栽培模式和关键技术理论以及药用价值等研究进展情况进行综述,并对未来桔梗的市场需求和发展前景进行展望。认为随着生活理念的变化,桔梗等药食兼用的功能性、保健型药用植物会成为一个新的热点,市场需求和社会效益会凸显,中医药与现代医学的相结合将引导中药材产业新的发展方向。
基于花青素苷合成和呈色机理的观赏植物花色改良分子育种
DOI:10.3864/j.issn.0578-1752.2016.03.011
[本文引用: 1]

花色是观赏植物最重要的品质性状之一,是植物自然进化过程中最具适应意义的表型性状,也是表观遗传学研究的重要内容。花青素苷是使花朵呈色的重要色素之一,被子植物中约有80%的科的花朵颜色由花青素苷决定;迄今从自然界分离和鉴定出的花青素苷多达600种,主要由6种花青素苷元衍生而来。花青素苷合成途径是迄今为止研究得最为清楚的植物次生代谢途径之一,它的合成首先取决于类黄酮代谢途径的生成,花青素苷种类的多样性则源于其不同分支途径的形成,在花青素苷元基本骨架上不同位置取代基的差异形成了多种多样的花青素苷。在花青素苷生物合成过程中,分支点酶的竞争机制和关键酶的底物特异性使花青素苷的种类及相应的花色表型具有种属特异性。花青素苷合成后需要转运到液泡中被包裹成色素体,植物细胞中的液泡积累和贮存色素体的能力是影响花青素苷呈色的重要因素,因此,花青素苷在花瓣中的最终呈色还受液泡pH、助色素含量以及金属离子的络合作用等多种细胞内因素的影响。目前,已经在多种植物中获得了与花青素苷合成及呈色相关的结构基因和调节基因,并解析了其功能,成功获得了一些转基因花卉,但是这些基因调控表达的机制,包括转录水平和转录后水平的调控、DNA序列本身的差异和DNA甲基化修饰的调控机制等仍不清楚,转基因植株花色改良的程度也很有限,对于如何将这些机制应用于花色改良的转基因育种也是一个前沿的课题。花青素苷对园艺作物器官呈色机制的解析有助于对花朵呈色机制的理解,观赏植物中花色形成机理的研究对于园艺作物器官呈色机制的解析同样具有重要的参考价值。因此,本文以观赏植物为例,从花青素苷合成分支途径形成的机理、花青素苷生物合成途径的遗传调控机理以及影响花青素苷呈色的主要因素及其遗传调控机理3个方面,对影响植物花朵呈色的机制进行了综述,并对近年来基于花青素苷代谢和呈色机理的花色改良分子设计育种,尤其是国际上广泛关注的蓝色花育种进行了梳理和总结,以期为定向培育具有新奇花色的观赏植物新品种提供参考。
Recent advances in the transcriptional regulation of the flavonoid biosynthetic pathway
Genetics and biochemistry of anthocyanin biosynthesis
DOI:10.1105/tpc.7.7.1071 PMID:12242398 [本文引用: 1]
Diversity of anthocyanin and proanthocyanin biosynthesis in land plants
基于转录组测序的细风轮花青素合成途径及关键酶基因分析
DOI:10.7525/j.issn.1673-5102.2020.06.011
[本文引用: 1]

为进一步理解细风轮花青素合成途径,本研究利用华大基因BGISEQ-500平台对细风轮中的根、茎、叶、花4个组织进行了转录组测序,从头组装后得到128 856个Unigene。KEGG通路表明有40个Unigene编码了细风轮的花青素生物合成途径中6个关键酶。我们对其中的关键酶DFR(二氢黄酮醇还原酶)进行同源比对和空间结构模拟,结果显示DFR序列和结构均具有良好的保守性,且具有高度保守的NAD<sup>+</sup>结合位点,其二级结构主要由α螺旋和β折叠组成,在空间上α螺旋包裹着β折叠,形成“夹心饼干”样结构。
The F-box protein COI1 functions up stream of MYB305 to regulate primary carbohydrate metabolism in tobacco (Nicotiana tabacum L. cv. TN90)
Characteristics of α-Gal epitope, anti-Gal antibody, α-1, 3 galactosyltrans ferase and its clinical exploitation(Review)
糖基转移酶在花瓣色泽形成中的作用研究进展
DOI:10.16420/j.issn.0513-353x.2021-0636
[本文引用: 1]

糖基转移酶属于催化糖基转移反应的超基因家族的一类酶。糖基化修饰能丰富花青素的结构,调节其水溶性、稳定性和可转运性,决定植物色泽形成。对参与观赏植物花色素成分合成代谢的糖基转移酶的种类及其生化、分子生物学研究进行梳理,并对其在蓝紫色、橙色–紫红色以及黄色等主要花瓣色泽形成中的作用进行归纳,展望色泽相关糖基转移酶的研究方向,以期为深入解析花色素物质的糖基化修饰机理提供参考,也为观赏植物花色改良提供新思路。
Biochemical and molecular characterization of a novel UDP-glucose: anthocyanin 3'-O-glucosyltransferase, a key enzyme for blue anthocyanin biosynthesis from gentian
Cloning and characterization of the UDP-glucose: anthocyanin 5-O- glucosyltransferase gene from blue-flowered gentian
DOI:10.1093/jxb/ern031
PMID:18375606
[本文引用: 1]

Blue-flowered gentian (Gentiana triflora) is known to accumulate gentiodelphin, a unique polyacylated delphinidin-type anthocyanin, in the petals. Almost all of the structural genes involved in gentiodelphin biosynthesis have been isolated, but an important gene encoding UDP-glucose:anthocyanin 5-O-glucosyltransferase (5GT) remained to be identified. In this study, an attempt was made to isolate and characterize gentian 5GT, which is responsible for glucosylation of anthocyanidin 3-glucoside. A PCR-based cloning strategy identified seven 5GT candidates from gentian flowers. Among them, the deduced amino acid sequence of the 5GT gene from gentian petal cDNA, designated Gt5GT7, exhibited 36.0-41.7% identities with those of 5GTs from other plant species, and phylogenic analysis also suggested that Gt5GT7 belongs to the 5GT subfamily. The expression analysis showed that Gt5GT7 transcripts were detected predominantly in petals and weakly in filaments but not in leaves, stems, and other floral organs. In addition, increased levels of Gt5GT7 transcripts in petals coincided with flower development, a pattern identical to that of 5GT enzymatic activity as determined by in vitro assay using petal crude proteins. The substrate specificity of Gt5GT7 was analysed in vitro using the recombinant enzyme produced by Escherichia coli. Gt5GT7 could transfer a glucosyl moiety to anthocyanidin 3-glycosides but not to other flavonoid compounds. Delphinidin 3-glucoside, the precursor of gentiodelphin, was the best substrate among several anthocyanidin 3-glycosides tested. Heterologous expression of Gt5GT7 in tobacco plants led to additional accumulation of cyanidin 3-rutinoside-5-glucoside, confirming that Gt5GT7 has a valid enzymatic activity in planta.
Flower colour and cytochromes P450
Generation of blue chrysanthemums by anthocyanin B-ring hydroxylation and glucosylation and its coloration mechanism
Recent advances in the research and development of blue flowers
DOI:10.1270/jsbbs.17132
PMID:29681750
[本文引用: 1]

Flower color is the most important trait in the breeding of ornamental plants. In the floriculture industry, however, bluish colored flowers of desirable plants have proved difficult to breed. Many ornamental plants with a high production volume, such as rose and chrysanthemum, lack the key genes for producing the blue delphinidin pigment or do not have an intracellular environment suitable for developing blue color. Recently, it has become possible to incorporate a blue flower color trait through progress in molecular biological analysis of pigment biosynthesis genes and genetic engineering. For example, introduction of the '' gene encoding flavonoid 3',5'-hydroxylase can produce delphinidin in various flowers such as roses and carnations, turning the flower color purple or violet. Furthermore, the world's first blue chrysanthemum was recently produced by introducing the '' gene encoding anthocyanin 3',5'--glucosyltransferase, in addition to '', into the host plant. The B-ring glucosylated delphinidin-based anthocyanin that is synthesized by the two transgenes develops blue coloration by co-pigmentation with colorless flavone glycosides naturally present in the ray floret of chrysanthemum. This review focuses on the biotechnological efforts to develop blue flowers, and describes future prospects for blue flower breeding and commercialization.
Heterologous expression of Platycodon grandiflorus PgF3'5'H modifies flower color pigmentation in tobacco
An overview of Real- time quantitative PCR: applications to quantify cytokine gene expression
DOI:10.1006/meth.2001.1261
PMID:11846608
[本文引用: 1]

The analysis of cytokine profiles helps to clarify functional properties of immune cells, both for research and for clinical diagnosis. The real-time reverse transcription polymerase chain reaction (RT-PCR) is becoming widely used to quantify cytokines from cells, body fluids, tissues, or tissue biopsies. Being a very powerful and sensitive method it can be used to quantify mRNA expression levels of cytokines, which are often very low in the tissues under investigation. The method allows for the direct detection of PCR product during the exponential phase of the reaction, combining amplification and detection in one single step. In this review we discuss the principle of real-time RT-PCR, the different methodologies and chemistries available, the assets, and some of the pitfalls. With the TaqMan chemistry and the 7700 Sequence Detection System (Applied Biosystems), validation for a large panel of murine and human cytokines and other factors playing a role in the immune system is discussed in detail. In summary, the real-time RT-PCR technique is very accurate and sensitive, allows a high throughput, and can be performed on very small samples; therefore it is the method of choice for quantification of cytokine profiles in immune cells or inflamed tissues.Copyright 2001 Elsevier Science (USA).
Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis
DOI:10.1104/pp.105.063743
PMID:16166256
[本文引用: 1]

Gene transcripts with invariant abundance during development and in the face of environmental stimuli are essential reference points for accurate gene expression analyses, such as RNA gel-blot analysis or quantitative reverse transcription-polymerase chain reaction (PCR). An exceptionally large set of data from Affymetrix ATH1 whole-genome GeneChip studies provided the means to identify a new generation of reference genes with very stable expression levels in the model plant species Arabidopsis (Arabidopsis thaliana). Hundreds of Arabidopsis genes were found that outperform traditional reference genes in terms of expression stability throughout development and under a range of environmental conditions. Most of these were expressed at much lower levels than traditional reference genes, making them very suitable for normalization of gene expression over a wide range of transcript levels. Specific and efficient primers were developed for 22 genes and tested on a diverse set of 20 cDNA samples. Quantitative reverse transcription-PCR confirmed superior expression stability and lower absolute expression levels for many of these genes, including genes encoding a protein phosphatase 2A subunit, a coatomer subunit, and an ubiquitin-conjugating enzyme. The developed PCR primers or hybridization probes for the novel reference genes will enable better normalization and quantification of transcript levels in Arabidopsis in the future.
Housekeeping genes as internal standards: use and limits
Transference of function shapes organ identity in the dove tree inflorescence
DOI:10.1111/j.1469-8137.2011.03915.x
PMID:21992614
[本文引用: 1]

• An important evolutionary mechanism shaping the biodiversity of flowering plants is the transfer of function from one plant organ to another. To investigate whether and how transference of function is associated with the remodeling of the floral organ identity program we studied Davidia involucrata, a species with conspicuous, petaloid bracts subtending a contracted inflorescence with reduced flowers. • A detailed ontogeny enabled the interpretation of expression patterns of B-, C- and E-class homeotic MADS-box genes using qRT-PCR and in situ hybridization techniques. We investigated protein-protein interactions using yeast two-hybrid assays. • Although loss of organs does not appear to have affected organ identity in the retained organs of the reduced flowers of D. involucrata, the bracts express the B-class TM6 (Tomato MADS box gene 6) and GLOBOSA homologs, but not DEFICIENS, and the C-class AGAMOUS homolog, representing a subset of genes also involved in stamen identity. • Our results may illustrate how petal identity can be partially transferred outside the flower by expressing a subset of stamen identity genes. This adds to the molecular mechanisms explaining the diversity of plant reproductive morphology.© 2011 The Authors. New Phytologist © 2011 New Phytologist Trust.
茉莉花实时荧光定量PCR内参基因的筛选与验证
DOI:10.7668/hbnxb.20191401
[本文引用: 1]

为筛选适合茉莉花的内参基因,以5种组织(根、茎、嫩叶、成熟叶、花)和4个发育阶段的花(幼蕾期、膨大花蕾期、最长花蕾期和初绽期)为试验材料,选择较常见的8个候选内参基因进行引物特异性分析,结果显示,8个内参基因均扩增出单一条带,溶解曲线均只有明显的单一峰。采用qRT-PCR技术分析8个内参基因在不同样品中的表达量,结果显示,内参基因在不同样品中的表达量存在差异,各基因在花中的表达量均显著低于其他样品。利用geNorm、NormFinder和BestKeeper软件对基因的表达稳定性进行评价,并通过RefFinder对表达稳定性进行综合分析,结果表明,适用于不同组织(有花组)的最优内参基因为SAND和UPL7;适用于不同组织(无花组)的最优内参基因为UPL7和GAPDH;而适宜不同发育阶段花的最优内参基因为Actin和EF1α。进一步利用JsDXS和JsPAL2对筛选的内参基因进行验证,结果显示,以稳定性好的Actin、EF1α及Actin+EF1αJsDXS和JsPAL2在花发育不同阶段的表达水平的变化趋势基本一致,而用最不稳定的PP2A为参照时,花发育不同阶段的表达水平的变化趋势不一致。总之,本研究对茉莉花内参基因进行了筛选,不同组织(有花组)的最优内参基因为SAND和UPL7;不同组织(无花组)的最优内参基因为UPL7和GAPDH;花发育不同阶段的最优内参基因为Actin和EF1α。