作物杂志,2018, 第6期: 53–57 doi: 10.16035/j.issn.1001-7283.2018.06.009
内蒙古胡麻地方品种资源遗传多样性分析
伊六喜1,斯钦巴特尔1,高凤云1,周宇1,贾霄云1,陈明哲2,张辉1,贾海滨3
- 1 内蒙古自治区农牧业科学院生物技术研究中心,010031,内蒙古呼和浩特
2 锡林郭勒盟农业科学研究所,026000,内蒙古锡林浩特
3 乌兰察布市农牧业科学研究院,012000,内蒙古集宁
Genetic Diversity of Flax Germplasm Resources in Inner Mongolia
Yi Liuxi1,Siqin Bateer1,Gao Fengyun1,Zhou Yu1,Jia Xiaoyun1,Chen Mingzhe2,Zhang Hui1,Jia Haibin3
- 1 Biotechnology research center of the Inner Mongolia Academy of Agriculture and Husbandry Sciences, Huhhot 010031, Inner Mongolia, China
2 Xilinguole Meng Institute of Agricultural Sciences, Xilinhaote 026000, Inner Mongolia, China
3 Wulanchabu Academy of Agriculture and Animal Husbandry, Jining 012000, Inner Mongolia, China
摘要:
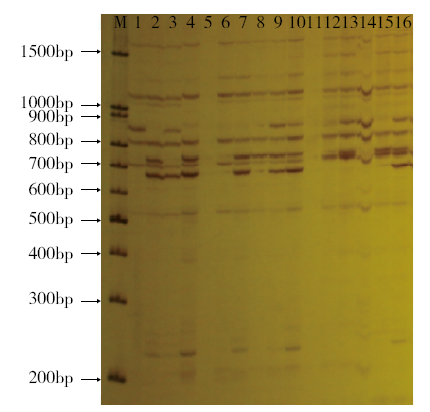
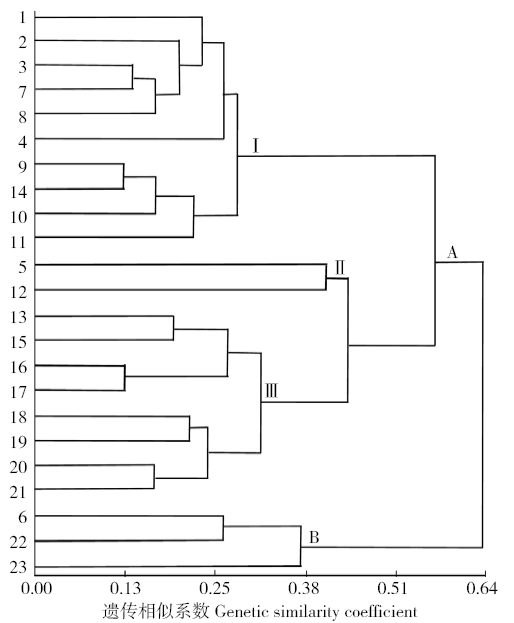
为了揭示内蒙古胡麻地方品种的遗传多样性和亲缘关系,采用SRAP分子标记对23份胡麻地方品种进行了遗传多样性分析。结果表明:胡麻25.0μL SRAP-PCR最佳反应体系为10μmol/L正反向引物0.3μL、2×Taq Master Mix 9.0μL、DNA模板量40ng;18对SRAP引物扩增得到219个条带,平均每对引物扩增的条带为12.17,多态性条带为136,多态性比率为62.10%,观测等位基因数为2.00,平均每个位点有效等位基因数1.59,多态性信息量(PIC)平均为0.61;有效等位基因数(Ne)、香农信息指数(I)和Nei’s遗传相似系数(H)分别为1.5630、0.6715和0.4738;在遗传相似系数0.55处供试胡麻分为两大类,在遗传相似系数0.38处,第一大类群分为3个亚群。结论认为,内蒙古胡麻地方品种的遗传多样性丰富,同一个地区培育的胡麻品种亲缘关系较近。
| [1] |
伊六喜, 斯钦巴特尔, 贾霄云 ,等. 胡麻种质资源、育种及遗传研究进展. 中国麻业科学, 2017,39(2):81-87.
doi: 10.3969/j.issn.1671-3532.2017.02.006 |
| [2] | 伊六喜, 斯钦巴特尔, 张辉 ,等. 胡麻种质资源遗传多样性及亲缘关系的SRAP分析. 西北植物学报, 2017,37(10):1941-1950. |
| [3] | 伊六喜, 萨如拉, 张辉 , 等. 胡麻种子产量与主要农艺性状的多重分析. 安徽农业科学, 2018,46(6):33-36. |
| [4] | 李明, 姜硕, 郑东泽 , 等. 亚麻SRAP标记连锁图谱的构建及3个数量性状的定位. 东北农业大学学报, 2014,45(2):12-18. |
| [5] |
安泽山, 严兴初, 党占海 , 等. 利用SRAP标记分析胡麻资源遗传多样性. 西南农业学报, 2014,27(2):530-534.
doi: 10.3969/j.issn.1001-4829.2014.02.013 |
| [6] | 伊六喜 , 斯钦巴特尔,张辉,等. 胡麻核心种质资源表型变异及SRAP分析. 中国油料作物学报, 2017,39(6):794-804. |
| [7] |
郝荣楷, 严兴初, 党占海 , 等. 我国胡麻育成品种的遗传多样性分析. 中国油料作物学报, 2014,36(3):334-342.
doi: 10.7505/j.issn.1007-9084.2014.03.007 |
| [8] |
吴建忠, 赵东升, 黄文功 , 等. 12个亚麻品种亲缘关系的SRAP分析. 中国麻业科学, 2012,34(4):153-157.
doi: 10.3969/j.issn.1671-3532.2012.04.001 |
| [9] | Stewart C, Via L . A rapid CTAB DNA isolation technique useful for RAPD fingerprinting and other PCR applications. Biotechniques, 1993,14(5):748-750. |
| [10] | Li G, Quiros C F . Sequence-related amplified polymorphism (SRAP),a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theoretical & Applied Genetics, 2001,103(2-3):455-461. |
| [11] | 孙翊, 张永春, 殷丽青 , 等. 朱顶红SRAP-PCR反应体系的建立和优化. 分子植物育种, 2017,15(5):1806-1813. |
| [12] |
Gresshoff P M . Fast and sensitive silver staining of DNA in polyacrylamide gels. Analytical Biochemistry, 1991,196(1):80-83.
doi: 10.1016/0003-2697(91)90120-I pmid: 1716076 |
| [13] |
张安世, 徐九文, 张利民 , 等. 北方部分粳稻品种遗传关系的SRAP分析. 作物杂志, 2009(6):55-58.
doi: 10.3969/j.issn.1001-7283.2009.06.014 |
| [14] |
石玲艳, 侯建华, 张永虎 , 等. 基于SRAP标记的玉米自交系遗传多样性与群体结构分析. 作物杂志, 2015(3):57-63.
doi: 10.16035/j.issn.1001-7283.2015.03.011 |
| [15] | 李锐, 白建荣, 王秀红 , 等. 144份甜玉米群体的遗传多样性分析. 作物杂志, 2018(2):17-24. |
| [16] | 张帅, 庞玉辉, 王征宏 , 等. 小麦种质资源农艺性状变异及其遗传多样性分析. 作物杂志, 2018(2):44-51. |
| [17] | Botstein D, White R L, Skolnick M , et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980,32(3):314-331. |
| [18] |
令狐斌, 侯思宇, 孙朝霞 , 等. 苦荞SRAP分子标记体系优化与遗传多样性分析. 中国农业大学学报, 2015,20(1):37-43.
doi: 10.11841/j.issn.1007-4333.2015.01.05 |
| [19] | 李维卫, 王爱兰, 聂臻臻 . S山茴香RAP反应体系正交设计优化及引物筛选. 基因组学与应用生物学, 2017,36(8):3103-3109. |
| [20] | 延娜, 李巧丽, 郭军战 . 果桑遗传差异的SRAP和EST-SSR分析. 西北农业学报, 2015,24(9):91-97. |
| [21] | 李霞, 李书华, 杨林 , 等. 芦笋种质资源的SRAP遗传多样性分析. 农学学报, 2016,6(3):40-45. |
| [22] | 林春华, 李兆龙, 乔燕春 , 等. SRAP分子标记分析体系的建立及白簕资源亲缘关系分析. 基因组学与应用生物学, 2012,31(5):498-504. |
| [23] | 张良波, 王解香, 李培旺 , 等. 梾木属SRAP-PCR反应体系的优化及引物筛选. 分子植物育种, 2014,12(5):1005-1010. |
| [24] |
杨永, 王豪杰, 张学军 , 等. 新疆甜瓜地方种质资源遗传多样性的SRAP分析. 植物遗传资源学报, 2017,18(3):436-448.
doi: 10.13430/j.cnki.jpgr.2017.03.008 |
| [25] |
黄伟, 鲁敏, 张起 , 等. 利用SRAP标记分析贵州砂梨资源遗传多样性. 西北植物学报, 2016,36(2):280-287.
doi: 10.7606/j.issn.1000-4025.2016.02.0280 |
| [26] | 朱海生, 叶新如, 陈敏氡 , 等. 丝瓜种质资源的SRAP分析. 分子植物育种, 2016,14(8):2217-2223. |
| [1] | 吴瑞香,杨建春,王利琴,郭秀娟. 应用多元分析法综合评价胡麻材料抗旱适应性[J]. 作物杂志, 2018, (5): 10–16 |
| [2] | 马孟莉,郑云,周晓梅,张婷婷,张晓倩,卢丙越. 云南哈尼梯田红米地方品种遗传多样性分析[J]. 作物杂志, 2018, (5): 21–26 |
| [3] | 田荟遥,蒋继志,李成斌,申芬,侯宁. 中国东北地区马铃薯致病疫霉遗传多样性分析[J]. 作物杂志, 2018, (3): 168–173 |
| [4] | 李锐,白建荣,王秀红,张丛卓,张效梅,闫蕾,杨瑞娟. 144份甜玉米群体的遗传多样性分析[J]. 作物杂志, 2018, (2): 17–24 |
| [5] | 张帅,庞玉辉,王征宏,王黎明,陈春燕,曾占奎,王春平. 小麦种质资源农艺性状变异及其遗传多样性分析[J]. 作物杂志, 2018, (2): 44–51 |
| [6] | 赵璐,杨治伟,部丽群,田玲,苏梅,田蕾,张银霞,杨淑琴,李培富. 宁夏和新疆水稻种质资源表型遗传多样性分析及综合评价[J]. 作物杂志, 2018, (1): 25–34 |
| [7] | 郭鹏燕,王彩萍,任杰成,赵吉平,许瑛,岳茂林. 不同地理来源绿豆农艺性状的遗传多样性研究[J]. 作物杂志, 2017, (6): 55–59 |
| [8] | 台莲梅,左豫虎,张亚玲,金永玲,郭永霞,靳学慧,金光辉. 黑龙江省马铃薯早疫病菌遗传多样性分析[J]. 作物杂志, 2017, (3): 151–156 |
| [9] | 曾潮武,梁晓东,李建疆. 中亚引进春小麦种质资源主要农艺性状的遗传多样性分析[J]. 作物杂志, 2017, (2): 67–71 |
| [10] | 郭秀娟,冯学金,杨建春,吴瑞香,王利琴. 不同种植密度和肥料配施对胡麻植株性状和经济产量的效应[J]. 作物杂志, 2017, (2): 135–138 |
| [11] | 郭秀娟,杨建春,冯学金,姜超. 不同前茬作物对胡麻干物质积累规律、品质及产量构成因子的影响[J]. 作物杂志, 2016, (2): 164–167 |
| [12] | 赵利,王利民,赵玮,党照,张建平,党占海. 胡麻RIL群体中粗脂肪和脂肪酸组分的分离及其相关性分析[J]. 作物杂志, 2016, (1): 33–37 |
| [13] | 张雪婷, 杨文雄, 王世红, 等. 甘肃省近年来育成冬小麦品种主要农艺性状的遗传多样性分析[J]. 作物杂志, 2015, (4): 27–32 |
| [14] | 石玲艳, 侯建华, 张永虎, 等. 基于sRAP标记的玉米自交系遗传多样性与群体结构分析[J]. 作物杂志, 2015, (3): 57–63 |
| [15] | 刘晓云, 孙新新, 刘昱, 等. 河北省豆科绿肥作物种质资源相关共生根瘤菌资源的研究[J]. 作物杂志, 2015, (3): 29–35 |
|
||