作物杂志,2024, 第3期: 82–89 doi: 10.16035/j.issn.1001-7283.2024.03.011
14个不同产地藜麦种质染色体核型分析
刘建霞1,2( ), 王文庆1, 薛乃雯1,2, 郭绪虎1,2, 马赛雅1, 朱国芳1, 温日宇3(
), 王文庆1, 薛乃雯1,2, 郭绪虎1,2, 马赛雅1, 朱国芳1, 温日宇3( )
)
- 1山西大同大学,037009,山西大同
2山西大同大学设施农业技术研发中心,037009,山西大同
3山西农业大学玉米研究所,034000,山西忻州
Chromosome Karyotype Analysis of 14 Quinoa Germplasms from Different Habitats
Liu Jianxia1,2( ), Wang Wenqing1, Xue Naiwen1,2, Guo Xuhu1,2, Ma Saiya1, Zhu Guofang1, Wen Riyu3(
), Wang Wenqing1, Xue Naiwen1,2, Guo Xuhu1,2, Ma Saiya1, Zhu Guofang1, Wen Riyu3( )
)
- 1Shanxi Datong University, Datong 037009, Shanxi, China
2Research and Development Center of Agricultural Facility Technology of Shanxi Datong University, Datong 037009, Shanxi, China
3Maize Research Institute, Shanxi Agricultural University, Xinzhou 034000, Shanxi, China
摘要:
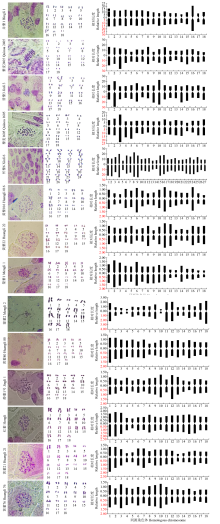
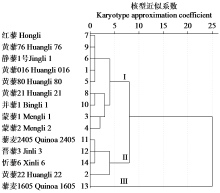
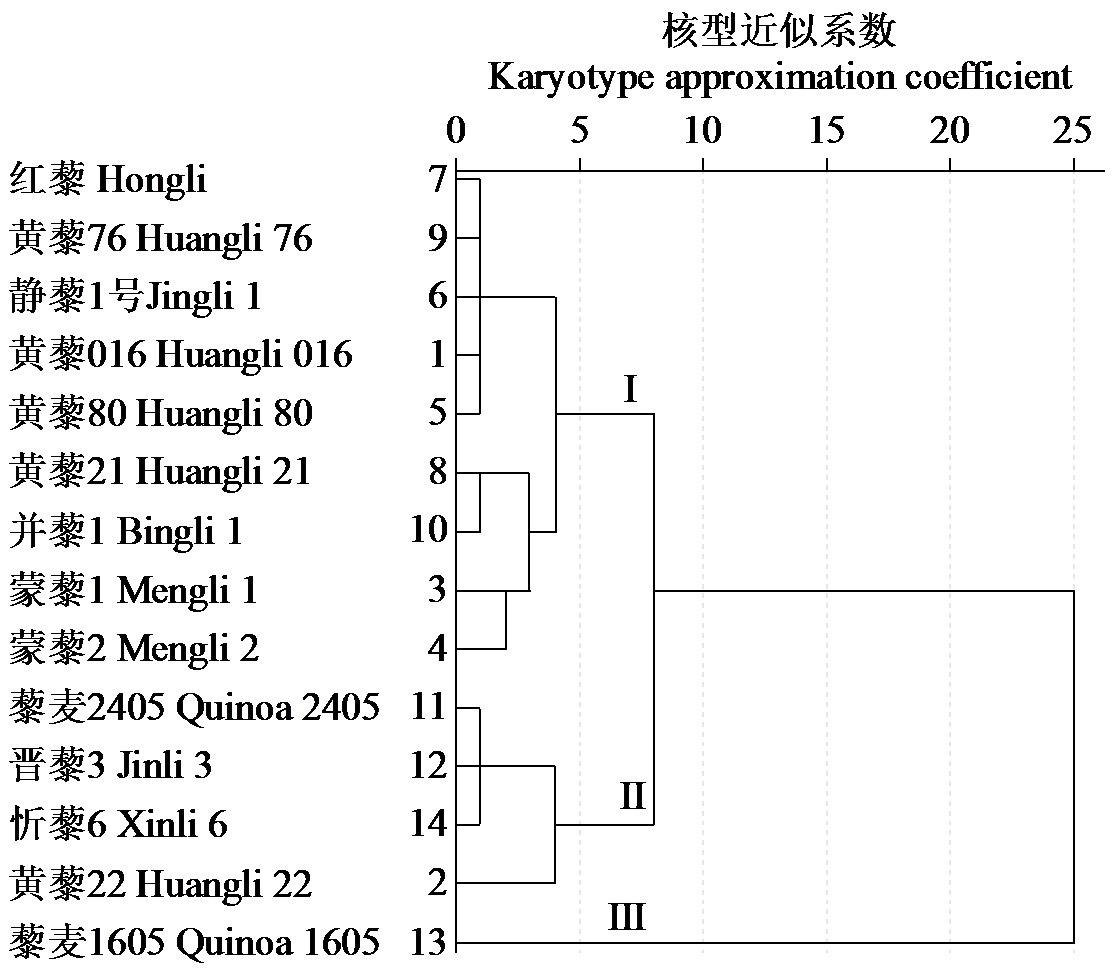
为明确不同产地藜麦的染色体数目、核型特征及亲缘与进化关系,采用常规压片法制片比较分析了14个藜麦种质染色体核型特征,对其进行近似数聚类。结果表明,忻藜6的染色体数为2n=54,其余13个种质的染色体数为2n=36;染色体核型共有1A、1B、1C和2B 4种,总体较为对称;核不对称系数范围为52.06%~59.76%。核型聚类分析结果显示,14个藜麦种质总体聚为两大类,藜麦1605单独为一类,其余13个藜麦种质为一类。来自同一地区的藜麦品种在亲缘关系和进化程度上较为相似,不同产地的藜麦品种因起源或在进化过程中受到外界环境干预程度不同,导致亲缘关系较远。该研究获得14个不同藜麦种质的核型信息,填充了有关藜麦染色体核型的数据库以及在细胞遗传学方面的研究。
| [1] | 任贵兴, 叶全宝. 藜麦生产与应用. 北京: 科学出版社, 2013:49-50. |
| [2] | 陆红法, 张永正, 方美娟, 等. 浙江庆元高山藜麦营养成分分析. 浙江师范大学学报(自然科学版), 2017, 40(4):441-445. |
| [3] | Hong S Y, Cheon K S, Yoo K O, et al. Complete chloroplast genome sequences and comparative analysis of Chenopodium quinoa and C.album. Frontiers in Plant Science, 2017, 8:1696. |
| [4] | 权有娟, 李想, 袁飞敏, 等. 基于荧光原位杂交的藜属植物核型分析. 广西植物, 2021, 41(12):1988-1995. |
| [5] | 蒙昌智, 朱剑宏, 汪义勇. 食物新资源-南美藜. 四川省营养学会2000年学术会议专题报告及论文摘要汇编, 2000. |
| [6] | 王黎明, 马宁, 李颂, 等. 藜麦的营养价值及其应用前景. 食品工业科技, 2014, 35(1):381-384,389. |
| [7] |
胡一晨, 赵钢, 秦培友, 等. 藜麦活性成分研究进展. 作物学报, 2018, 44(11):1579-1591.
doi: 10.3724/SP.J.1006.2018.01579 |
| [8] |
Brittany L G, Patricio R S, Leonel E R. Innovations in health value and functional food development of quinoa (Chenopodium quinoa Willd.). Comprehensive Reviews in Food Science and Food Safety, 2015, 14:431-445.
pmid: 27453695 |
| [9] | 王艳青, 卢文洁, 李春花, 等. 10个藜麦新品系主要农艺性状分析与综合评价. 南方农业学报, 2019, 50(3):540-545. |
| [10] | 刘文瑜, 何斌, 杨发荣, 等. 不同品种藜麦幼苗对干旱胁迫和复水的生理响应. 草业科学, 2019, 36(10):2656-2666. |
| [11] |
杨发荣, 刘文瑜, 黄杰, 等. 河西地区2个藜麦品种引种试验研究. 草地学报, 2018, 26(5):1273-1276.
doi: 10.11733/j.issn.1007-0435.2018.05.034 |
| [12] | 魏玉明, 杨发荣, 刘文瑜, 等. 陇东旱塬区复种不同藜麦品种(系)的适应性初步评价. 西北农业学报, 2020, 29(5):675- 686. |
| [13] |
陆敏佳, 蒋玉蓉, 陆国权, 等. 利用SSR标记分析藜麦品种的遗传多样性. 核农学报, 2015, 29(2):260-269.
doi: 10.11869/j.issn.100-8551.2015.02.0260 |
| [14] | 权有娟, 刘博, 袁飞敏, 等. 不同籽粒颜色藜麦品种的核型分析. 华北农学报, 2020, 35(增1):72-77. |
| [15] | 雷海英, 侯沁文, 白凤麟, 等. 八种不同产地苦参的染色体数目及核型分析. 植物生理学报, 2019, 55(7):967-974. |
| [16] | Kawatani T, Ohno T.Chromosome numbers of genus Chenopodium I. Japanese Journal of Genetics, 1950, 25:177-180. |
| [17] | Kawatani T, Ohno T. Chromosome numbers of genus Chenopodium II. Japanese Journal of Genetics, 1956, 31:15-17. |
| [18] | Palomino G, Hemandez L T, Torres E D. Nuclear genome size and chromosome analysis in Chenopodium quinoa and C. berlandieri subsp. nuttalliae. Euphytica, 2008, 164:221-230. |
| [19] | Bhargava A, Shukia S, Ohri D. Karyotypic studies on some cultivated and wild species of Chenopodium (Chenopodiaceae). Genetic Resources and Crop Evolution, 2006, 53:1309-1320. |
| [20] | Kolano B, Tomczak H, Molewska R, et al. Distribution of 5S and 35S rRNA gene sites in 34 Chenopodium species (Amaranthaceae). Botanical Journal of the Linnean Society, 2012, 170:220-231. |
| [21] | 何燕, 邓永辉, 李梦寒, 等. 藜麦品系的染色体数目及核型分析. 西南大学学报(自然科学版), 2019, 41(1):27-31. |
| [22] | 乔永刚, 宋芸. 利用EXCEL制作核型模式图. 农业网络信息, 2006(10):97-98. |
| [23] | 李懋学, 陈瑞阳. 关于植物核型分析的标准化问题. 武汉植物学研究, 1985(4):297-302. |
| [24] |
乔永刚, 王勇飞, 曹亚萍, 等. 13种蒲公英属植物核型似近系数聚类分析. 草地学报, 2020, 28(1):285-290.
doi: 10.11733/j.issn.1007-0435.2020.01.035 |
| [25] | 杨光穗, 冷青云, 王呈丹, 等. 16个红掌品种的核型分析. 热带作物学报, 2016, 37(12):2283-2287. |
| [26] |
Ricroch A, Yockteng R, Brown S C, et al. Evolution of genome size across some cultivated Allium species. Genome, 2005, 48 (3):511-520.
pmid: 16121247 |
| [27] | Bhargava A, Rana T S, Shukla S, et al. Seed protein electrophoresis of some cultivated and wild species of Chenopodium. Biologia Plantarum, 2005, 49(4),505-511. |
| [28] | 夏雪, 田玉肖, 王祉琪, 等. 基于核型分析对芥蓝分类地位的探究. 分子植物育种, 2019, 17(7):2291-2296. |
| [29] | 闫素丽, 安玉麟, 孙瑞芬. 内葵杂3号染色体核型分析. 植物遗传资源学报, 2012, 11(6):784-788. |
| [30] |
卫尊征, 殷选红, 熊敏, 等. 3个彩色马蹄莲引进品种的核型分析. 植物遗传资源学报, 2012, 13(4):650-654.
doi: 10.13430/j.cnki.jpgr.2012.04.025 |
| [31] |
Wilson H, Manhart J. Crop/weed gene flow Chenopodium quinoa Willd. and C.berlandieri Moq. Theoretical and Applied Genetics, 1993, 86(5):642-648.
doi: 10.1007/BF00838721 pmid: 24193715 |
| [32] | Barker R E, Kilgore J A, Cook R L, et al. Use of flow cytometry to determine ploidy level of ryegrass. Seed Science and Technology, 2001, 29:493-502. |
| [33] | Kamemoto H, Shindo K, Kosoki K, et al. Chromosome homology in the Ceratobium, Phalaenanthe, and Latourea sections of the genus dendrobium. Pacific Science, 1964, 18:104-115. |
| [34] | Yan J, Zhang J, Sun K, et al. Ploidy level and DNA content of erianthus arundinaceus as determined by flow cytometry and the association with biological characteristics. PLoS ONE, 2016, 11(3):e0151948 |
| [35] |
任丽娟, 赵连生, 陈雅坤, 等. 基于主成分分析和聚类分析方法综合评价东北地区不同品种全株玉米青贮饲料的青贮品质. 动物营养学报, 2020, 32(8):3856-3868.
doi: 10.3969/j.issn.1006-267x.2020.08.044 |
| [1] | 合佳敏, 张永清, 张萌, 梁萍, 王丹, 严翻翻. 烯效唑浸种对盐碱胁迫下藜麦农艺性状及生理特性的影响[J]. 作物杂志, 2024, (2): 234–241 |
| [2] | 杨恩泽, 王树彦, 刘瑞香, 石丰源, 张锦豪, 李佳娜, 李志伟, 郭占斌. 基于SRAP的藜麦种质资源遗传多样性分析[J]. 作物杂志, 2023, (6): 79–85 |
| [3] | 李兴河, 王海涛, 刘存敬, 唐丽媛, 张素君, 蔡肖, 张香云, 张建宏. 利用海岛棉染色体片段导入系定位纤维品质性状QTL[J]. 作物杂志, 2023, (5): 1–9 |
| [4] | 郭红霞, 王创云, 邓妍, 赵丽, 张丽光, 郭虹霞, 秦丽霞, 高飞, 席瑞珍. 藜麦对低氮胁迫的响应研究[J]. 作物杂志, 2023, (3): 221–229 |
| [5] | 陈翠萍, 闫殿海, 张书苗, 左皓南, 高森, 刘洋. 藜麦SSR指纹图谱构建及遗传多样性分析[J]. 作物杂志, 2023, (3): 35–42 |
| [6] | 梁萍, 张永清, 张萌, 薛小娇, 李平平, 张文燕, 王丹, 赵刚. PAM施用深度对盐碱胁迫下藜麦生长及生理指标的影响[J]. 作物杂志, 2023, (2): 178–185 |
| [7] | 梅丽. 北京藜麦适应性栽培研究进展及展望[J]. 作物杂志, 2022, (6): 14–22 |
| [8] | 王思宇, 左文博, 朱凯莉, 郭慧敏, 邢宝, 郭雨晴, 包玉英, 杨修仕, 任贵兴. 71份藜麦品种资源的农艺性状及营养品质分析与评价[J]. 作物杂志, 2022, (3): 63–72 |
| [9] | 余镁霞, 邓浩东, 谭景艾, 宋贵廷, 吴光亮, 陈利平, 刘睿琦, 邹安东, 贺浩华, 边建民. 利用染色体片段置换系定位低温影响水稻萌芽期根长和芽长QTL[J]. 作物杂志, 2021, (6): 36–45 |
| [10] | 亓晓蕾, 李兴锋, 吕广德, 王瑞霞, 王君, 孙宪印, 孙盈盈, 陈永军, 钱兆国, 吴科. 基于SNP分子标记的泰山/泰科麦系列小麦遗传解析[J]. 作物杂志, 2021, (5): 64–71 |
| [11] | 李娥贤, 殷富有, 张敦宇, 陈越, 余腾琼, 雷涌涛, 肖素勤, 程在全, 柯学. 云南药用野生稻高质量染色体DNA的制备[J]. 作物杂志, 2020, (5): 103–109 |
| [12] | 王中秋, 应鹏飞, 陈梦涛, 贺琼颖, 胡鑫. 普通小麦-野生二粒小麦染色体臂置换系籽粒与品质性状分析[J]. 作物杂志, 2020, (4): 37–44 |
| [13] | 王艳青,李勇军,李春花,卢文洁,孙道旺,尹桂芳,洪波,王莉花. 藜麦主要农艺性状与单株产量的相关和通径分析[J]. 作物杂志, 2019, (6): 156–161 |
| [14] | 丁海燕,汪春林,武燕,任国领,翟祖欢,马军. 龙葵杂10号向日葵染色体核型分析[J]. 作物杂志, 2019, (2): 99–102 |
| [15] | 崔宏亮,邢宝,姚庆,张琴萍,杨修仕,么杨,任贵兴,秦培友. 新疆伊犁河谷藜麦产业发展的SWOT分析[J]. 作物杂志, 2019, (1): 32–37 |
|
||