我国糯玉米种质资源丰富,但却没有系统地进行亲缘关系划分,从而导致资源利用的盲目性。随着生物学特别是遗传学和分子生物学的发展,检测遗传多样性的方法不断改进和完善。从形态学水平、细胞学(染色体)水平、生理生化水平逐步发展到分子水平。然而,无论在何种水平上进行研究,其目的都是揭示遗传物质的变异[6-7]。近年来,基因芯片和测序技术受到科研学者的青睐,SNP标记具有标记多态性好、物理位置明确和通量高等特点,是继SSR标记之后应用最广的第三代基因标记技术[8⇓⇓-11]。目前,SNP芯片技术大多应用在普通玉米杂种优势群的划分。吴金凤等[12]利用1041个SNP位点对51份玉米自交系进行类群划分,最终将其划分为7个杂种优势群。卢柏山[13]通过SNP标记将39份甜玉米自交系划分为5个类群。范競升等[14]通过10K玉米基因芯片技术,将广西207份糯玉米农家种和6份糯玉米自交系划分为4个类群。
目前利用SNP标记对糯玉米进行研究还较少,关于黑龙江地区尚未见报道。本研究采用Maize 6H-60K芯片对黑龙江省内常用糯玉米自交系进行遗传多样性分析,为黑龙江地区糯玉米种质创新和新品种选育提供理论基础。
1 材料与方法
1.1 试验材料
试验材料为黑龙江省农业科学院玉米研究所提供的黑龙江省常用的96份糯玉米自交系,包括外引系、改良系和二环系,编号FZ001~FZ100,其中编号FZ067、FZ073、FZ087、FZ092因样品种子没发芽数据缺失。
1.2 DNA提取
在大田幼苗3叶1心时取嫩叶,采用磁珠法提取样品DNA,通过琼脂糖电泳检测DNA的纯度,用紫外分光光度计检测质量状况,用酶标仪双链定量法检测DNA浓度。选择DNA条带单一、明亮、完整无降解且无拖尾现象的DNA进行琼脂糖电泳检测,用紫外分光光度计检测OD260/280值为1.8~2.0,OD260/230值>2.0,DNA浓度>20 ng/μL。
1.3 基因型鉴定
采用北京市农林科学院玉米研究中心自主研制的Maize 6H-60K芯片[15]进行基因分型分析。
1.4 数据处理
利用TASSEL软件完成基因型数据描述性分析和玉米种质资源间遗传距离、聚类分析,利用R语言中LEA、GGplot2、ScatterPlot 3d等数据包完成群体遗传分析。用PowerMarker V3.25软件计算多态性信息含量(PIC)、平均等位变异和基因多样性等。
2 结果与分析
2.1 SNP标记筛选过程
根据芯片数据结果中Assay QC文件,筛选到高分辨率SNP 39 381个,分布在玉米10条染色体上,10号染色体上SNP数量最少,为2660个,1号染色体上SNP数量最多,为6237个。
2.2 基因型数据的描述性分析
将缺失数据超过20%、杂合性超过20%、最小等位基因频率小于0.05的SNP标记过滤掉,最终获得14 312个SNP用于进一步分析。
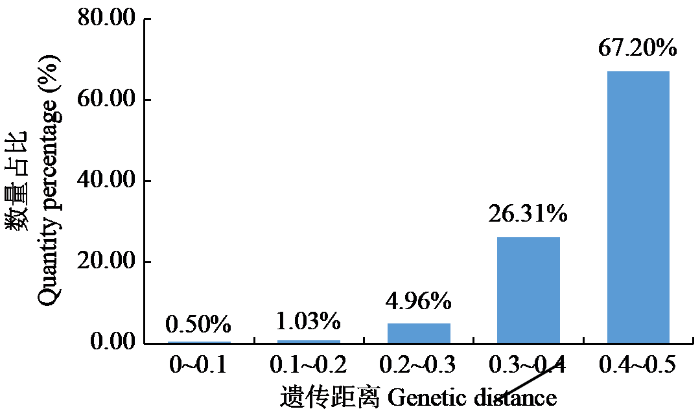
统计(图1)分析发现,糯玉米自交系之间的遗传距离在0到0.50之间变化,总体平均距离为0.38。大多数(67.2%)遗传距离在0.4~0.5。最小的遗传距离(0.009)出现在近交系FZ001和FZ007之间,这2个近交系均为白糯材料,亲缘关系较近。最大的遗传距离(0.499)出现在自交系FZ001和FZ090之间,FZ001为白糯材料,FZ090为黑糯材料,亲缘关系较远。
图1
图2
表1 基因型总结
Table 1
| 项目Item | 数值Numeric |
|---|---|
| 样品数量Sample number | 96 |
| 标记数量Number of markers | 38 693 |
| 杂合比例Heterozygous ratio | 0.034 |
| 最小等位基因频率Minimum allele frequency | 0.380 |
| 基因多样性Gene diversity | 0.387 |
| PIC | 0.306 |
2.3 供试玉米资源间主成分分析和聚类分析
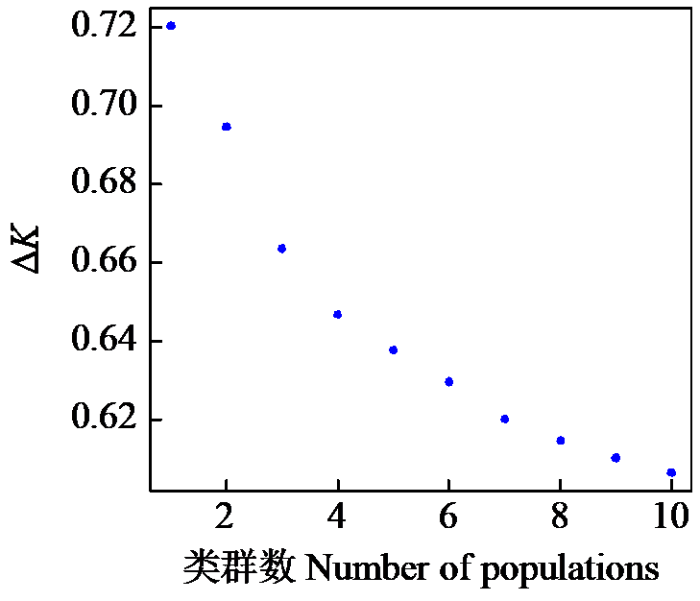
利用R软件对96份糯玉米自交系进行群体遗传系统性解析,结果如图3显示,当类群数(K)为1~10时,ΔK=3时下降趋势开始减缓,并结合育种实际情况,本研究选择ΔK=3进一步分析。当ΔK=3时表明96个供试自交系可分为3个类群。
图3
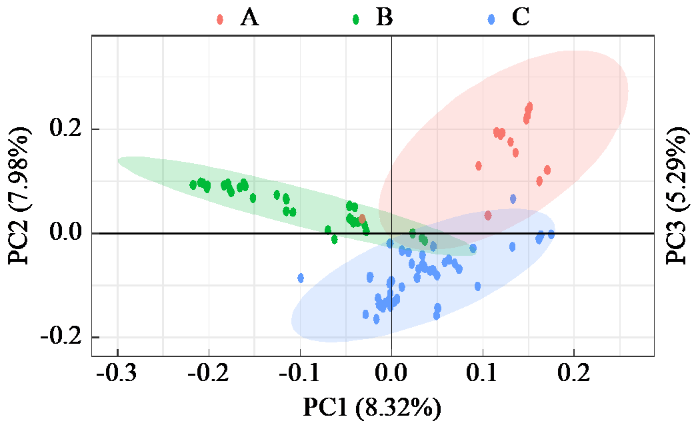
对96份自交系进行了PCA分析,比例最高的3个主成分分别占比为8.32%、7.98%和5.29%,从PCA分析结果(图4)发现3个群基本被划分开,根据种质资源在育种中的应用,3个群之间可以形成强杂种优势。
图4
图4
96份糯玉米自交系主成分分析
Fig.4
Principal component analysis of 96 waxy maize inbred lines
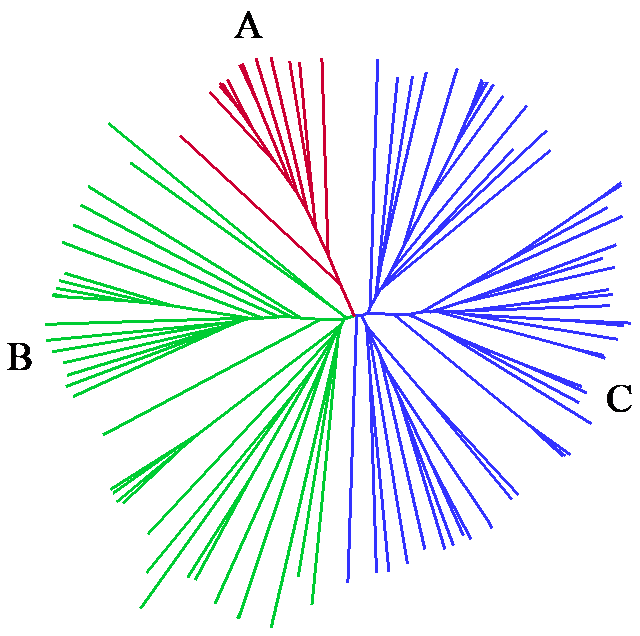
进一步计算供试资源间的遗传距离,采用N-J方法进行聚类分析。结果如图5所示,整体上供试的96份玉米资源可以被分成3个类群,其中红色为A类群,包含了FZ-099、FZ017、FZ031、FZ045、FZ052、FZ055、FZ049、FZ047、FZ053、FZ050、FZ059、FZ048、FZ058、FZ038共14份自交系;绿色代表B类群,包含了FZ-002、FZ090、FZ098、FZ060、FZ061、FZ095、FZ065、FZ068、FZ094、FZ097、FZ096、FZ062、FZ072、FZ066、FZ071、FZ009、FZ063、FZ083、FZ093、FZ076、FZ069、FZ084、FZ064、FZ070、FZ074、FZ077、FZ078、FZ082、FZ079、FZ091、FZ081、FZ085、FZ088共33份自交系的糯玉米群;蓝色为C类群,包含了FZ-001、FZ004、FZ030、FZ042、FZ005、FZ044、FZ053、FZ056、FZ100、FZ046、FZ051、FZ007、FZ075、FZ057、FZ003、FZ010、FZ043、FZ080、FZ011、FZ021、FZ022、FZ012、FZ026、FZ020、FZ027、FZ033、FZ035、FZ023、FZ041、FZ014、FZ039、FZ040、FZ086、FZ025、FZ028、FZ037、FZ006、FZ015、FZ08、FZ034、FZ013、FZ018、FZ024、FZ089、FZ036、FZ029、FZ016、FZ032、FZ019共49份自交系的糯玉米群。A类群中包含了垦粘1母本(FZ045)以及亲缘关系近的13份种质资源,B类群包含了先糯父本(FZ060)以及亲缘关系近的32份种质资源,其中先糯父本为籽粒用糯玉米种质资源,C类群包含了垦粘1父本(FZ044)以及亲缘关系相近的48份种质资源,在实际育种应用中,选育鲜食糯玉米优先考虑A和C类群间的杂种优势,选育生产中籽粒型糯玉米则考虑A和B以及C和B类群间的杂种优势。
图5
2.4 群体遗传结构分析
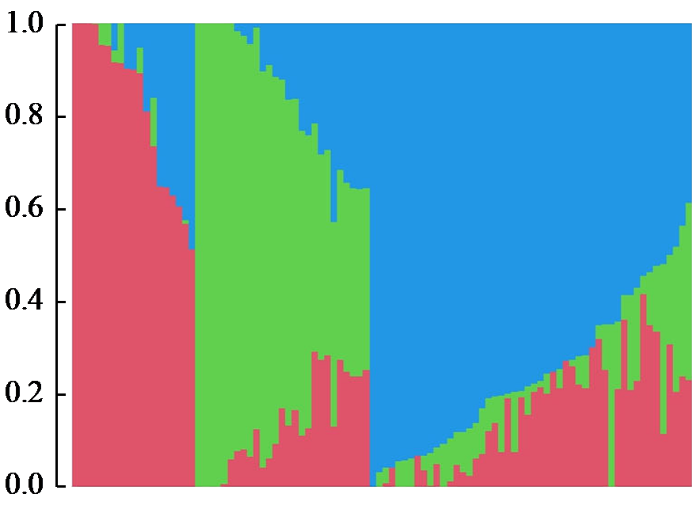
群体遗传结构是指遗传变异在物种或群体中的一种非随机分布。可按照地理分布或其他标准将一个类群分为若干亚群,处于同一亚群内的不同个体亲缘关系较高,而亚群与亚群间的亲缘关系较远。通过R软件对96份糯玉米自交系进行群体结构分析,结果如图6所示,以红色(A)为主有14份自交系,亲缘关系与垦粘1母本相近;以绿色(B)为主有33份自交系,亲缘关系与先糯父本相近;以蓝色(C)为主有49份自交系,亲缘关系与垦粘1父本相近。群体结构结果与主成分分析和聚类分析结果相同,证明96份糯玉米种质划分为3个类群较为合理。
图6
图6
96份糯玉米自交系群体遗传结构分析
Fig.6
Group genetic structure analysis of 96 waxy maize inbred lines
3 讨论
近年来,随着社会的发展和人们生活水平的不断提高,糯玉米也越来越受人们的喜爱,市场需求逐年增加。糯玉米不仅可以鲜食,而且在畜牧业和加工行业也有广泛应用,因此糯玉米具有广阔的市场前景。我国是世界第二大糯玉米生产国,也是第一大进口国。通过选育优良的糯玉米杂交种是改变这一现状的途径之一。尽管我国糯玉米资源丰富,但育种水平相对落后[16]。
由于糯玉米的种质材料大多数来源不详,也导致亲缘关系不清楚,因此系谱追踪法并不能用于其遗传多样性研究,而配合力测定和同工酶标记等方法都具有一定的局限性。
基因芯片技术具有高通量、高分辨率和分型准确等优点,随着基因芯片技术成本的降低也逐渐应用于植物育种中。吴金凤[23]利用SNP标记技术将72份玉米自交系划分为7个杂种优势群,其结果与SSR分群结果相同。高嵩等[24]通过SNP标记技术对205份玉米材料进行遗传结构等分析,将材料划分为7个类群。基因芯片在糯玉米中的应用鲜有报道,本研究利用SNP芯片技术对糯玉米种质进行遗传多样性分析,结果发现,材料FZ001(白糯)与FZ090(黑糯)的遗传距离最大,PIC平均值为0.306,这一数值与前人[25⇓-27]在玉米研究中的PIC结果相似,说明SNP芯片技术在糯玉米种质遗传分析中的可行性。本研究进一步利用PCA分析、聚类分析、群体结构分析,其结果一致,均将96份糯玉米自交系划分为3个类群。与普通玉米相比,糯玉米没有明确的类群划分,本研究对东北常用糯玉米自交系进行类群划分,能够避免组配的盲目性,减少育种工作量,进而缩短育种年限,为糯玉米杂交种的选育提供了理论基础。
4 结论
本研究利用14 312个SNP位点对96份玉米自交系进行基因型分析,最终发现有8033个SNP标记基因多样性处于0.40~0.50,占总数的56.1%,说明具有较高的多态性水平,结果显示最小等位基因频率范围为0~0.5,平均值为0.380;PIC范围为0.094~0.375,平均值为0.306;群体结构分析将96份糯玉米自交系划分为3个类群,A群为垦粘1母本血缘群,共有14份糯玉米自交系;B群为先糯父本血缘群,共计33份,C群为垦粘1父本血缘群,共计49份。
参考文献
Genetic distance based on simple sequence repeats and heterosis in tropical maize populations
Genetic diversity assessment of extra-early maturing yellow maize inbreds and hybrid performance in Striga-infested and Striga-free environments
Association mapping for evaluation of population structure, genetic diversity, and physiochemical traits in drought-stressed maize germplasm using SSR markers
Diversity analysis in Cannabis sativa based on large-scale development of expressed sequence tag-derived simple sequence repeat markers
Single nucleotide polymorphism (SNP) discovery and association study of flowering times, crude fat and fatty acid composition in rapeseed (Brassica napus L.) mutant lines using genotyping-by-sequencing (GBS)
New resources for genetic studies in maize (Zea mays L.): a genome-wide Maize6H-60K single nucleotide polymorphism array and its application
Efficiency of RAPD, ISSR, iPBS, SCoT and phytochemical markers in the genetic relationship study of five native and economical important bamboos of North-East India
Sequencing of the chloroplast genomes of cytoplasmic male-sterile and male-fertile lines of soybean and identification of polymorphic markers
BAC-end sequence-based SNPs and Bin mapping for rapid integration of physical and genetic maps in apple
DOI:10.1016/j.ygeno.2008.11.005
PMID:19059473
[本文引用: 1]

A genome-wide BAC physical map of the apple, Malus x domestica Borkh., has been recently developed. Here, we report on integrating the physical and genetic maps of the apple using a SNP-based approach in conjunction with bin mapping. Briefly, BAC clones located at ends of BAC contigs were selected, and sequenced at both ends. The BAC end sequences (BESs) were used to identify candidate SNPs. Subsequently, these candidate SNPs were genetically mapped using a bin mapping strategy for the purpose of mapping the physical onto the genetic map. Using this approach, 52 (23%) out of 228 BESs tested were successfully exploited to develop SNPs. These SNPs anchored 51 contigs, spanning approximately 37 Mb in cumulative physical length, onto 14 linkage groups. The reliability of the integration of the physical and genetic maps using this SNP-based strategy is described, and the results confirm the feasibility of this approach to construct an integrated physical and genetic maps for apple.
Importance of genetic diversity assessment in crop plants and its recent advances: an overview of its analytical perspectives
基于SNP芯片揭示中国玉米育种种质的遗传多样性与群体遗传结构
DOI:10.3864/j.issn.0578-1752.2018.04.003
[本文引用: 1]

【目的】选择具有重要育种价值的玉米自交系进行遗传多样性与群体遗传结构解析,为玉米育种实践提供指导和参考。【方法】选用344份具有广泛代表性和时效性的玉米自交系,其中包括美国主要杂种优势群、由国内地方种质发展来的杂种优势群、由美国商业化杂交种选系发展来的杂种优势群以及近年来在中国玉米育种中应用的新种质。利用北京市农林科学院玉米研究中心自主研发的包含3 072个SNP位点的MaizeSNP3072芯片对供试自交系进行全基因组扫描,揭示其遗传多样性与群体遗传结构。【结果】在344份自交系中,3 072个SNP标记所检测到的基因多样性为0.028—0.646,平均为0.442;多态信息含量(PIC)为0.028—0.570,平均PIC值为0.344。群体遗传结构分析表明,K=8时,△K值最大,即本研究所采用的自交系群体可以划分为8个类群,分别为旅大红骨群、黄改群(又称塘四平头群)、Iodent群、兰卡斯特群、P群、改良瑞德群、瑞德群和X群,其中前7个群已有报道且基本被育种家所公认,第8个群为近年来以X1132X等杂交种作为基础材料选育出的优新种质,命名为X群。比较8个类群,遗传分化系数(Fst)为0.319—0.512,遗传距离为0.229—0.514。AMOVA结果表明类群间存在显著的遗传变异,占总遗传变异的38.6%,类群内的遗传变异占58.1%。PCA(主成分分析)结果显示,X群与黄改群、兰卡斯特群遗传关系较远,与Iodent群遗传关系较近。各类群平均基因多样性分析结果表明,随着类群改良年代的增加,类群平均基因多样性降低,其中X群种质平均基因多样性最高;进一步分析表明,美国种质类群(兰卡斯特群、瑞德群和Iodent群)和国内地方种质改良系(旅大红骨群和黄改群)核心材料多样性下降幅度较大,P群和改良瑞德群核心材料下降幅度较小,X群核心材料则没有下降趋势,说明X群核心材料仍然保留了较高的遗传多样性,未来还有很大的育种潜力可挖掘。【结论】近年来,以X1132X等杂交种所构建的基础材料选育而成的京724等系列优良自交系,区别于其他已知的7大类群,可以单独成群,称之为X群。该群与黄改群之间存在较远的遗传距离,从分子水平验证了“X群×黄改群”这种强杂优模式具有良好的应用潜力。