作物杂志,2023, 第1期: 30–37 doi: 10.16035/j.issn.1001-7283.2023.01.005
基于叶绿体SSR分子标记的苦参种质资源遗传多样性分析
宋芸1,2( ), 张鑫瑞1, 贺嘉欣1, 李政1, 孙哲1, 李澳旋1, 乔永刚1,2(
), 张鑫瑞1, 贺嘉欣1, 李政1, 孙哲1, 李澳旋1, 乔永刚1,2( )
)
- 1山西农业大学生命科学学院,030801,山西晋中
2中兽医药现代化山西省重点实验室,030801,山西晋中
Genetic Diversity Analysis of Sophora flavescens Ait. Germplasm Resources Based on cpSSR Markers
Song Yun1,2( ), Zhang Xinrui1, He Jiaxin1, Li Zheng1, Sun Zhe1, Li Aoxuan1, Qiao Yonggang1,2(
), Zhang Xinrui1, He Jiaxin1, Li Zheng1, Sun Zhe1, Li Aoxuan1, Qiao Yonggang1,2( )
)
- 1College of Life Sciences, Shanxi Agricultural University, Jinzhong 030801, Shanxi, China
2Shanxi Key Laboratory for Modernization of TCVM, Jinzhong 030801, Shanxi, China
摘要:
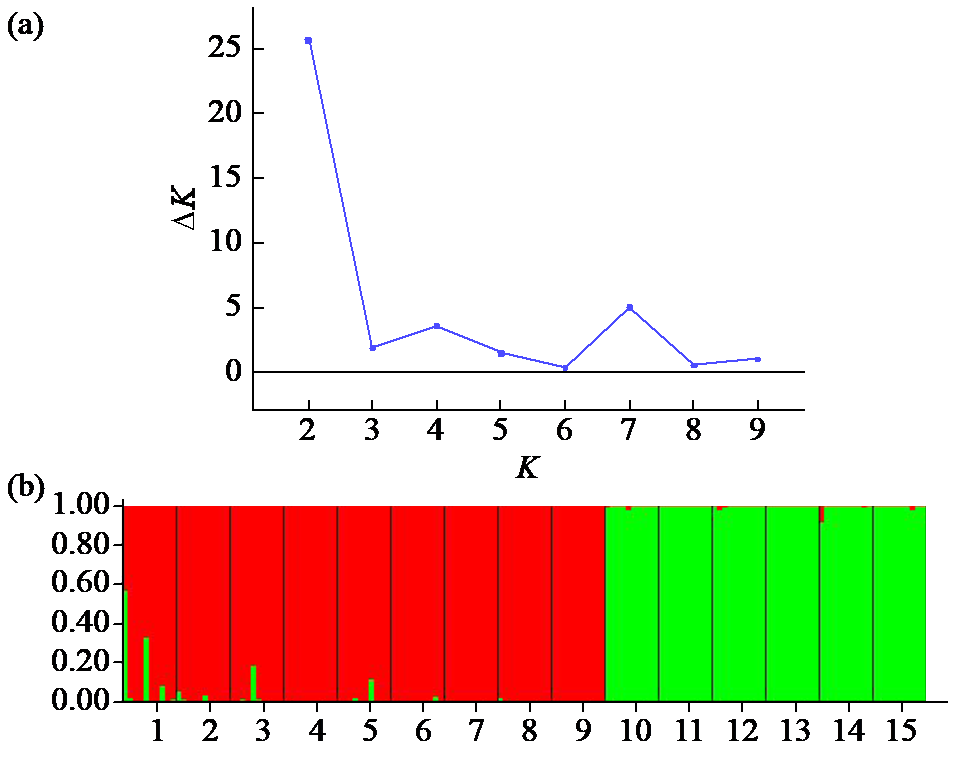
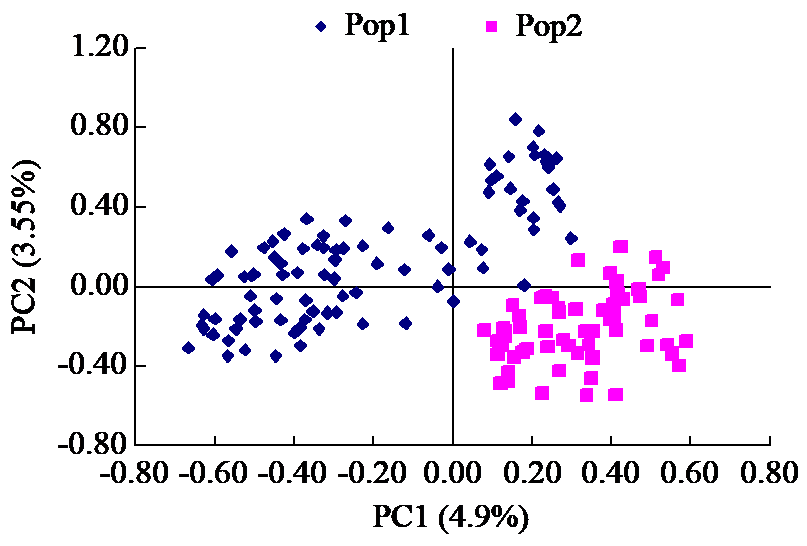
以来自15个不同产地的150份苦参为材料,采用cpSSR分子标记技术探究不同产地苦参种质资源的遗传多样性。结果表明,18对cpSSR引物共扩增出311个条带,检测出97.47个等位基因,平均每对cpSSR引物扩增出17.28个条带。平均检测到的等位基因数(Na)为5.415,有效等位基因数(Ne)为4.395,Shannon's信息指数(I)为1.535,多样性指数(h)为0.748,无偏多样性指数(uh)为0.832,多态信息含量(PIC)为0.886,PIC>0.5说明18对cpSSR引物具有较高的多态性。不同产地的苦参遗传多样性丰富,其中山西武乡土河坪种质(THP)的I为1.600,Ne为4.786,其他遗传多样性指数均较大,是遗传多样性较丰富的苦参产地。居群分子方差分析(AMOVA)表明,苦参种质内个体间差异大,苦参种质内遗传变异率大于种质间的。聚类分析、主坐标分析(PCoA)和STRUCTURE软件进行的遗传结构分析结果均将不同产地的苦参分为2个类群,并且分类结果有明显的地理相关性。第1类群中的9个苦参种质主要来自山西、河北、陕西和内蒙古等地,第2类群主要来自山东、河南、江苏以及安徽等地。18对cpSSR引物在苦参内具有很好的适用性。苦参种质资源的遗传多样性研究为发掘、利用与保护苦参种质资源以及优良品种的选育提供一定的理论基础。
| [1] | 中国科学院《中国植物志》编辑委员会. 中国植物志. 北京: 科学出版社, 1994. |
| [2] | 国家药典委员会. 中国人民共和国药典(一部). 北京: 中国医药科技出版社, 2020:211. |
| [3] | 张晓雯, 李凌宇, 尚海, 等. 苦参碱及其类似物的结构修饰研究进展. 中草药, 2019, 50(23):5892-5900. |
| [4] | 葛淑俊, 孟义江, 李广敏, 等. 我国药用植物遗传多样性研究进展. 中草药, 2006(10):1584-1589. |
| [5] |
Li Q, Su X, Ma H, et al. Development of genic SSR marker resources from RNA-seq data in Camellia japonica and their application in the genus Camellia. Scientific Reports, 2021, 11(1):9919.
doi: 10.1038/s41598-021-89350-w |
| [6] |
Grover A, Sharma P C. Development and use of molecular markers:past and present. Critical Reviews in Biotechnology, 2016, 36(2):290-302.
doi: 10.3109/07388551.2014.959891 pmid: 25430893 |
| [7] | 张征锋, 肖本泽. 基于生物信息学与生物技术开发植物分子标记的研究进展. 分子植物育种, 2009, 7(1):130-136. |
| [8] | 张全芳, 姜明松, 陈峰, 等. 山东省水稻品种(系)的遗传多样性分析. 作物杂志, 2021(4):26-31. |
| [9] | 李琼, 常世豪, 武婷婷, 等. 120份大豆种质资源遗传多样性和亲缘关系分析. 作物杂志, 2021(4):51-58. |
| [10] |
Tonti-Filippini J, Nevill P G, Dixon K, et al. What can we do with 1000 plastid genomes?. The Plant Journal, 2017, 90(4):808-818.
doi: 10.1111/tpj.13491 pmid: 28112435 |
| [11] |
Zhou J, Chen X, Cui Y, et al. Molecular structure and phylogenetic analyses of complete chloroplast genomes of two aristolochia medicinal species. International Journal of Molecular Sciences, 2017, 18(9):1839.
doi: 10.3390/ijms18091839 |
| [12] |
Yu X Q, Drew B T, Yang J B, et al. Comparative chloroplast genomes of eleven Schima (Theaceae) species:insights into DNA barcoding and phylogeny. PLoS ONE, 2017, 12:e0178026.
doi: 10.1371/journal.pone.0178026 |
| [13] | Xu C, Dong W P, Li W Q, et al. Comparative analysis of six Lagerstroemia complete chloroplast genomes. Frontiers in Plant Science, 2017, 8:15. |
| [14] |
Gu C H, Tembrock L R, Zheng S Y, et al. The complete chloroplast genome of Catha edulis:a comparative analysis of genome features with related species. International Journal of Molecular Sciences, 2018, 19(2):525.
doi: 10.3390/ijms19020525 |
| [15] | 杜久军, 左力辉, 刘易超, 等. 裂叶榆叶绿体基因组及CP-SSR位点分析. 植物遗传资源学报, 2018, 19(6):1187-1196. |
| [16] | 薛宏, 易自力, 肖亮, 等. 芒属植物叶绿体InDel标记的开发与应用. 现代农业科技, 2015(7):153-155,159. |
| [17] | Zhang S A, Gao M Q, Zaitlin D. Molecular linkage mapping and marker-trait associations with NIRPT,a downy mildew resistance gene in Nicotiana langsdorffii. Frontiers in Plant Science, 2012, 3:185. |
| [18] |
Breidenbach N, Gailing O, Krutovsky K V. Genetic structure of coast redwood (Sequoia sempervirens [D. Don] Endl.) populations in and outside of the natural distribution range based on nuclear and chloroplast microsatellite markers. PLoS ONE, 2020, 15 (12):e0243556.
doi: 10.1371/journal.pone.0243556 |
| [19] |
Gichira A W, Avoga S, Li Z Z, et al. Comparative genomics of 11 complete chloroplast genomes of Senecioneae (Asteraceae) species:DNA barcodes and phylogenetics. Botanical Studies, 2019, 60(1):17.
doi: 10.1186/s40529-019-0265-y |
| [20] | 高源, 王大江, 王昆, 等. 新疆野苹果叶绿体DNA变异与遗传进化分析. 植物遗传资源学报, 2020, 21(3):579-587. |
| [21] |
Aecyo P, Marques A, Huettel B, et al. Plastome evolution in the Caesalpinia group (Leguminosae) and its application in phylogenomics and populations genetics. Planta, 2021, 254(2):27.
doi: 10.1007/s00425-021-03655-8 pmid: 34236509 |
| [22] | 张靖国, 曹玉芬, 陈启亮, 等. 基于叶绿体DNA变异的湖北梨属种质系统进化及遗传多样性分析. 植物遗传资源学报, 2016, 17(4):766-772. |
| [23] | 王荣升, 杨庆文. 基于叶绿体基因多样性的中国水稻起源进化研究. 植物遗传资源学报, 2011, 12(5):686-693. |
| [24] |
Shahzadi I, Ahmed R, Hassan A, et al. Optimization of DNA extraction from seeds and fresh leaf tissues of wild marigold (Tagetes minuta) for polymerase chain reaction analysis. Genetics and Molecular Research, 2010, 9(1):386-393.
doi: 10.4238/vol9-1gmr747 pmid: 20309824 |
| [25] | 乔永刚, 贺嘉欣, 王勇飞, 等. 药用植物苦参的叶绿体基因组及其特征分析. 药学学报, 2019, 54(11):2106-2112. |
| [26] |
Beier S, Thiel T, Münch T, et al. MISA-web:a web server for microsatellite prediction. Bioinformatics, 2017, 33(16):2583-2585.
doi: 10.1093/bioinformatics/btx198 |
| [27] |
Peakall R, Smouse P E. GenAlEx 6.5:genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, 2012, 28(19):2537-2539.
pmid: 22820204 |
| [28] |
Liu K J, Muse S V. Power Marker:an integrated analysis environment for genetic marker analysis. Bioinformatics, 2005, 21(9):2128-2129.
doi: 10.1093/bioinformatics/bti282 |
| [29] |
Kumar S, Stecher G, Li M, et al. MEGA X:molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 2018, 35(6):1547-1549.
doi: 10.1093/molbev/msy096 |
| [30] |
Saitou N, Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution, 1987, 4(4):406-425.
doi: 10.1093/oxfordjournals.molbev.a040454 pmid: 3447015 |
| [31] |
Evanno G, Regnaut S, Goudet J. Detexting the number of clusters of individuals using the software STRUCTURE:a simulation study. Molecular Ecology, 2005, 14(8):2611-2620.
doi: 10.1111/j.1365-294X.2005.02553.x pmid: 15969739 |
| [32] |
Earl D A, Vonholdt B M. STRUCTURE HARVESTER:a website and program for visualizing STRUCTURE output and implementing the Evanno method. Conservation Genetics Resources, 2012, 4(2):359-361.
doi: 10.1007/s12686-011-9548-7 |
| [33] | 王崇, 王连军, 杨新笋, 等. 104个甘薯品种的cpSSR指纹图谱构建及遗传多样性分析. 热带作物学报, 2021, 42(6):1549-1556. |
| [34] | 魏潇, 章秋平, 刘威生, 等. 基于叶绿体SSR单倍型的普通杏演化关系. 植物遗传资源学报, 2018, 19(4):705-712. |
| [35] | Yan Y D, Li X Y, Worth J R P, et al. Development of chloroplast microsatellite markers for Glyptostrobus pensilis (Cupressaceae). Applications in Plant Sciences, 2019, 7(7):e11277. |
| [36] | 李祥栋, 石明, 陆秀娟, 等. 利用叶绿体基因组SSR标记揭示薏苡属种质资源的遗传多样性. 华北农学报, 2019, 34(增1):6-14. |
| [37] | 段永红, 渠云芳, 王长彪, 等. 药用植物苦参SSR-PCR体系的优化与验证. 中国农业大学学报, 2014, 19(5):95-100. |
| [38] |
刘亚令, 耿雅萍, 解潇冬, 等. 基于SSR分子标记的药用黄芪遗传多样性与遗传结构分析. 草地学报, 2019, 27(5):1154-1162.
doi: 10.11733/j.issn.1007-0435.2019.05.006 |
| [39] |
程丽莉, 胡广隆, 苏淑钗, 等. 板栗及其近缘种叶绿体SSR遗传多样性分析. 华北农学报, 2015, 30(2):145-149.
doi: 10.7668/hbnxb.2015.02.026 |
| [1] | 黄贵斌, 关耀兵, 牛永岐, 周丽蕾, 赵永峰. 103份鹰嘴豆种质资源12个主要农艺性状综合鉴定评价[J]. 作物杂志, 2023, (1): 6–13 |
| [2] | 郭欢乐, 汤彬, 李涵, 曹钟洋, 曾强, 刘良武, 陈志辉. 湖南省玉米地方品种表型性状综合评价及类群划分[J]. 作物杂志, 2022, (6): 33–41 |
| [3] | 李王胜, 王雪倩, 尹希龙, 石杨, 刘大丽, 谭文勃, 兴旺. 甜菜种质资源苗期耐旱性综合评价[J]. 作物杂志, 2022, (6): 54–60 |
| [4] | 赵小琴, 贾瑞玲, 刘军秀, 刘彦明, 文殷花, 师丽丽, 张娟宁, 马宁. 120份谷子种质资源的农艺性状表现和遗传多样性分析[J]. 作物杂志, 2022, (6): 61–69 |
| [5] | 齐广勋, 董岭超, 张伟, 袁翠平, 刘晓冬, 王英男, 董英山, 王玉民, 赵洪锟. 国外大豆种质资源对大豆花叶病毒株系3(SMV3)的抗性评价[J]. 作物杂志, 2022, (6): 70–74 |
| [6] | 雷蕾, 关哲允, 曹士亮, 王玉民, 林春晶, 彭宝, 刘鹏, 赵丽梅, 李志刚, 张春宝. 基于产量相关性状SSR分子标记的大豆杂种优势群划分[J]. 作物杂志, 2022, (4): 54–61 |
| [7] | 王思宇, 左文博, 朱凯莉, 郭慧敏, 邢宝, 郭雨晴, 包玉英, 杨修仕, 任贵兴. 71份藜麦品种资源的农艺性状及营养品质分析与评价[J]. 作物杂志, 2022, (3): 63–72 |
| [8] | 赵艳飞, 王继永. 种质资源保护和育种创新现状及发展对策——以湖南和海南两省为例[J]. 作物杂志, 2022, (2): 1–5 |
| [9] | 李文略, 陈常理, 骆霞虹, 柳婷婷, 安霞, 金关荣, 朱关林. 浙江红麻资源表型性状的遗传多样性分析[J]. 作物杂志, 2022, (1): 50–55 |
| [10] | 郜战宁, 王树杰, 冯辉, 薛正刚, 杨永乾, 宋晓朋, 介元芬. 二棱大麦品种(系)的综合评价[J]. 作物杂志, 2022, (1): 70–76 |
| [11] | 张全芳, 姜明松, 陈峰, 朱文银, 周学标, 杨连群, 徐建第. 山东省水稻品种(系)的遗传多样性分析[J]. 作物杂志, 2021, (4): 26–31 |
| [12] | 李琼, 常世豪, 武婷婷, 耿臻, 杨青春, 舒文涛, 李金花, 张东辉, 张保亮. 120份大豆种质资源遗传多样性和亲缘关系分析[J]. 作物杂志, 2021, (4): 51–58 |
| [13] | 贾瑞玲, 赵小琴, 南铭, 陈富, 刘彦明, 魏立平, 刘军秀, 马宁. 64份苦荞种质资源农艺性状遗传多样性分析与综合评价[J]. 作物杂志, 2021, (3): 19–27 |
| [14] | 项超, 孙素丽, 朱振东, 宗绪晓, 杨涛, 刘荣, 杨梅, 鲜东锋, 杨秀燕. 四川豌豆种质资源白粉病抗性及分子鉴定[J]. 作物杂志, 2021, (3): 51–56 |
| [15] | 潘晓雪, 胡明瑜, 王忠伟, 吴红, 雷开荣. 不同水稻种质资源重要农艺性状与发芽期耐寒性鉴定研究[J]. 作物杂志, 2021, (1): 47–53 |
|
||