目前,对作物遗传多样性的鉴定主要采用形态学和分子标记技术[6]。形态学标记侧重于观察作物表型性状的差异,较为直观,但易受气候环境、地理变异和人为因素的影响。随着生物技术育种的迅速发展,分子标记技术成为基因图谱构建和定位、遗传多样性分析和杂交后代鉴定的重要工具,其具有基因覆盖完全、重复性好、多态性高的优点,且不受材料、环境和季节的影响[7]。基于PCR的简单序列重复(simple sequence repeat,SSR)分子标记技术是以1~6个碱基为重复组成单元的串联重复序列,具有植物基因覆盖全面、共显性、频带稳定、多态性高及价格低廉的特点,该技术被广泛应用于马铃薯杂交种鉴定、遗传差异分析、标记开发等方面[8-
SSR技术在马铃薯无性系的多样性分析和鉴定中也有广泛研究。Sapinder等[15]利用23个高度多态性的全基因组SSR标记分析了264个赤褐色和非赤褐色无性后代的DNA指纹图谱,发现整个育种材料具有显著多样性,并且多样性在所有区域育种计划中均匀分布。Karaagac等[16]利用25个SSR标记对54个马铃薯品种(系)进行基因型分析,划分为薯条、薯片和鲜食三大类。明确品种无性后代群体的遗传差异和亲缘关系可快速构建核心种质,对品种改良及育成至关重要。Pandey等[17]利用单核苷酸多态性(SNP)标记对214个马铃薯无性系进行基因型鉴定,获得了43个核心种质,占总数的20%,最终将核心种质作为重要资源进行收集保存。利用SSR分子标记技术对350个马铃薯栽培品种的遗传多样性进行了分析,获得了48个核心种质[18]。利用SSR标记从喀麦隆西部高地地区的138份马铃薯品种中获得27个核心基因型[19]。McGregor等[20]利用AFLP标记技术分析了荷兰遗传资源中心保存的314个野生马铃薯种质系列的遗传多样性,发现地理距离与种质资源遗传多样性之间存在不一致的关系。Moisan-Thiery等[21]利用4~5个SSR标记对286个法国马铃薯品种进行了完全鉴定。Galarreta等[22]利用SSR分子标记对西班牙105个地方马铃薯品种进行了评价,结果与主成分分析(PCA)的结果基本一致。前人对马铃薯种质资源的研究方法准确可靠,根据其亲缘关系对马铃薯种质资源进行分类,为后期杂交亲本的选择和选配提供依据。
目前,乌兰察布地区马铃薯的主栽品种主要是从国外或区外引进,自主选育的马铃薯品种较少。本单位保存的马铃薯种质资源之间的亲缘关系尚不明确,存在杂交亲本盲目选择和组合等问题。本研究利用SSR分子标记技术对63份马铃薯优异种质材料进行遗传多样性研究,旨在明确其亲缘关系,并建立其遗传指纹图谱,加强马铃薯种质资源的保存和高效利用,加快乌兰察布地区马铃薯品种选育和改良进程。
1 材料与方法
1.1 供试材料
试验材料为63份马铃薯种质资源(表1),其中Mira和Agria为荷兰资源,Russet Burbank和Jardinor分别为美国和德国资源,3个YSZ野生种为二倍体,其余56个为现有马铃薯种质资源。试验于2021年4-10月在集宁师范学院园艺实训基地(113°16′ E,40°93′ N)进行,海拔1396 m,无霜期110 d。63份马铃薯资源采用高垄种植,随机排列,每个种质资源种植20株,株距25 cm×90 cm。
表1 供试马铃薯品种(系)
Table 1
| 编号 Number | 品种(系) Variety (line) | 编号 Number | 品种(系) Variety (line) | 编号 Number | 品种(系) Variety (line) | 编号 Number | 品种(系) Variety (line) | 编号 Number | 品种(系) Variety (line) |
|---|---|---|---|---|---|---|---|---|---|
| ZY001 | YSZ-1 | ZY014 | YS165 | ZY027 | 晋薯16号 | ZY040 | 青薯168 | ZY053 | 东农310 |
| ZY002 | YSZ-2 | ZY015 | Agria | ZY028 | 同薯24号 | ZY041 | 陇薯7号 | ZY054 | Jardinor |
| ZY003 | Russet Burbank | ZY016 | 大同里外黄 | ZY029 | 鄂马铃薯16号 | ZY042 | 高原7号 | ZY055 | 丽薯15号 |
| ZY004 | 中加1号 | ZY017 | YS177 | ZY030 | YS179 | ZY043 | Favorita | ZY056 | YS189 |
| ZY005 | 内农薯2号 | ZY018 | 后旗红 | ZY031 | 晋薯26号 | ZY044 | 希森6号 | ZY057 | YS195 |
| ZY006 | YS152 | ZY019 | 鄂马铃薯14号 | ZY032 | 青薯9号 | ZY045 | 中薯早35 | ZY058 | 早大白 |
| ZY007 | 冀张薯8号 | ZY020 | 民丰红 | ZY033 | YS178 | ZY046 | 希森8号 | ZY059 | 尤金 |
| ZY008 | YS098 | ZY021 | 冀张薯15号 | ZY034 | 同薯20号 | ZY047 | 陇薯9号 | ZY060 | 内农薯1号 |
| ZY009 | 同薯28号 | ZY022 | 华渝8号 | ZY035 | 中薯3号 | ZY048 | 京张薯1号 | ZY061 | 冀张薯14号 |
| ZY010 | 康尼贝克 | ZY023 | 云薯304 | ZY036 | 北方001 | ZY049 | 兴佳2号 | ZY062 | 克新27号 |
| ZY011 | 华渝5号 | ZY024 | 郑薯5号 | ZY037 | LK99 | ZY050 | 龙薯7号 | ZY063 | 克新25号 |
| ZY012 | Mira | ZY025 | 中薯5号 | ZY038 | 甘农薯7号 | ZY051 | 北方002 | ||
| ZY013 | YSZ-3 | ZY026 | 冀张薯12号 | ZY039 | 中加2号 | ZY052 | 京张薯3号 |
1.2 马铃薯基因组DNA提取
在马铃薯苗期,随机取63份马铃薯材料的健康、新鲜的幼嫩叶片,采用植物基因组DNA提取试剂盒[天根生化科技(北京)有限公司]提取叶片的基因组DNA,用1.0%琼脂糖凝胶电泳检测DNA纯度,置于-40 ℃冰箱中保存备用。
1.3 SSR引物来源及筛选
本试验共132对SSR引物,其中79对碱基序列来源于NCBI网站上公布的马铃薯SSR特异性引物,53对C系列引物的碱基序列由本课题组自行开发。课题组在马铃薯基因组网站(
表2 SSR引物序列
Table 2
| 引物名称Primer name | 正向引物Forward primer(5’-3) | 反向引物Reversed primer(5’-3’) |
|---|---|---|
| C3 | CCGGGAGAATTAGACGATGA | TTTATGGGGAGCAGTTGAGG |
| C32 | CCCCTTACCCCTCTTCAATC | AACCCGTGTCAACTTCTGCT |
| C55 | ACTGCTGTGGTTGGCGTC | ACGGCATAGATTTGGAAGCATC |
| C57 | GACTGGCTGACCCTGAACTC | GACAAAATTACAGGAACTGCAAA |
| C59 | TCATCACAACGTGACCCCA | GGGCTTGAATGATGTGAAGCTC |
| C61 | CGCAAATCTTCATCCGATTC | TCCGGCGGATAATACTTGTT |
| S25 | GCGAATGACAGGACAAGAGG | TGCCACTGCTACCATAACCA |
| S007 | GACTGGCTGACCCTGAACTC | GACAAAATTACAGGAACTGCAAA |
| S118 | AGAGATCGATGTAAAACACGT | GTGGCATTTTGATGGATT |
| S122 | GACACGTTCACCATAAAA | AGAAGAATAGCAAAGCAA |
| S148 | CAGCAAAATCAGAACCCGAT | GGATCATCAAATTCACCGCT |
| S151 | GCTGCTAAACACTCAAGCAGAA | CAACTACAAGATTCCATCCACAG |
| S153 | TATGTTCCACGCCATTTCAG | ACGGAAACTCATCGTGCATT |
| S168 | CTGCCGCAAAAAGTGAAAAC | TGAATGTAGGCCAAATTTTGAA |
| S170 | CGCAAATCTTCATCCGATTC | TCCGGCGGATAATACTTGTT |
| S174 | TGAGGGTTTTCAGAAAGGGA | CATCCTTGCAACAACCTCCT |
| S184 | TCATCACAACGTGACCCCA | GGGCTTGAATGATGTGAAGCTC |
| S189 | CCTTGTAGTACAGCAGTGGTC | TCCGCCAAGACTGATGCA |
| S192 | AATTCATGTTTGCGGTACGTC | ATGCAGAAAGATGTCAAAATTGA |
| STI005 | CGCAAATCTTCATCCGATTC | TCCGGCGGATAATACTTGTT |
| STI027 | ACTGCTGTGGTTGGCGTC | ACGGCATAGATTTGGAAGCATC |
| STI049 | TCATCACAACGTGACCCC | CGGGCTTGAATGATGTGAAGCT |
| STACC | ACAGGAATCACACCTGCACA | TTCAACATCCGCCTGTCATA |
| STGBSS | AATCGGTGATAAATGTGAATGC | ATGCTTGCCATGTGATGTGT |
| STL001 | CAGCAAATCAGAACCCGAT | GGATCATCAAATTCACCGCT |
| STL002 | ACAGGAATCACACCTGCACA | TTCAACATCCGCCTGTGATA |
| STM0030 | AGAGATCGATGTAAAACACGT | GTGGCATTTTGATGGATT |
| STM1031 | GTGGTTTGTTTTTCTGTAT | AATTCTATCCTCATCTCTA |
| STM1058 | ACAATTTAATTCAAGAAGCTAGG | CCAAATTTGTATACTTCAATCTGA |
| STM1064 | GTTCTTTTGGTGGTTTTCCT | TTATTTCTCTGTTGTTGCTG |
| STM1104 | TGATTCTCTTGCCTACTGTAATCG | GAAAGTGGTGTGAAGCTGTGA |
| STM2030 | TCTTCCCAAATCTAGAATACATGC | AAAGTTAGCATGGACAGCATTTC |
1.4 SSR扩增系统和程序
SSR扩增体系详见表3,其中Mg2+、DNA聚合酶、dNTPs、Buffer和DNA marker DL100均来源于北京全式金生物技术股份有限公司。
表3 SSR-PCR反应体系
Table 3
| 编号 Number | 体系组分 System component | 浓度 Concentration | 用量 Dosage (μL) |
|---|---|---|---|
| 1 | ddH2O | - | 14.1 |
| 2 | 10×Buffer (Mg2+) | 50 μmol/L | 2.0 |
| 3 | dNTPs | 2.5 mmol/L | 1.6 |
| 4 | Taq DNA聚合酶 | 5 U/μL | 0.1 |
| 5 | 正向引物 | 50 μmol/L | 0.6 |
| 6 | 反向引物 | 50 μmol/L | 0.6 |
| 7 | DNA模板 | 50 ng/μL | 1.0 |
| 8 | 总体积 | - | 20.0 |
SSR扩增程序:95 ℃预变性4 min;94 ℃变性30 s,56 ℃退火45 s,72 ℃延伸90 s,36个循环;72 ℃延伸10 min[16]。试验用PCR仪为上海皓庄仪器有限公司生产的LNB48+型号。
1.5 聚丙烯酰胺凝胶电泳和银染检测
用8%非变性聚丙烯酰胺凝胶电泳对上述63个马铃薯材料的PCR产物进行检测,上样量为4 μL,功率70 W,电泳1.5 h,试验用电泳仪为北京六一生物科技有限公司DYY-12型[23]。
银染步骤:(1)固定液(蒸馏水2 L、酒精200 mL、冰乙酸20 mL)中固定15 min;(2)漂洗3 min;(3)用1.5% AgNO3溶液染色15 min;(4)速漂5 s;(5)显影液(蒸馏水2 L、NaOH60 g、甲醛10 mL)显色10 min;(6)漂洗;(7)扫描胶板。
1.6 数据处理与分析
数据用[1,0]赋值法,即胶板上有条带的记为‘1’,无带的记为‘0’。用PIC-Calc0.6计算多态性信息含量(polymorphism information contents,PIC)[24];用NTSYS 2.10e计算遗传距离和相似系数,利用MEGA 7.0绘制聚类图[25];用Popgene 32计算有效等位基因数(number of effective alleles,Ne)、Shannon信息指数(shannon information index,I)、Nei’s遗传多样性指数(Nei’s genetic diversity,H)和多态条带比率(percentage of polymorphic bands,PPB);使用QR Code 2.14对供试品种(系)的SSR电泳结果进行QR编码[26]。
2 结果与分析
2.1 63份马铃薯种质资源的DNA分析结果
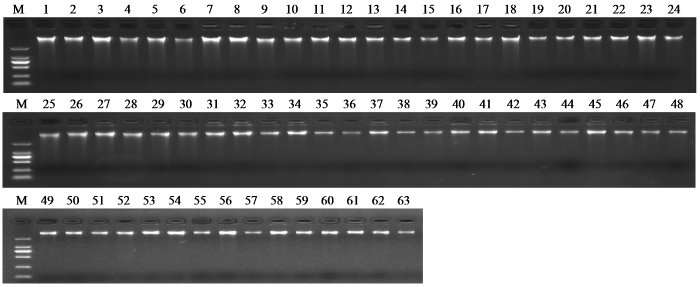
图1是63份马铃薯种质材料的DNA检测结果。63份马铃薯种质材料基因组DNA条带清晰,无蛋白质和RNA残留,无拖尾现象。经微量分光光度计测定,63份马铃薯种质资源的基因组DNA浓度介于500~1000 ng/μL,可满足SSR遗传多样性分析。
图1
图1
63份马铃薯材料基因组DNA纯度检测
M:DNA分子量标准DL2000,1~63表示ZY001~ZY063品种,下同。
Fig.1
Genomic DNA purity test of 63 potato materials
M: DL2000 DNA marker, 1-63 indicate variety ZY001-ZY063, the same below.
2.2 63份马铃薯种质资源的SSR标记分析
2.2.1 SSR标记的遗传多样性分析
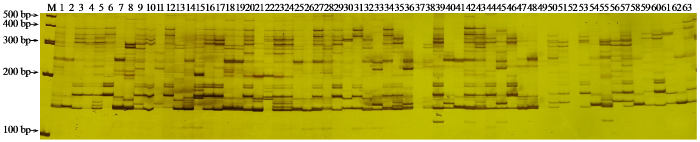
图2
图2
引物S118对63份马铃薯品种(系)的扩增结果
Fig.2
Amplification results of S118 primer for 63 potato varieties (lines)
用筛选出的32对SSR引物对63份马铃薯种质材料遗传多样性的分析,结果(表4)显示,32对引物共扩增出164个等位位点,其中多态性位点145个,各引物扩增的等位位点数介于2~12,平均等位位点数5.13;各引物PCR扩增产物大小集中在100~600 bp,PPB介于75.00%~100.00%,平均为90.61%;PIC介于0.4778~0.9367,平均为0.8144,其中引物STM0030的PIC(0.9367)最高,多态性位点数也最多,与PIC结果一致,表明其遗传多样性信息丰富。
表4 基于32对SSR引物的马铃薯材料遗传多样性分析
Table 4
| 引物 Primer | 退火温度 Tm (℃) | 多态性条带数 NBP | 总条带数 Total bands | 多态条带比率 PPB (%) | 有效等位基因 Ne | Nei’s遗传多样性指数 H | Shannon指数 I | 多态信息含量 PIC |
|---|---|---|---|---|---|---|---|---|
| C3 | 56 | 3 | 4 | 75.00 | 1.6965 | 0.3607 | 0.5054 | 0.8108 |
| C32 | 55 | 2 | 2 | 100.00 | 1.8985 | 0.4718 | 0.6645 | 0.6712 |
| C55 | 55 | 3 | 3 | 100.00 | 1.7493 | 0.4229 | 0.6128 | 0.7951 |
| C57 | 54 | 2 | 2 | 100.00 | 1.8438 | 0.4573 | 0.6498 | 0.5318 |
| C59 | 53 | 3 | 4 | 75.00 | 1.5632 | 0.3200 | 0.4631 | 0.7484 |
| C61 | 57 | 4 | 5 | 80.00 | 1.6947 | 0.3715 | 0.5256 | 0.8381 |
| S25 | 55 | 7 | 9 | 77.78 | 1.7241 | 0.3744 | 0.5245 | 0.9198 |
| S7 | 60 | 2 | 2 | 100.00 | 1.8331 | 0.4500 | 0.6413 | 0.6683 |
| S118 | 58 | 8 | 10 | 80.00 | 1.5121 | 0.3275 | 0.4668 | 0.9248 |
| S122 | 50 | 3 | 3 | 100.00 | 1.5146 | 0.3379 | 0.5202 | 0.6927 |
| S148 | 53 | 3 | 4 | 75.00 | 1.5926 | 0.3307 | 0.4745 | 0.8231 |
| S151 | 56 | 3 | 3 | 100.00 | 1.5545 | 0.3494 | 0.5310 | 0.7959 |
| S153 | 53 | 7 | 8 | 87.50 | 1.8486 | 0.4306 | 0.5996 | 0.9085 |
| S168 | 53 | 2 | 2 | 100.00 | 1.8504 | 0.4579 | 0.6501 | 0.4778 |
| S170 | 53 | 7 | 8 | 87.50 | 1.7930 | 0.4144 | 0.5828 | 0.9142 |
| S174 | 52 | 3 | 3 | 100.00 | 1.8569 | 0.4585 | 0.6503 | 0.7954 |
| S184 | 53 | 4 | 5 | 80.00 | 1.7028 | 0.3730 | 0.5269 | 0.8500 |
| S189 | 53 | 4 | 4 | 100.00 | 1.8574 | 0.4733 | 0.6661 | 0.8483 |
| S192 | 54 | 6 | 7 | 85.71 | 1.6462 | 0.3635 | 0.5261 | 0.9084 |
| STI005 | 56 | 6 | 7 | 85.71 | 1.7180 | 0.3878 | 0.5521 | 0.9033 |
| STI027 | 55 | 5 | 6 | 83.33 | 1.5798 | 0.3330 | 0.4883 | 0.8766 |
| STI049 | 53 | 3 | 3 | 100.00 | 1.9212 | 0.4790 | 0.6719 | 0.7895 |
| STACC | 55 | 4 | 4 | 100.00 | 1.4989 | 0.3322 | 0.5141 | 0.8521 |
| STGBSS | 54 | 3 | 3 | 100.00 | 1.4977 | 0.3304 | 0.5117 | 0.7962 |
| STL001 | 56 | 6 | 7 | 85.71 | 1.6921 | 0.3778 | 0.5410 | 0.8971 |
| STL002 | 55 | 3 | 4 | 75.00 | 1.6706 | 0.3527 | 0.4971 | 0.7754 |
| STM0030 | 52 | 11 | 12 | 91.67 | 1.7709 | 0.4142 | 0.5893 | 0.9367 |
| STM1031 | 50 | 5 | 5 | 100.00 | 1.7607 | 0.4217 | 0.6097 | 0.8660 |
| STM1058 | 51 | 4 | 4 | 100.00 | 1.9674 | 0.4914 | 0.6845 | 0.8102 |
| STM1064 | 56 | 6 | 7 | 85.71 | 1.6896 | 0.3702 | 0.5304 | 0.8880 |
| STM1104 | 57 | 5 | 5 | 100.00 | 1.5269 | 0.3082 | 0.4562 | 0.8210 |
| STM2030 | 55 | 8 | 9 | 88.89 | 1.8170 | 0.4234 | 0.5945 | 0.9262 |
63个马铃薯种质材料的Ne范围在1.4977~ 1.9674,平均为1.7139;H介于0.3082~0.4914,平均为0.3927;I范围在0.4562~0.6845,平均为0.5632;其中引物STM1058的Ne(1.9674)、H(0.4914)和I(0.6845)均表现为最高。综上所述,63个马铃薯品种(系)的遗传多样性处于中等水平。
2.2.2 63个马铃薯品种(系)的遗传距离和聚类分析
由表5可知,63份材料遗传距离(GD值)介于0.12~0.71,平均为0.297,表明各材料间存在不同程度的遗传差异。其中,遗传距离最大的为YSZ-1与青薯168,二者相似系数最小,为0.500,表明其亲缘关系最远;遗传距离最小的为冀张薯8号和冀张薯15号,二者相似系数最大,为0.810,表明其亲缘关系最近。
表5 63份马铃薯品种(系)遗传距离
Table 5
| 遗传距离区间 Genetic distance interval | 平均遗传距离 Mean genetic distance | 品种间最大遗传距离相似系数 Similarity coefficient of maximum genetic distance between varieties | 品种间最小遗传距离相似系数 Similarity coefficient of minimum genetic distance between varieties |
|---|---|---|---|
| 0.12~0.71 | 0.297 | 0.500 | 0.810 |
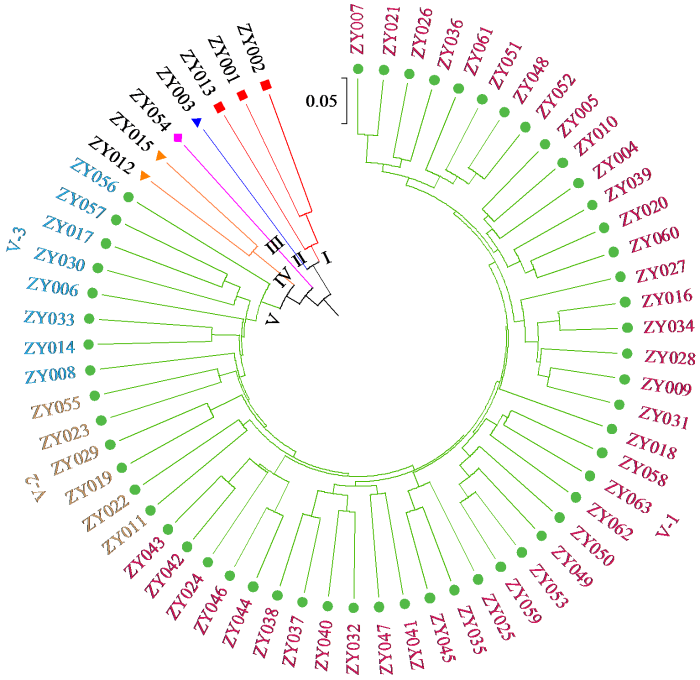
由图3可知,GD值为0.180时,将63份马铃薯供试材料分为5个类群。其中,YSZ-1、YSZ-2和YSZ-3聚为类群Ⅰ,占总数的4.76%,主要为二倍体野生资源;Russet Burbank单独聚为类群Ⅱ,占总数的1.59%,为引进的美国资源;Jardinor单独聚为类群Ⅲ,占总数的1.59%,为引进的德国资源;Mira和Agria聚为类群Ⅳ,占总数的3.17%,为引进的荷兰资源;其余56个品种资源聚为类群Ⅴ,是材料最多的类群,占总数的88.89%,主要包括中国各地的种质资源。
图3
在GD值为0.13处类群Ⅴ分为3个亚群Ⅴ-1、Ⅴ-2和Ⅴ-3。Ⅴ-1亚群包含42个我国北方马铃薯种质资源,其中东北区域资源7个,分别为早大白、克新25号、克新27号、龙薯7号、兴佳2号、东农310和尤金;西北区域资源6个,分别为陇薯7号、陇薯9号、青薯9号、青薯168、LK99和甘农薯7号;其余为华北、华中区域资源29个,主要为中薯系列、冀张薯系列、北方系列、内薯系列、郑薯系列、晋薯系列和希森系列。Ⅴ-2亚群包括我国南方马铃薯资源6个,分别为华渝5号、华渝8号、鄂马铃薯14号、鄂马铃薯16号、云薯304和丽薯15号。Ⅴ-3亚群为黑龙江省农业科学院克山分院引进的8个YS资源,分别为YS098、YS152、YS165、YS177、YS178、YS179、YS189和YS195。
分析63份马铃薯种质资源的亲本来源发现,杂交选育品种44个,占比为69.80%,中薯3号天然结实后代选育品种1个(中薯5号),芽变选育品种1个(中加1号),Burbank突变体选育品种1个(Russet Burbank),组培苗特异植株选育品种1个(LK99),占比均为1.59%,亲本来源不明确的品种15个(野生种系列3个、YS系列8个、后旗红、康尼贝克、华渝8号和同薯24号),占比为23.80%;根据育成品种的熟性可知,早熟品种7个(云薯304、郑薯5号、中薯5号、中薯3号、中薯早35、早大白和克新25号);中早熟材料5个(华渝5号、冀张薯15号、LK99、北方002和尤金);中熟材料9个(内农薯2号、北方001、Favorita、希森6号、希森8号、京张薯1号、兴佳2号、龙薯7号和内农薯1号);晚熟材料15个(野生种系列3个、Russet Burbank、中加1号、冀张薯8号、康尼贝克、后旗红、甘农薯7号、青薯168、陇薯7号、YS165、YS177、YS178和YS179);中晚熟材料27个;根据育成品种的用途发现,鲜食品种53个,占84.13%,加工品种5个(Russet Burbank、Agria、云薯304、甘农薯7号和东农310);鲜食、加工兼用品种5个(内农薯2号、大同里外黄、晋薯16号、LK99和克新27号);根据品种选育时间可知,1900-2000年间育成品种7个(Russet Burbank、Mira、Agria、青薯168、高原7号、Favorita和Jardinor),13个品种(系)登记时间不明确(3个野生种系列、8个YS系列、华渝8号和同薯24号),2000-2024年育成品种43个;分析亲本来源发现,除15个品种的亲本不详外,其余48个品种由86个亲本育成,包括国内品种25个,国外品种23个,其余38个亲本均为代号,不明确其来源,由大西洋为母本育成的品种有4个(同薯28号、冀张薯12号、甘农薯7号和内农薯1号),由陇薯系列为父本育成的品种有3个(内农薯1号、内农薯2号和甘农薯7号)。此外,我们还发现63份马铃薯种质资源聚类结果与地域关联较大,可为下一步种质资源的利用、改良及杂交亲本的选配提供理论依据。
2.2.3 主成分分析
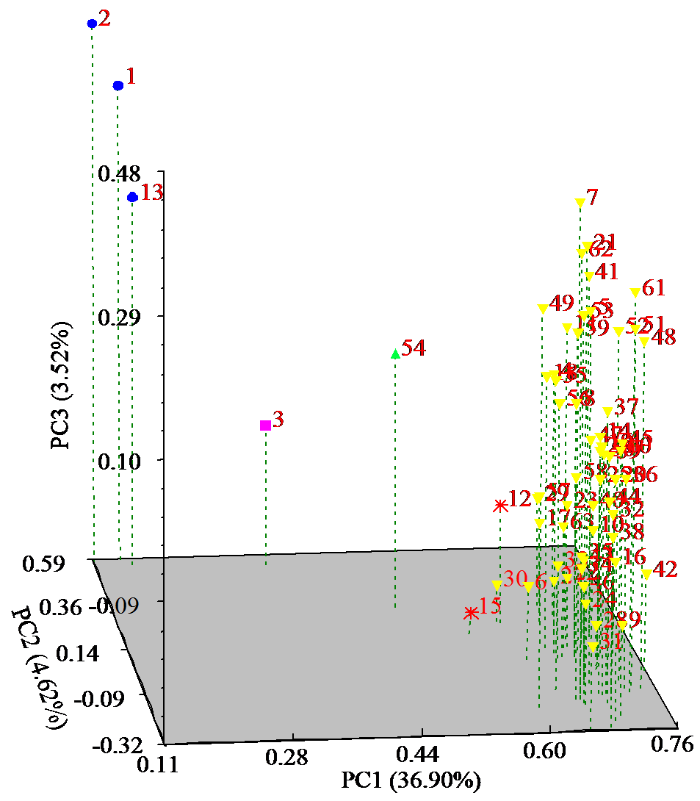
图4为63份马铃薯种质资源的PCA分析结果,结果表明PC1、PC2和PC3变异系数分别为36.90%、4.62%和3.52%,总变异系数为45.04%。可将63个种质资源划分为5个类群(G1~G5),其中G1包括3个材料,均为二倍体野生种资源;G2包括1个材料,为美国资源;G3包括1个材料,为德国资源;G4包括2个材料,为荷兰资源;G5包括56个材料,为国内各地区马铃薯品种。PCA结果与上述聚类结果一致,表明各品种资源的分类与地域高度关联。
图4
图4
63份马铃薯种质资源遗传多样性的主成分分析
Fig.4
Principle component analysis of genetic diversity of 63 potato germplasm resources
2.2.4 指纹图谱构建
根据63份马铃薯种质资源的SSR指纹图谱结果(表6)可知,引物STL001可将马铃薯种质资源中YSZ-3、YS165、Agria、大同里外黄、民丰红、冀张薯15号、晋薯26号、北方001、陇薯9号、龙薯7号、YS189、YS195和冀张薯14号共13个种质资源进行区分;除上述13个种质资源外,引物S192可将其他50个种质材料中的46个种质材料进行区分,而冀张薯8号与早大白、Mira与中薯5号带型一致,无法区分;引物S118可将上述4个种质材料进行区分。综上所述,利用3对SSR引物构建了63份马铃薯种质材料的指纹图谱,为后期种质资源利用、改良和品种知识产权保护提供理论依据。
表6 63份马铃薯品种(系)的SSR指纹图谱
Table 6
| 编号 Number | 品种(系) Variety (line) | 指纹Fingerprint | 编号 Number | 品种(系) Variety (line) | 指纹Fingerprint | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| STL001 | S192 | S118 | STL001 | S192 | S118 | |||||||||||||
| ZY001 | YSZ-1 | 1010001 | 0000001 | 1101111111 | ZY009 | 同薯28号 | 1011111 | 0111011 | 1111111111 | |||||||||
| ZY002 | YSZ-2 | 1110001 | 1111111 | 1101100111 | ZY010 | 康尼贝克 | 1111011 | 0111011 | 1110111111 | |||||||||
| ZY003 | Russet Burbank | 1011001 | 1011101 | 1111111111 | ZY011 | 华渝5号 | 1111001 | 1111101 | 1001100111 | |||||||||
| ZY004 | 中加1号 | 1111001 | 0000001 | 1110111111 | ZY012 | Mira | 1111001 | 1000111 | 1111111111 | |||||||||
| ZY005 | 内农薯2号 | 1011001 | 0110011 | 1111111101 | ZY013 | YSZ-3 | 1010011 | 0110001 | 1100100111 | |||||||||
| ZY006 | YS152 | 1011111 | 0000111 | 0111111111 | ZY014 | YS165 | 1001011 | 0000101 | 1101100111 | |||||||||
| ZY007 | 冀张薯8号 | 1110001 | 1110111 | 1101100011 | ZY015 | Agria | 0111001 | 0011001 | 1111111111 | |||||||||
| ZY008 | YS098 | 1110001 | 0010111 | 1101101001 | ZY016 | 大同里外黄 | 1110011 | 1011101 | 1111111111 | |||||||||
| ZY017 | YS177 | 1111011 | 0010111 | 1111111111 | ZY041 | 陇薯7号 | 1111101 | 0101111 | 1101100101 | |||||||||
| ZY018 | 后旗红 | 1111001 | 1110111 | 1101111111 | ZY042 | 高原7号 | 1011101 | 0100111 | 1111111111 | |||||||||
| ZY019 | 鄂马铃薯14号 | 1110001 | 1010111 | 1101111111 | ZY043 | Favorita | 1111111 | 0100111 | 1111111111 | |||||||||
| ZY020 | 民丰红 | 1000001 | 0000001 | 1111111111 | ZY044 | 希森6号 | 1111111 | 0110111 | 1110111011 | |||||||||
| ZY021 | 冀张薯15号 | 1110111 | 0110111 | 1101100011 | ZY045 | 中薯早35 | 1111111 | 1110111 | 1101100011 | |||||||||
| ZY022 | 华渝8号 | 1111011 | 0000101 | 1111111111 | ZY046 | 希森8号 | 1011111 | 1100111 | 1110111011 | |||||||||
| ZY023 | 云薯304 | 0000001 | 0111011 | 1111111001 | ZY047 | 陇薯9号 | 0101101 | 0100011 | 1101100011 | |||||||||
| ZY024 | 郑薯5号 | 1111111 | 0111011 | 1111111001 | ZY048 | 京张薯1号 | 1111101 | 1100111 | 1101100011 | |||||||||
| ZY025 | 中薯5号 | 1111001 | 1000111 | 1111111011 | ZY049 | 兴佳2号 | 1011111 | 0111101 | 0000111001 | |||||||||
| ZY026 | 冀张薯12号 | 1111001 | 0011111 | 1101100111 | ZY050 | 龙薯7号 | 1100101 | 0111111 | 1111110001 | |||||||||
| ZY027 | 晋薯16号 | 1110001 | 1010001 | 1111111111 | ZY051 | 北方002 | 1111011 | 0110111 | 1111100001 | |||||||||
| ZY028 | 同薯24号 | 1111111 | 1011011 | 1101111111 | ZY052 | 京张薯3号 | 1111101 | 1111111 | 0000100001 | |||||||||
| ZY029 | 鄂马铃薯16号 | 1111011 | 1000111 | 1111111111 | ZY053 | 东农310 | 1111101 | 0111011 | 1110110001 | |||||||||
| ZY030 | YS179 | 0000001 | 0000011 | 1100111111 | ZY054 | Jardinor | 1011111 | 0000001 | 1101101111 | |||||||||
| ZY031 | 晋薯26号 | 0001111 | 1011111 | 1110111101 | ZY055 | 丽薯15号 | 1111001 | 1111011 | 1101100011 | |||||||||
| ZY032 | 青薯9号 | 1111101 | 1000001 | 1101110101 | ZY056 | YS189 | 1101101 | 0010001 | 1111100001 | |||||||||
| ZY033 | YS178 | 0000001 | 0000111 | 1101100111 | ZY057 | YS195 | 0111011 | 0000001 | 1111101111 | |||||||||
| ZY034 | 同薯20号 | 1110001 | 1100011 | 1111111111 | ZY058 | 早大白 | 1011111 | 1111111 | 1101111111 | |||||||||
| ZY035 | 中薯3号 | 1111111 | 1111011 | 1110111011 | ZY059 | 尤金 | 1011101 | 0110111 | 1100110011 | |||||||||
| ZY036 | 北方001 | 0100001 | 0110111 | 1101100111 | ZY060 | 内农薯1号 | 1101001 | 0000001 | 1111111101 | |||||||||
| ZY037 | LK99 | 1010001 | 1011001 | 0000100001 | ZY061 | 冀张薯14号 | 1111001 | 1111111 | 1101100001 | |||||||||
| ZY038 | 甘农薯7号 | 1011111 | 1000011 | 1001100001 | ZY062 | 克新27号 | 1110001 | 1110111 | 1101101101 | |||||||||
| ZY039 | 中加2号 | 1110001 | 0111011 | 1111111111 | ZY063 | 克新25号 | 1011101 | 0000011 | 1111111111 | |||||||||
| ZY040 | 青薯168 | 1111111 | 0111011 | 1101100101 | ||||||||||||||
2.3 63份马铃薯种质资源的QR码
图5为63份马铃薯品种(系)的QR(quick response)编码,编码信息为每个马铃薯品种(系)指纹图谱和SSR标记电泳信息,可通过编码图扫码查看。QR编码可用于马铃薯种质资源利用改良、亲本选配及品种鉴定等方面,有效避免引进品种的同名异种、同种异名及种质资源间亲缘关系不明确等问题,明确了63份马铃薯品种(系)的遗传信息,可极大提高育种效率。
图5
3 讨论
分子标记是一种基于DNA序列的标记。在生物基因组中,DNA序列差异是生物遗传多样性的直接体现。通过DNA水平的聚类分析来探索植物品种间的遗传差异,可以减少亲本选择的盲目性,提高育种效率。目前,SSR和SNP分子标记是分析遗传多样性和群体结构的可靠方法,检测结果灵敏度高。SSR分子标记技术是一种条带稳定、多态性高的共显性标记,广泛应用于群体结构和遗传差异分析、指纹图谱构建等方面。
Singh等[29]利用SSR和SNP分子标记对375份水稻(Oryza sativa L.)进行了遗传多样性分析,结果表明,SSR标记共扩增出80个等位基因,SNP标记共扩增出72个等位基因。SSR的平均多态性信息含量为0.25,SNP的为0.23。主成分分析结果表明,SSR标记在K=5时Δk最大,而SNP标记在K=15时Δk最大,说明SNP标记的群体分辨率较高,SSR标记在多样性分析上更有效。
Carvalho等[30]用23个SSR和251个SNP标记对206个巴西地方菜豆品种和59个优良菜豆品系进行了遗传多样性分析,认为SSR和SNP分子标记对基因型具有较高的区分能力。在马铃薯遗传多样性分析中,可应用的分子标记类型种类丰富,其中简单重复序列(SSR)与单核苷酸多态性(SNP)应用最为广泛。研究[31-32]表明,对于群体规模较大的组合,其遗传参数可通过SSR标记信息进行估计,这是因为SSR标记不仅被证实适用于群体遗传推断,且相较于双等位基因标记,具有更高的信息含量。Wu等[33]认为SSR是植物遗传研究中信息量最大的分子标记之一。迄今为止,通过高通量测序在许多植物中开发了大量SSR标记,如Angelica gigas[34]、Lentinula edodes[35]和Spondias tuberosa[36]。Jayabalan等[37]利用28个全基因组SSR标记、11个盐响应基因SSR标记和8个Saltol QTL连锁SSR标记,对来自印度地理差异较大的沿海地区的47个水稻地方品种的遗传亲缘性和群体结构进行了估计,结果将地方品种分为5个不同的种群,分类结果与它们的地理位置相关。Emanuelli等[38]利用SSR和SNP标记分析了2273个驯化葡萄品种(Vitis vinifera)的遗传多样性,结果表明,SSR位点的平均遗传多样性(0.81)高于SNP(0.34)。SNP是基因组中最丰富的标记,在深层进化尺度上最不容易饱和,而SSR在物种的个体之间以及分化的种群之间具有高度多态性[39]。
育种的有效性在很大程度上取决于可利用种质资源的数量及其性状表现和遗传模式研究的深度,因此,准确全面地评价马铃薯种质资源之间的形态差异和遗传多样性对种质资源的挖掘、利用和保护具有重要意义[40]。植物表型性状的分析方法主要有聚类分析、主成分分析和群体结构分析。潘哲超等[41]利用马铃薯新品系形态特征聚类分析来选育新品种。然而,表型性状分析结果容易受环境影响,准确性和可靠性不高。目前,利用生物技术在遗传水平上分析马铃薯、小麦和水稻的遗传差异,操作简便,结果准确可靠,可补充表型性状分析,降低植物生长发育和环境差异的影响,提高育种选择效率。张招娟等[42]利用SSR技术将19个马铃薯品种划分为5个类群,发现遗传多样性丰富,品种间差异较大。宋峥等[43]利用10对SSR引物对湖北44份马铃薯资源进行遗传多样性分析,将其划分为5类,并建立指纹图谱,为后期的亲本选择和品种鉴定奠定了基础。El-Esawi等[44]利用AFLP标记技术对25份爱尔兰甘蓝材料的遗传多样性进行了分析,结果表明,总遗传变异中大约33.7%的遗传变异存在于材料间,66.3%的遗传变异存在于材料内。这种高水平的变异可为羽衣甘蓝的未来育种提供了参考。吴立萍等[45]利用SSR分子标记技术对54个俄罗斯马铃薯种质资源进行了分析,将其分为10个类群,试验从基因层面揭示了它们的亲缘关系。
本试验利用SSR标记对63份马铃薯种质资源进行遗传多样性分析,在GD值为0.180将其划分为5个类群。聚类结果与主成分分析结果高度一致,分类与种质资源的地理位置有关。本研究中大同里外黄、鄂马铃薯14号与晋薯14号被聚为一大类,而大同里外黄与晋薯14号被划为同一个亚类,鄂马铃薯14号被划为一个亚类。毛向红等[46]对61份马铃薯种质资源进行遗传多样性分析,结果显示鄂马铃薯14号与晋薯14号聚为一类,大同里外黄聚为一类,本研究结果与其存在差异。安苗等[47]对52个马铃薯品种(系)聚类结果表明,大同里外黄和民丰红聚为一个亚类,冀张薯8号、京张薯1号和京张薯3号聚为一个亚类,这一结果与本研究相同,将晋薯16单独划分一类,陇薯7号与青薯9号划分在2个亚类,本研究与其不一致。张云帅[48]对24个马铃薯品种的遗传多样性研究表明青薯9号、陇薯7号、冀张薯8号、中薯5号、北方001和北方002聚为一大类,Favorita、大西洋、冀张薯12号和希森6号聚为一大类,而本研究中上述品种划为一大类,其中青薯9号和陇薯7号划为一个亚类,其余品种划为一个亚类。
本文中56份国内马铃薯种质资源聚为一个主要类群,在GD值为0.13时类群Ⅴ又划分为3个亚类群。包含42个从黑龙江省农业科学院克山分院引种的北方马铃薯资源、6个南方马铃薯资源、8个YS资源,表明它们亲缘关系较近,遗传背景相似;本研究结果与段绍光等[13]的结果一致。因此,仍需加大野生种质资源或国外种质资源的引种力度,以改善国内马铃薯种质资源遗传背景狭窄的现状,为培育优良品种提供丰富的种质材料。同时建立了63份马铃薯种质资源的指纹图谱和二维码DNA分子身份证,以避免后期出现同名异义现象。本研究的分类仅采用了分子标记技术方法,未来的研究应结合农艺性状调查和生物形态统计分析方法,更全面地分析马铃薯品种的遗传多样性,这样可以更准确、快速地为杂交育种提供详细的亲本信息,加快育种进程,研究结果对马铃薯新品种的选育具有指导意义。
4 结论
利用从132对SSR引物中筛选出多态性高的32对引物,分析63份马铃薯种质资源的遗传差异,结果表明,63份马铃薯种质材料的平均多态性信息含量为0.8144,平均有效等位基因数为1.7139,平均Nei’s遗传多样性指数为0.3927,平均Shannon指数为0.5632;遗传距离介于0.12~0.71,平均遗传距离为0.2969;SSR聚类分析和主成分分析将63份马铃薯种质资源可分为5个类群,其划分结果与地域关联较大,建立了63份马铃薯种质资源的SSR指纹图谱和QR编码信息。
参考文献
6个彩色马铃薯品种的ISSR分析
DOI:10.7668/hbnxb.2010.05.024
[本文引用: 2]

为了解彩色马铃薯黑美人、拉塞特-01、底西芮、青薯168、水洗红、涩皮红6个品种在DNA分子水平上的遗传差异性,对彩色马铃薯种质鉴定及杂交选育提供依据,试验利用ISSR分子标记技术对其多态性进行了分析.结果表明:12个ISSR适宜引物共扩增出151个条带,其DNA片段长度多为250~2 000 bp,其中多态性条带136个,多态性条带百分率为88.2%.6个彩色马铃薯品种间的遗传距离(GD)为0.38 8~0.6,平均为0.539.以GD值0.55为基准,6个材料聚为3类:第一类为底西芮、青薯168和涩皮红,第二类为拉塞特-01和黑美人,水洗红单独聚为一类,6个彩色马铃薯品种间的亲缘关系存在一定的差异.
马铃薯遗传育种研究:现状与展望
DOI:10.3864/j.issn.0578-1752.2017.06.003
[本文引用: 1]

马铃薯是世界第三大粮食作物,马铃薯产业的可持续发展对保障世界和中国的粮食安全具有重要意义。优良品种是支撑马铃薯产业发展的基础。马铃薯经常遭受病虫害的侵袭和非生物胁迫,加工业的迅速发展和人们对食物营养的重视,迫切需要选育出更抗病、更耐逆、更高产、更优质和专用的马铃薯新品种。培育一个优良马铃薯品种,种质资源是基础,重要性状的遗传学是理论指导,先进的育种技术是保障,完善的推广和栽培模式是支撑。世界范围内,保存了大约65 000份马铃薯种质资源,通过对种质资源抗病、抗逆和品质方面的系统评价,并应用多种资源利用技术,将三大类约17个野生种的种质导入到普通栽培种中,应用于育种和遗传学研究。利用纯合双单倍体材料作为测序对象,马铃薯基因组序列已经被揭示,预测出了39 031个蛋白编码基因,目前更多的种质资源正在被重测序以揭示更多的等位变异。马铃薯普通栽培品种是无性繁殖四倍体作物,具有四体遗传特性,尽管如此,许多植株发育和形态、块茎品质和抗病抗逆等重要性状的遗传特性基本明确,并定位和克隆了大量重要性状相关基因。目前,马铃薯育种技术主要涵盖传统育种技术、倍性育种技术、标记辅助选择育种技术、基因工程育种技术和新兴的基因组选择育种技术。中国马铃薯遗传育种研究队伍不断壮大,品种选育取得了重大进展。荷兰马铃薯遗传育种水平居于世界前列,合作育种模式推动了商业化育种。不断完善马铃薯综合育种技术,创新育种模式和机制,充分利用现有种质资源培育突破性、专用型品种将是未来马铃薯遗传育种发展的主要方向。
甘肃省主栽马铃薯品种遗传多样性的AFLP与SSR分子标记分析
SSR analysis of genetic diversity among 192 diploid potato cultivars
Evaluation of genetic diversity among Russet potato clones and varieties from breeding programs across the United States
Molecular analysis of potatoes from the pacific northwest tri-state variety development program and selection of markers for practical DNA fingerprinting applications
Genetic diversity and population structure of advanced clones selected over forty years by a potato breeding program in the USA
DOI:10.1038/s41598-021-87284-x
PMID:33863959
[本文引用: 1]

Knowledge regarding genetic diversity and population structure of breeding materials is essential for crop improvement. The Texas A&M University Potato Breeding Program has a collection of advanced clones selected and maintained in-vitro over a 40-year period. Little is known about its genetic makeup and usefulness for the current breeding program. In this study, 214 potato clones were genotyped with the Infinium Illumina 22 K V3 Potato Array. After filtering, a total of 10,106 single nucleotide polymorphic (SNP) markers were used for analysis. Heterozygosity varied by SNP, with an overall average of 0.59. Three groups of tetraploid clones primarily based on potato market classes, were detected using STRUCTURE software and confirmed by discriminant analysis of principal components. The highest coefficient of differentiation observed between the groups was 0.14. Signatures of selection were uncovered in genes controlling potato flesh and skin color, length of plant cycle and tuberization, and carbohydrate metabolism. A core set of 43 clones was obtained using Core Hunter 3 to develop a sub-collection that retains similar genetic diversity as the whole population, minimize redundancies, and facilitates long-term conservation of genetic resources. The comprehensive molecular characterization of our breeding clone bank collection contributes to understanding the genetic diversity of existing potato resources. This analysis could be applied to other breeding programs and assist in the selection of parents, fingerprinting, protection, and management of the breeding collections.
Development of a potato cultivar core collection, a valuable tool to prospect genetic variation for novel traits
Genetic diversity and core collection for potato cultivars from cameroon as revealed by SSR markers
Analysis of the wild potato germplasm of the series Acaulia with AFLPs: implications for ex situ conservation
The wild potato germplasm of the series Acaulia maintained at the Centre for Genetic Resources, The Netherlands, currently consists of 314 accessions. This collection comprises seed samples of the species Solanum acaule (ssp. acaule, ssp. aemulans, ssp. palmirense and ssp. punae) and Solanum albicans collected from South America. In order to validate taxonomic classification, to investigate the extent of redundancy and to study the distribution of genetic diversity across the collection area, the entire collection was analysed with two AFLP primer pairs on two plants per accession. Within the entire sample a total number of 130 polymorphic bands were scored for the two primer pairs. An UPGMA cluster analysis grouped the majority of plants according to the species and subspecies. A total number of 16 misclassifications were identified, including four cases that did not seem to belong to the series Acaulia. Two accessions were found to consist of plants of different AFLP clusters. AFLP data also allowed the taxonomic classification of the subspecies of 97 accessions that previously were described as S. acaule only. For 126 accessions the two individuals studied displayed identical AFLP profiles. Forty six of these 126 accessions shared their profiles with both or single plants of other accessions. These were all tested for identical profiles for a third primer pair, resulting in 15 duplication groups consisting of a total number of 22 accessions and 14 single plants. Analyses of molecular variance (AMOVA) were performed to examine the distribution of genetic variation. Comparison of geographic distances between the collection site of plants and the number of AFLP polymorphisms revealed no consistent relationship between geographic distance and genetic diversity. AFLP analysis appeared to be an efficient method to verify taxonomic classification and to identify redundancies in the wild germplasm of the series Acaulia. Implications of the results for the ex situ conservation of wild potato germplasm are discussed.
Potato cultivar identification using simple sequence repeats markers (SSR)
Genetic relationships among local potato cultivars from Spain using SSR markers
Multiplex PCR system optimization with potato SSR markers
Assessment of genetic diversity incultivated radishes (Raphanus sativus) by agronomic traits and SSR markers
Genetic diversity between and within the natural populations of Garcinia indica (Thouars) Choisy: a high value medicinal plant from Northern Western Ghats of India using ISSR markers
Microsatellite markers: what they mean and why they are so useful
DOI:10.1590/1678-4685-GMB-2016-0027
PMID:27561112
[本文引用: 1]

Microsatellites or Single Sequence Repeats (SSRs) are extensively employed in plant genetics studies, using both low and high throughput genotyping approaches. Motivated by the importance of these sequences over the last decades this review aims to address some theoretical aspects of SSRs, including definition, characterization and biological function. The methodologies for the development of SSR loci, genotyping and their applications as molecular markers are also reviewed. Finally, two data surveys are presented. The first was conducted using the main database of Web of Science, prospecting for articles published over the period from 2010 to 2015, resulting in approximately 930 records. The second survey was focused on papers that aimed at SSR marker development, published in the American Journal of Botany's Primer Notes and Protocols in Plant Sciences (over 2013 up to 2015), resulting in a total of 87 publications. This scenario confirms the current relevance of SSRs and indicates their continuous utilization in plant science.
基于高基元SSR构建黍稷种质资源的分子身份证
DOI:10.3724/SP.J.1006.2022.14034
[本文引用: 1]

为快速鉴定黍稷(Panicum miliaceum)资源, 建立大数据管理平台, 为种质身份标识和溯源管理提供理论依据。本研究以来源于4个生态栽培区的130份资源为材料, 基于35个高基元SSR (四、五和六碱基重复各21、10和4个)构建分子身份证。结果表明, 35个标记中有30个扩增条带稳定, 可用于分子身份证构建。30个标记共检出等位变异(Na) 90个, 平均为30个; 有效等位变异(Ne)为2.3186~2.9982, 平均为2.7607; Shannon多样性指数(I)为0.9158~1.0873, 平均为1.0472; Nei’s基因多样性指数(Nei)为0.5687~0.6665, 平均为0.6360; 多态性信息含量(PIC)为0.5151~0.7898, 平均为0.6966; 观测杂合度(Ho)为0.5000~ -0.8678, 平均为0.7168; 期望观测杂合度(He)为0.5710~0.6691, 平均为0.6386。基于UPGMA聚类将材料划为3个类群(I、II、III), 就山西省材料而言, 地方品种和育成品种分别划归类群I和III, 农家种在3个类群中均有分布。主成分分析将试材归为4类, 聚类结果与其地理来源一致。基于最少标记区分最多种质的原则, 剔除相似系数高的3个标记(RYW23、RYW49和RYW51), 筛选其余27个标记, 发现仅用17个标记组合(RYW35、RYW40、RYW37、RYW18、RYW30、RYW16、RYW20、RYW19、RYW8、RYW5、RYW3、RYW7、RYW1、RYW14、RYW9、RYW6和RYW10)可将全部材料区分。用ID Analysis 4.0、在线条形码生成器和二维码技术(http://barcode.cnaidc.com/app/html/bcgcode128.php和 https://cli.im/)构建了130份资源的字符串、条形码和二维码DNA分子身份证。
Comparison of SSR and SNP markers in estimation of genetic diversity and population structure of Indian rice varieties
Genetic diversity and structure of landrace accessions, elite lineages and cultivars of common bean estimated with SSR and SNP markers
Population structure and genetic diversity characterization of a sunflower association mapping population using SSR and SNP markers
Population structure,genetic diversity and linkage disequilibrium in elite winter wheat assessed with SNP and SSR markers
DOI:10.1007/s00122-013-2065-1
PMID:23429904
[本文引用: 1]

Modern genomics approaches rely on the availability of high-throughput and high-density genotyping platforms. A major breakthrough in wheat genotyping was the development of an SNP array. In this study, we used a diverse panel of 172 elite European winter wheat lines to evaluate the utility of the SNP array for genomic analyses in wheat germplasm derived from breeding programs. We investigated population structure and genetic relatedness and found that the results obtained with SNP and SSR markers differ. This suggests that additional research is required to determine the optimum approach for the investigation of population structure and kinship. Our analysis of linkage disequilibrium (LD) showed that LD decays within approximately 5-10 cM. Moreover, we found that LD is variable along chromosomes. Our results suggest that the number of SNPs needs to be increased further to obtain a higher coverage of the chromosomes. Taken together, SNPs can be a valuable tool for genomics approaches and for a knowledge-based improvement of wheat.
Genetic diversity and population structure analysis in a large collection of Vicia amoena in China with newly developed SSR markers
Development of genome-wide SSR markers from Angelica gigas Nakai using next generation sequencing
Development of 44 novel polymorphic SSR markers for determination of shiitake mushroom (Lentinula edodes) cultivars
Genome survey and development of 18 microsatellite markers to assess genetic diversity in Spondias tuberosa Arruda Câmara (Anacardiaceae) and cross-amplification in congeneric species
DOI:10.1007/s11033-019-04768-w
PMID:30915689
[本文引用: 1]

Spondias tuberosa Arr. Cam belongs to the Anacardiaceae family, an economically important family of plants whose fruits are consumed by humans and animals. The aim of this study was to develop microsatellite markers using sequences from high-throughput sequencing and a magnetic bead enrichment method. The sequences were used to obtain contigs with a minimum of 500 nucleotides using Ray software and the mining of the simple sequence repeats (SSR) was performed with Phobos software, while the primers were designed by Primer3. We developed 18 polymorphic nuclear microsatellite markers and successfully cross-amplified them to three Spondias species. In S. tuberosa, the alleles ranged from 2 to 5 for each locus and Hardy-Weinberg equilibrium was found for 16 loci, with an expected and observed heterozygosity at 0.095-0.755 and 0.1-0.75, respectively. Cross-transferability was obtained for all loci in S. bahiensis, S. dulcis and S. purpurea. We concluded that the microsatellite markers developed in this study are useful in genetic population and conservation studies, as well as for investigating the hybrid origins of Spondias species.
Analysis of genetic diversity and population structure using SSR markers and validation of a Cleavage Amplified Polymorphic Sequences (CAPS) marker involving the sodium transporter OsHKT1;5 in saline tolerant rice (Oryza sativa L. ) landraces
Genetic diversity and population structure assessed by SSR and SNP markers in a large germplasm collection of grape
Genetic diversity and population structure revealed by SSR markers on endemic species osmanthusserrulatus rehder from southwestern Sichuan basin, China
Genetic diversity, analysis of some agro-morphological and quality traits and utilization of plant resources of Alfalfa
AFLP analysis of genetic diversity and phylogenetic relationships of Brassica oleracea in Ireland
52个马铃薯遗传多样性分析及SSR分子身份证构建
DOI:10.13560/j.cnki.biotech.bull.1985.2023-0512
[本文引用: 1]

马铃薯(Solanum tuberosum L.)种质资源遗传多样性评价是开展遗传育种的前提和基础,为探究马铃薯种质资源的遗传背景,进而为新品种的选育提供育种材料。以52个马铃薯品种(系)为研究材料,采用SSR标记分析亲缘关系,进而评价其遗传多样性,并构建指纹图谱。从25对SSR引物中筛选出多态性丰富、稳定性好的16对引物,并对52份马铃薯品种(系)进行SSR扩增分析,16对SSR引物扩增出147个多态性条带,引物平均多态条带比率为64.71%,平均Nei's遗传多样性(Nei's genetic diversity, H)和平均Shannon's指数(Shannon index, I)分别为0.14和0.24。在遗传相似系数0.23处,52份材料经聚类分析分为3大类,类群I包括49个品种(系),46个是北方品种(系),3个国外引进品种(系),均适宜晋北地区种植;类群II包括2个品种,‘北方016’和‘晋薯16号’,特点是薯皮黄色、干物质含量高;类群III仅包括‘晋薯15号’,在大类的划分上未按照地理来源进行聚类。在遗传相似系数0.36处,类群I又可以分为7个亚类。基于最少引物鉴定最多种质的原则,利用C59、S25、C33、S151共4对引物组合可区分全部供试品种,构建出基于SSR标记的数字化指纹图谱及分子身份证。利用SSR标记技术多态性引物可以评价马铃薯种质资源的遗传多样性,通过分子身份证能够准确鉴别以及溯源不同马铃薯品种(系)。