作物杂志,2024, 第1期: 65–72 doi: 10.16035/j.issn.1001-7283.2024.01.009
所属专题: 小麦专题
小麦bHLH家族转录因子的鉴定及其在盐胁迫条件下的表达分析
吕宝莲1,2( ), 杨宇昕2, 崔立操2, 史峰3, 马亮3, 孔秀英2, 张立超2(
), 杨宇昕2, 崔立操2, 史峰3, 马亮3, 孔秀英2, 张立超2( ), 倪志勇1(
), 倪志勇1( )
)
- 1新疆农业大学生命科学学院,830052,新疆乌鲁木齐
2中国农业科学院作物科学研究所,100081,北京
3石家庄市农林科学研究院,050041,河北石家庄
Identification of bHLH Family Transcription Factors of Wheat and Expression Analysis under Salt Stress
Lü Baolian1,2( ), Yang Yuxin2, Cui Licao2, Shi Feng3, Ma Liang3, Kong Xiuying2, Zhang Lichao2(
), Yang Yuxin2, Cui Licao2, Shi Feng3, Ma Liang3, Kong Xiuying2, Zhang Lichao2( ), Ni Zhiyong1(
), Ni Zhiyong1( )
)
- 1College of Life Sciences, Xinjiang Agricultural University, Urumqi 830052, Xinjiang, China
2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China
3Shijiazhuang Academy of Agricultural and Forestry Sciences, Shijiazhuang 050041, Hebei, China
摘要:
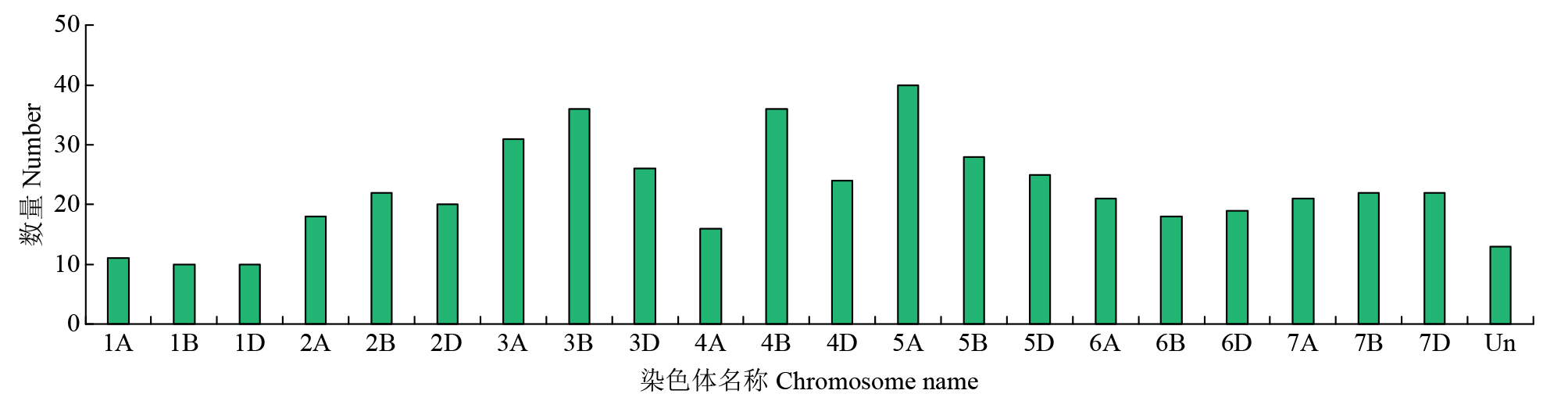
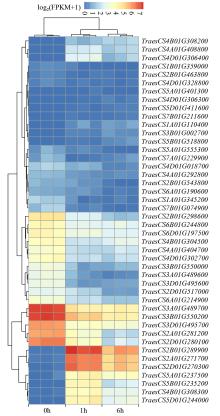
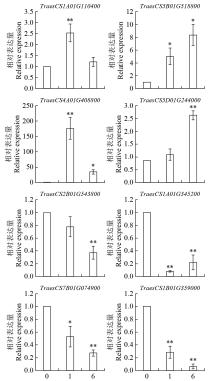
碱性螺旋―环―螺旋(basic helix-loop-helix,bHLH)转录因子作为植物中第二大类转录因子家族,在植物生长发育、信号传导、生物合成以及响应非生物胁迫等方面发挥重要功能。为了探讨小麦bHLH转录因子在盐胁迫应答中的功能,以冬性小麦科农199为试验材料,通过转录组测序和生物信息学分析的方法对小麦bHLH转录因子在盐胁迫下的表达特性进行系统分析。结果表明,小麦中共有489个bHLH转录因子,分布在21条染色体上;构建系统进化树将bHLH家族进一步划分为9个亚族,其中VI亚族成员最多,IV亚族成员最少;盐胁迫下小麦根转录组测序结果表明,在盐胁迫处理后1和6h差异表达基因(DEG)共有44个,其中上调表达的基因有17个,下调表达的有27个;GO分析表明,DEG富集在水、盐胁迫、生长素调节等方面;KEGG注释分析表明,DEGs主要富集在淀粉和蔗糖代谢途径以及氨基糖和核苷酸糖代谢等信号通路;对部分DEG进行qRT-PCR验证,结果表明DEG的表达模式与转录组测序结果一致,证明了RNA-Seq结果的准确性。
| [1] |
Zhang H M, Zhu J H, Gong Z Z, et al. Abiotic stress responses in plants. Nature Reviews Genetics, 2022, 23(2):104-119.
doi: 10.1038/s41576-021-00413-0 |
| [2] |
Bhanbhro N, Xiao B B, Han L, et al. Adaptive strategy of allohexaploid wheat to long-term salinity stress. BMC Plant Biology, 2020, 20(1):210.
doi: 10.1186/s12870-020-02423-2 pmid: 32397960 |
| [3] |
马彦军, 段慧荣, 魏佳, 等. NaCl胁迫下黑果枸杞转录组测序分析. 生物技术通报, 2020, 36(2):100-109.
doi: 10.13560/j.cnki.biotech.bull.1985.2019-0492 |
| [4] | 朱涛, 李芳菲, 杨海涵, 等. 山药bHLH基因家族鉴定及表达分析. 信阳师范学院学报(自然科学版), 2022, 35(3):393-399. |
| [5] |
Hao Y Q, Zong X M, Ren P, et al. Basic helix-loop-helix (bHLH) transcription factors regulate a wide range of functions in Arabidopsis. International Journal of Molecular Sciences, 2021, 22(13):7152.
doi: 10.3390/ijms22137152 |
| [6] | Wang Y G, Li H Y, University H, et al. Sugar beet BvBHLH 92 tissue expression and subcellular localization. Journal of Engineering of Heilongjiang University, 2017, 8(3):45-49,2. |
| [7] | Lee H Y, Seo J S, Um T Y.OsbHLH148 confers drought tolerance in Arabidopsis. International Plant and Animal Genome Conference, January 14-18, 2012. California: San Diego, 2012. |
| [8] | 孙玉合, 孙晋浩, 牛文利, 等. 烟草NtbHLH112基因的克隆、鉴定及表达模式分析. 中国烟草科学, 2020, 41(5):8-14. |
| [9] |
悦曼芳, 张春, 郑登俞, 等. 玉米转录因子ZmbHLH91对非生物逆境胁迫的应答. 作物学报, 2022, 48(12):3004-3017.
doi: 10.3724/SP.J.1006.2022.13060 |
| [10] |
Zhai Y Q, Zhang L C, Xia C, et al. The wheat transcription factor, TabHLH39, improves tolerance to multiple abiotic stressors in transgenic plants. Biochemical and Biophysical Research Communications, 2016, 473(4):1321-1327.
doi: S0006-291X(16)30577-0 pmid: 27091431 |
| [11] |
Wang F B, Zhu H, Kong W L, et al. The Antirrhinum AmDEL gene enhances flavonoids accumulation and salt and drought tolerance in transgenic Arabidopsis. Planta, 2016, 244(1):59-73.
doi: 10.1007/s00425-016-2489-3 |
| [12] |
Sun X, Wang Y, Sui N. Transcriptional regulation of bHLH during plant response to stress. Biochemical and Biophysical Research Communications, 2018, 503(2):397-401.
doi: S0006-291X(18)31625-5 pmid: 30057319 |
| [13] | 毕晨曦, 杨宇昕, 于月华, 等. 小麦bZIP家族转录因子的鉴定及其在盐胁迫条件下的表达分析. 分子植物育种, 2021, 19 (15):4887-4895. |
| [14] |
Gabriela T, Enamul H, Peter H Q. The Arabidopsis basic/ helix-loop-helix transcription factor family. Plant Cell, 2003, 15:1749-1770.
doi: 10.1105/tpc.013839 |
| [15] |
Lorenzo C P, Anahit G, Irma R V, et al. Genome-wide classification and evolutionary analysis of the bHLH family of transcription factors in Arabidopsis, poplar, rice, moss, and algae. Plant Physiology, 2010, 153(3):1398-1412.
doi: 10.1104/pp.110.153593 |
| [16] | 赵小波, 闫彩霞, 李春娟, 等. 花生转录因子AhbHLH18克隆与功能分析. 花生学报, 2022, 51(2):1-8. |
| [17] | 施田野, 顾宇蓝, 张磊, 等. 粗山羊草响应盐胁迫转录组分析. 分子植物育种, 2020, 18(21):7015-7022. |
| [18] | 田烨, 王爽, 路正禹, 等. 甜菜应答盐胁迫诱导表达bHLH基因的鉴定与分析. 黑龙江大学自然科学学报, 2020, 37(6):712-717. |
| [19] | 徐秀荣, 杨克彬, 王思宁, 等. 毛竹bHLH转录因子的鉴定及其在干旱和盐胁迫条件下的表达分析. 植物科学学报, 2019, 37(5):610-620. |
| [20] | 唐文武, 吴秀兰, 钟佩桥, 等. 白菜bHLH转录因子家族的全基因鉴定及表达特征分析. 江西农业学报, 2020, 32(6):1-5. |
| [21] | 黄小芳, 毕楚韵, 王和寿, 等. 甘薯基因组bHLH转录因子鉴定与逆境胁迫表达分析. 福建农林大学学报(自然科学版), 2021, 50(4):440-450. |
| [22] | 孙颖琦, 孟亚轩, 赵心月, 等. 谷子bHLH转录因子家族基因鉴定及生物信息学分析. 种子, 2021, 40(12):45-55. |
| [23] |
何洁, 顾秀容, 魏春华, 等. 西瓜bHLH转录因子家族基因的鉴定及其在非生物胁迫下的表达分析. 园艺学报, 2016, 43(2):281-294.
doi: 10.16420/j.issn.0513-353x.2015-0886 |
| [1] | 刘红杰, 任德超, 葛君, 张素瑜, 吕国华, 何勋. 积温和播种密度对小麦越冬前生长状况的影响[J]. 作物杂志, 2024, (1): 141–147 |
| [2] | 刘哲文, 郭丹丹, 常旭虹, 王德梅, 杨玉双, 刘希伟, 王玉娇, 石书兵, 王艳杰, 赵广才. 氮肥追施时期和比例对强筋小麦籽粒灌浆及其生理机制的影响[J]. 作物杂志, 2024, (1): 174–179 |
| [3] | 郝小聪, 李欣宇, 侯起岭, 杨吉芳, 安春会, 王长华, 叶志杰, 张风廷. 施氮量对二系杂交小麦品质的影响[J]. 作物杂志, 2024, (1): 187–192 |
| [4] | 章蓉, 姜恩熙, 陈思, 余徐润, 陈刚, 冉莉萍, 熊飞. 乙烯利与1-甲基环丙烯调控小麦穗部籽粒形成的研究[J]. 作物杂志, 2023, (6): 101–107 |
| [5] | 刘哲文, 郭丹丹, 常旭虹, 王德梅, 王艳杰, 杨玉双, 刘希伟, 王玉娇, 石书兵, 赵广才. 强筋小麦花后氮素积累和转运对氮肥追施时期和比例的响应[J]. 作物杂志, 2023, (6): 114–120 |
| [6] | 刘希伟, 王德梅, 王艳杰, 杨玉双, 赵广才, 常旭虹. 小麦生育中后期干旱高温对籽粒产量形成过程的影响机制及缓解措施[J]. 作物杂志, 2023, (6): 17–25 |
| [7] | 陈丹, 熊芙蓉, 伍少云, 白晓东, 周国雁, 武晓阳, 蔡青. 云南省小麦地方品种抗条锈病基因检测与地理分布[J]. 作物杂志, 2023, (6): 41–46 |
| [8] | 王义凡, 任宁, 董向阳, 赵亚南, 叶优良, 汪洋, 黄玉芳. 控释尿素与普通尿素配施对小麦产量、氮素吸收及经济效益的影响[J]. 作物杂志, 2023, (5): 117–123 |
| [9] | 杨梅, 杨卫君, 高文翠, 贾永红, 张金汕. 生物质炭与氮肥配施对灌区冬小麦干物质转运、农艺性状及产量的影响[J]. 作物杂志, 2023, (5): 138–144 |
| [10] | 黄杰, 葛昌斌, 王君, 曹燕燕, 乔冀良, 廖平安, 宋丹阳, 卢雯瑩. 基于主成分回归模型的漯河市小麦相对气象千粒重的模拟模型[J]. 作物杂志, 2023, (5): 212–218 |
| [11] | 刘书含, 陈磊, 张建朝, 胡甘, 孙君艳, 刘东涛, 王军卫. TMS5基因在小麦BNS不育系育性转换中的差异表达分析[J]. 作物杂志, 2023, (5): 24–29 |
| [12] | 张东旭, 胡丹珠, 闫金龙, 冯丽云, 邬志远, 张俊灵, 李岩华. 不同茬口晚播小麦喷施链霉菌剂对产量及光合特性的影响[J]. 作物杂志, 2023, (5): 255–263 |
| [13] | 宋桂成, 余桂红, 张鹏, 马鸿翔. 不同小麦品种(系)拔节期耐渍性评价[J]. 作物杂志, 2023, (5): 30–36 |
| [14] | 葛昌斌, 秦素研, 乔冀良, 王君, 齐双丽, 卢雯瑩, 张振永. 2001-2021年豫南和江苏淮河以南审定小麦品种农艺、品质性状和病害演变对比分析[J]. 作物杂志, 2023, (5): 49–58 |
| [15] | 杨程, 张德奇, 杜思梦, 张丽佳, 靳海洋, 李滢, 邵运辉, 王汉芳, 方保停, 李向东, 刘美君. 黑暗和强光下脱水对小麦离体叶片光系统活性的影响[J]. 作物杂志, 2023, (5): 98–103 |
|
||