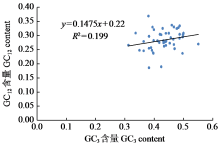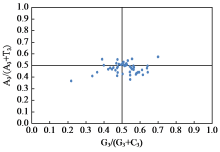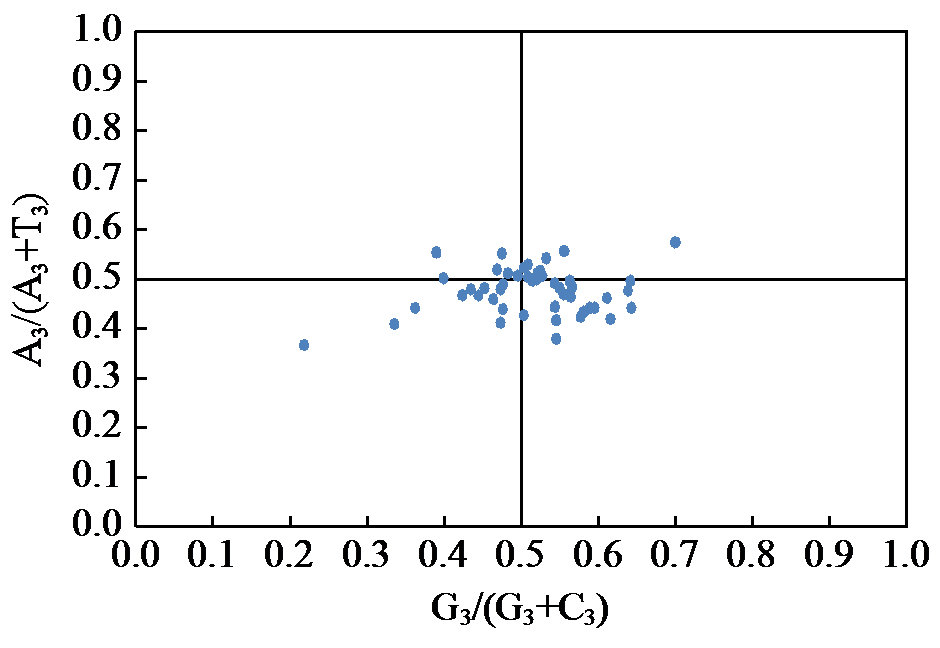作物杂志,2025, 第6期: 83–90 doi: 10.16035/j.issn.1001-7283.2025.06.011
抗列当食用向日葵杂交种经葵1408叶绿体基因组特征及密码子偏好性分析
马军1( ), 张祥雷2, 安呈洁2, 杨学文3, 张博琳2, 张武2(
), 张祥雷2, 安呈洁2, 杨学文3, 张博琳2, 张武2( ), 王鹏冬4(
), 王鹏冬4( )
)
- 1
黑龙江省农业科学院经济作物研究所, 150086, 黑龙江哈尔滨
2佳木斯大学理学院, 154007, 黑龙江佳木斯
3佳木斯大学生物与农业学院, 154007, 黑龙江佳木斯
4山西农业大学经济作物研究所 030031, 山西太原
Chloroplast Genome Characteristics and Codon Bias Analysis of Anti-Broomrape Confectionery Sunflower Hybrid Jingkui 1408
Ma Jun1( ), Zhang Xianglei2, An Chengjie2, Yang Xuewen3, Zhang Bolin2, Zhang Wu2(
), Zhang Xianglei2, An Chengjie2, Yang Xuewen3, Zhang Bolin2, Zhang Wu2( ), Wang Pengdong4(
), Wang Pengdong4( )
)
- 1
Institute of Industrial Crops ,Heilongjiang Academy of Agricultural Sciences Harbin 150086, Heilongjiang, China
2College of Science ,Jiamusi University Jiamusi 154007, Heilongjiang, China
3College of Biology and Agriculture ,Jiamusi University Jiamusi 154007, Heilongjiang, China
4Research Institute of Economic Crops ,Shanxi Agricultural University, Taiyuan 030031 Shanxi, China
摘要:
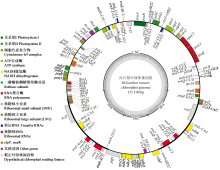
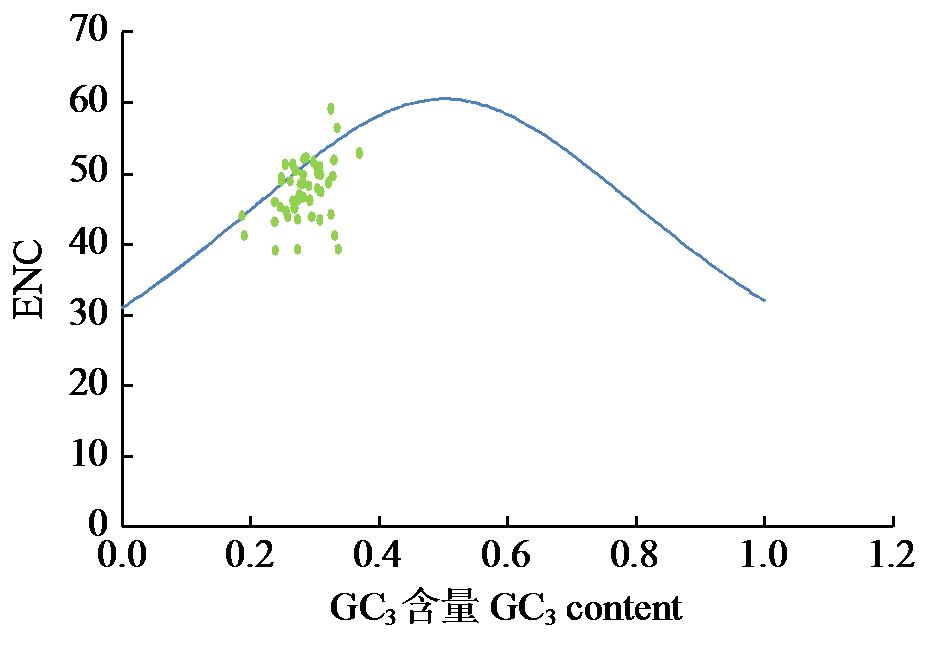
为探究抗列当食用向日葵杂交种经葵1408的叶绿体基因组特征及密码子偏好性,通过高通量测序技术获取其全基因组数据,对其叶绿体基因组进行组装和注释,筛选出50条编码序列(coding sequence,CDS)用于密码子偏好性分析。结果显示,经葵1408的叶绿体基因组总长151 100 bp,包含1对IR区(inverted repeat)、1个大单拷贝区(large single-copy region,LSC)和1个小单拷贝区(small single-copy region,SSC);共注释出129个基因,其中有85个基因编码蛋白质,8个基因编码rRNA,36个基因编码tRNA。50条CDS序列GC1、GC2、GC3和GCall平均GC含量分别为47.06%、39.29%、28.36%和38.24%,有效密码子数(ENC)平均值为47.62;中性绘图分析和PR2-plot偏倚分析表明密码子使用偏好主要受到自然选择影响;50条CDS共有20 244个密码子,最优密码子为CUU、CCA、ACU、GCU、UAU、CAU、AAA、GAU和GGA。
| [1] | 陈晓丹, 冯玉玺, 郭海洋, 等. 裸果木叶绿体基因组密码子使用偏性分析. 分子植物育种, 2023, 21(23):7715-7724. |
| [2] | 吴宪明, 吴松锋, 任大明, 等. 密码子偏性的分析方法及相关研究进展. 遗传, 2007, 29(4):420-426. |
| [3] | 赵耀, 刘汉梅, 顾勇, 等. 玉米waxy基因密码子偏好性分析. 玉米科学, 2008, 16(2):16-21. |
| [4] | 唐晓芬, 陈莉, 马玉韬. 密码子使用偏性量化方法研究综述. 基因组学与应用生物学, 2013, 32(5):660-666. |
| [5] | Sharp P M, Matassi G. Codon usage and genome volution. Current Opinion in Genetics & Development, 1994, 4(6):851-860. |
| [6] | Dierckxsens N, Mardulyn P, Smits G. NOVOPlasty: de novo assembly of organelle genomes from whole genome data. Nucleic Acids Research, 2017, 45(4):e18. |
| [7] | Tillich M, Lehwark P, Pellizzer T, et al. GeSeq-versatile and accurate annotation of organelle genomes. Nucleic Acids Research, 2017, 45(1):6-11. |
| [8] | 李冬梅, 吕复兵, 朱根发, 等. 文心兰叶绿体基因组密码子使用的相关分析. 广东农业科学, 2012, 39(10):61-65. |
| [9] | 王雪, 马军, 杨学文, 等. 栽培型向日葵叶绿体基因组密码子使用偏好性分析. 分子植物育种, 2024, 22(4):1096-1102. |
| [10] | 原晓龙, 李云琴, 王毅, 等. 西南桦叶绿体基因组密码子偏好性分析. 基因组学与应用生物学, 2020, 39(12):5758-5764. |
| [11] | 颜超, 徐钦, 李志, 等. 黎平瘤果茶的叶绿体基因组特征及系统发育分析. 分子植物育种,(2024-02-29)[2024-07-26]. https://link.cnki.net/urlid/46.1068.S.20240228.1659.008. |
| [12] |
杨国锋, 苏昆龙, 赵怡然, 等. 蒺藜苜蓿叶绿体密码子偏好性分析. 草业学报, 2015, 24(12):171-179.
doi: 10.11686/cyxb2015016 |
| [13] | 张文达, 赵逸伦, 王湘雨, 等. 再力花叶绿体基因组特征和系统发育分析. 分子植物育种,(2024-04-03)[2024-07-26]. https://link.cnki.net/urlid/46.1068.s.20240403.0936.006. |
| [14] | 崔雨丝, 韩骐泽, 张诗文, 等. 绣球叶绿体基因组特征及密码子偏好性分析. 分子植物育种,(2024-03-29)[2024-07-26]. https://link.cnki.net/urlid/46.1068.S.20240329.0938.007. |
| [15] | 李志芳, 陈丽玲, 罗淑洁, 等. 西南牡蒿叶绿体基因组特征及系统发育分析. 广西植物, 2024, 44(9):1732-1745. |
| [16] |
Hunt R C, Simhadri V L, Iandoli M, et al. Exposing synonymous mutations. Trends Genet, 2014, 30(7):308-321.
doi: 10.1016/j.tig.2014.04.006 pmid: 24954581 |
| [17] | 马楠, 叶奕含, 邹晨鑫, 等. 人参叶绿体全基因组特征及密码子偏好性分析. 分子植物育种,(2022-11-30)[2024-07-26]. https://link.cnki.net/urlid/46.1068.S.20221129.1638.028. |
| [18] |
余志伟, 陈垣, 郭凤霞, 等. 菘蓝叶绿体基因组密码子使用偏好性分析. 草地学报, 2024, 32(8):2374-2385.
doi: 10.11733/j.issn.1007-0435.2024.08.005 |
| [19] | 代国娜, 尚明越, 王嘉乐, 等. 百合属药用植物叶绿体基因组密码子偏好性及系统发育研究. 中草药, 2024, 55(11):3835-3844. |
| [20] | 黄思琦, 张麒功, 叶泽霖, 等. 5种柏科植物叶绿体基因组密码子偏好性分析. 福建农林大学学报, 2024, 53(2):214-220. |
| [21] | 魏亚楠, 龚明贵, 白娜, 等. 梁山慈竹叶绿体基因组密码子偏好性分析. 浙江农林大学学报, 2024, 41(4):696-705. |
| [22] | 张智, 张国帅, 张新可, 等. 硬尖神香草和欧神香草叶绿体基因组密码子偏好性分析. 分子植物育种,(2024-04-22)[2024-07-26]. https://link.cnki.net/urlid/46.1068.S.20240420.1355.002. |
| [23] | 谢刘义, 任璇, 应文博, 等. 栽培大麦和野生大麦叶绿体基因组密码子偏好性比较分析. 云南民族大学学报, 2024, 34(3):268-276. |
| [1] | 周菲, 黄绪堂, 谢鹏远, 王静, 刘岩, 崔佳伟, 唐立郦, 王文军. TRV介导的向日葵VIGS体系的构建[J]. 作物杂志, 2025, (6): 45–50 |
| [2] | 王贺亚, 罗静静, 孟玲, 艾海峰, 王斌, 李怀胜, 徐靖鹏, 徐向阳. 塔额盆地食用向日葵品种产量敏感性分析[J]. 作物杂志, 2025, (3): 30–37 |
| [3] | 邸娜, 郑喜清, 王靖, 韩海军, 李娜. 向日葵应对列当寄生的生理响应差异性研究[J]. 作物杂志, 2025, (2): 123–127 |
| [4] | 张之为, 孙学桃, 解国华, 王斐羽, 张宇宽, 杜磊, 赵君, 张键. 不同浓度甲氧咪草烟处理对向日葵列当寄生的影响[J]. 作物杂志, 2024, (6): 226–231 |
| [5] | 刘春晖, 杜瑞霞, 王永行, 杨钦方, 单飞彪, 胥福勋, 赵宇新, 陈舒舒, 刘伟. 气象因子与向日葵DUS测试测量型性状的关系[J]. 作物杂志, 2024, (3): 156–162 |
| [6] | 杜超, 李军, 王刚, 邬雪瑞, 任志远, 张俊峰, 包海柱, 温埃清. 河套灌区中度盐碱地高垄覆膜滴灌对向日葵生长及水分利用的影响[J]. 作物杂志, 2024, (1): 111–116 |
| [7] | 吕增帅, 董红业, 王鹏, 段维, 刘胜利, 柳延涛. 向日葵除草剂抗性机制及育种研究应用进展[J]. 作物杂志, 2024, (1): 16–22 |
| [8] | 吴胜, 段玉, 张婷婷, 安昊, 张君, 梁俊梅, 张胜. 食葵干物质积累、转运和产量的关系以及对水氮互作的响应[J]. 作物杂志, 2023, (6): 243–251 |
| [9] | 李鹤鹏, 张匀华, 孟庆林, 马立功, 于洪涛, 李海燕, 李易初, 刘佳, 石凤梅, 杨帆, 刘亮. 向日葵盘腐型菌核病诱抗剂筛选及超敏蛋白应用技术研究[J]. 作物杂志, 2023, (6): 257–260 |
| [10] | 凌一波, 王斌杰, 胡一民, 海那尔·毛地热合曼, 陈年来. 向日葵干物质转运及产量形成对密度与行距的响应[J]. 作物杂志, 2023, (5): 197–203 |
| [11] | 依兵, 刘金刚, 宋殿秀, 王德兴, 赵明珠, 刘晓宏, 孙恩玉, 崔良基. 干旱地区向日葵与谷子间作的土地生产力及种间竞争力的研究[J]. 作物杂志, 2023, (5): 219–223 |
| [12] | 朱孔艳, 韩升才, 赵榕, 温玉洁, 胡昊驰, 乔益民, 卢佳锋, 曹凯, 许政晗, 包海柱, 高聚林. 向日葵籽粒拮抗核盘菌的内生菌分离筛选及鉴定[J]. 作物杂志, 2023, (5): 280–284 |
| [13] | 贾秀苹, 卯旭辉, 梁根生, 刘润萍, 刘风, 王兴珍. 向日葵抗盐碱生理生化机制与生长发育特性分析[J]. 作物杂志, 2022, (5): 146–152 |
| [14] | 刘继霞, 山军建, 王平. 不同花色观赏向日葵杂交后代主要观赏性状遗传规律研究[J]. 作物杂志, 2022, (5): 56–61 |
| [15] | 周菲. 向日葵HaLACS7基因的生物信息学和表达分析[J]. 作物杂志, 2022, (3): 104–108 |
|
||