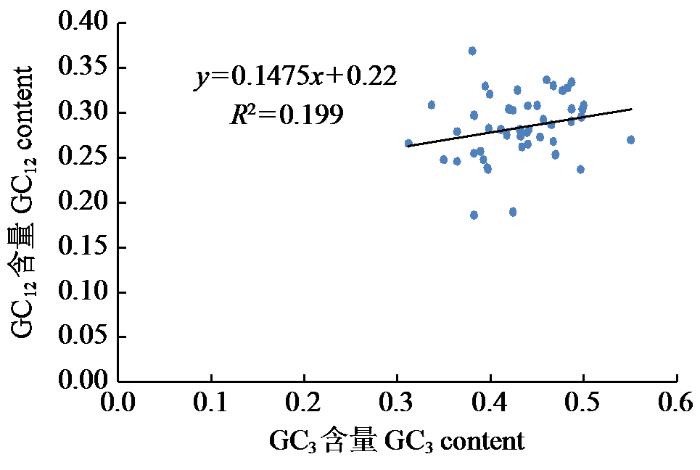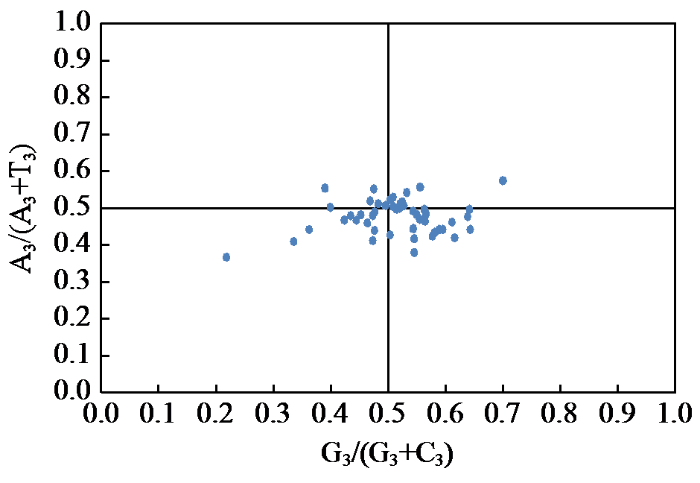叶绿体是植物特有的细胞器,在植物细胞中扮演能量转换的角色,并参与多个重要的生命活动[1]。叶绿体基因组结构简单,长度一般在107~218 kb,包含大量的遗传信息,叶绿体具有独立于细胞核的基因组,与核基因组相比,叶绿体基因组具有拷贝数高、进化速率适中且全基因组序列更容易获取等特点。随着测序技术的发展,叶绿体基因组研究迅速,叶绿体全基因组学成为物种鉴定分类和基因组进化分析的重要工具。目前,在NCBI数据库上收集的向日葵属叶绿体完整基因组仅有60个,亟需补充更多的向日葵属叶绿体基因组数据,从而理清菊科植物系统发育进化过程,为菊科植物向日葵品种的定位、分类、开发与改良奠定基础。
向日葵作为一种重要的特色油料作物,有着非常显著的经济价值。向日葵原产于美洲,发展于欧洲,而后传入亚洲。我国向日葵种质资源匮乏,大多数种质资源存在遗传背景狭窄和谱系溯源不清等问题。在已登记的向日葵品种中“一品多名”现象尤为突出,严重制约我国向日葵种业的创新与发展。作物种质资源是作物新品种选育的基础材料,生产推广的栽培品种是向日葵种质资源来源的重要途径之一。抗列当食用向日葵杂交种经葵1408是食用向日葵杂交种[登记编号GPD向日葵(2021)140009],具有品种权保护,授权号为CNA20211008289,该品种作为山西省农业农村厅优选出的适宜农业生产的主推品种,具有抗列当和抗菌核病的优良特性。本研究对食用向日葵杂交种经葵1408叶绿体全基因组数据(GenBank登录号:PQ009581)进行分析,在叶绿体基因组水平上为人们利用优质向日葵种质资源创新改良食用向日葵品种提供数据支持。
1 材料与方法
1.1 试验材料
1.2 密码子相关参数与绘图分析
2 结果与分析
2.1 JK1408叶绿体基因组结构与特征
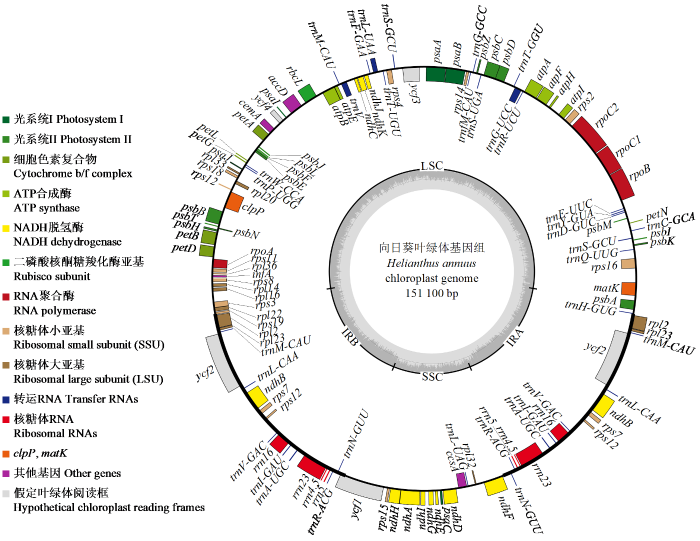
JK1408的叶绿体基因组呈现典型的四分体结构,全长151 100 bp,LSC长度为83 531 bp,占全长55.3%;SSC长度为18 319 bp,占全长12.1%;IR的长度均为24 625 bp,共占全长32.6%(图1)。
图1
2.2 叶绿体基因组注释
JK1408叶绿体基因组共注释出129个基因,其中编码蛋白质基因85个,编码rRNA基因8个,编码tRNA基因36个。蛋白质编码基因中,复制相关基因29个,光合作用相关基因46个,其他功能基因6个,未知功能基因4个。LSC区共有81个基因,其中编码蛋白基因60个,编码tRNA基因21个。SSC区共有12个基因,其中编码蛋白基因11个,编码tRNA基因1个。反向重复区IRa区与IRb区各有17个基因,其中编码tRNA基因7个,编码蛋白质基因6个,编码rRNA基因4个。注释结果还表明,在JK1408叶绿体基因组中蛋白质编码基因rps19位于IRb与LSC的边界,蛋白质编码基因ycf1位于IRb与LSC的边界。注释结果中还有一个5′端位于LSC区,3′端位于2个IR区的分裂基因rps12(图1,表1)。
表1 JK1408叶绿体基因组基因分类
Table 1
| 功能 Function | 家族 Family name | 基因代码 Code | 基因 Gene |
|---|---|---|---|
| 复制基因 Duplicate gene | 核糖体小亚基 | rps | rps11,rps12,rps12,rps14,rps15,rps16,rps18,rps19,rps2,rps3,rps4,rps7,rps7,rps8 |
| rRNA | rrn | rrn16,rrn23,rrn4.5,rrn5,rrn5,rrn4.5,rrn23,rrn16 | |
| 核糖体大亚基 | rpl | rpl14,rpl16,rpl2,rpl2,rpl20,rpl22,rpl23,rpl23,rpl32,rpl33,rpl36 | |
| 脱氧核糖核酸依赖性RNA聚合酶 | rpo | rpoA,rpoB,rpoC1,rpoC2 | |
| tRNA | trn | trnM-CAU,trnL-CAA,trnV-GAC,trnI-GAU,trnA-UGC,trnR-ACG,trnN-GUU,trnL-UAG,trnN-GUU,trnR-ACG,trnA-UGC,trnI-GAU, trnV-GAC,trnL-CAA,trnM-CAU,trnP-UGG,trnW-CCA,trnM-CAU,trnV-UAC,trnF-GAA,trnL-UAA,trnT-UGU,trnS-GCU,trnfM-CAU,trnG-GCC,trnS-UGA,trnT-GGU,trnG-UCC,trnR-UCU,trnE-UUC,trnY-GUA,trnD-GUC,trnC-GCA,trnS-GCU,trnQ-UUG,trnH-GUG | |
| 光合作用基因 Photosynthesis gene | ATP合酶亚基 | atp | atpA,atpB,atpE,atpF,atpH,atpI |
| NADH-脱氢酶亚基 | ndh | ndhA,ndhB,ndhB,ndhC,ndhD,ndhE,ndhF,ndhG,ndhH,ndhI,ndhJ,ndhK | |
| 细胞色素b/f复合物的亚基 | pet | petA,petB,petD,petG,petL,petN | |
| 光系统I的亚基 | psa | psaA,psaB,psaC,psaI,psaJ,ycf3 | |
| 光系统II的亚基 | psb | psbH,psbA,psbB,psbC,psbD,psbE,psbF,psbL,psbI,psbJ,psbK,psbM,psbN,psbT,psbZ | |
| 二磷酸核酮糖氧合酶/羧化酶亚基 | rbc | rbcL | |
| 其他基因Other gene | 乙酰辅酶A-羧化酶亚基 | acc | accD |
| 包膜蛋白基因 | cem | cemA | |
| c型细胞色素合成基因 | ccs | ccsA | |
| 蛋白酶基因 | clp | clpP | |
| 成熟酶基因 | mat | matK | |
| 平移起始因子 | Inf | infA | |
| 未知基因Unknown gene | 假定叶绿体阅读框 | ycf | ycf1,ycf2,ycf2,ycf4 |
2.3 密码子偏好性分析
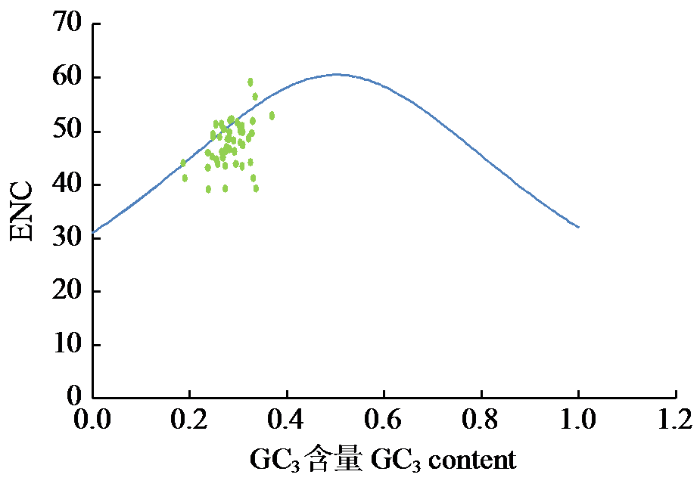
在JK1408叶绿体全基因组中,共获得129条CDS,为减小误差选取序列长度大于300 bp的CDS,共计50条用于分析密码子偏好性获取相关参数。50条CDS的GC1、GC2、GC3和GCall平均GC含量分别为47.06%(32.82%~59.39%)、39.29%(27.43%~55.47%)、28.36%(18.63%~36.91%)和38.24%(29.7%~45.74%)(表2),平均GC含量GC1>GC2>GC3,表明密码子第1、2和3位的碱基种类含量存在差异,第3位碱基的GC值较小,JK1408叶绿体基因密码子偏向于以A或U(T)作为第3位碱基。以有效密码子数(ENC)=45为标准衡量基因密码子使用偏好性程度,取值范围在20~61,20表示每个氨基酸只有1个有效密码子数,61表示所有的密码子都均衡使用。50条CDS的ENC值大于45的有37个基因(占比74%),小于45的有13个(占比26%)。ENC值最大的是ycf3基因,为59.17,最小的是rpl22基因,为39.13。ENC平均值为47.62,表明JK1408叶绿体基因密码子使用偏好性较弱[9]。分析发现,JK1408叶绿体基因组密码子适应指数(CAI)平均值为0.1719,更为接近0,与ENC值共同表明密码子使用偏好性较弱。JK1408叶绿体基因组最优密码子使用频率(FOP)平均值为0.3537,表明最优密码子的使用程度并不高。
表2 JK1408叶绿体基因组密码子不同位置的GC含量及ENC值
Table 2
| 基因 Gene | GC1 (%) | GC2 (%) | GC3 (%) | GCall (%) | 有效密码子数 ENC | 基因 Gene | GC1 (%) | GC2 (%) | GC3 (%) | GCall (%) | 有效密码子数 ENC |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ycf4 | 44.32 | 40.54 | 30.27 | 38.38 | 51.04 | psaA | 52.86 | 43.68 | 32.76 | 43.10 | 49.59 |
| ycf3 | 47.34 | 38.46 | 32.54 | 39.45 | 59.17 | petD | 51.55 | 39.13 | 27.33 | 39.34 | 39.25 |
| ycf2 | 41.89 | 34.24 | 36.91 | 37.68 | 52.81 | petB | 49.07 | 41.67 | 27.31 | 39.35 | 43.52 |
| ycf1 | 34.94 | 27.43 | 26.61 | 29.70 | 46.14 | petA | 52.65 | 37.38 | 30.84 | 40.29 | 50.97 |
| rps8 | 41.48 | 40.74 | 28.15 | 36.79 | 48.57 | ndhK | 43.81 | 44.25 | 26.55 | 38.20 | 51.26 |
| rps7 | 53.21 | 46.15 | 23.72 | 41.03 | 46.00 | ndhJ | 50.94 | 37.11 | 30.82 | 39.62 | 43.40 |
| rps4 | 53.47 | 39.60 | 28.71 | 40.59 | 52.21 | ndhI | 41.92 | 35.93 | 25.75 | 34.53 | 43.82 |
| rps3 | 46.12 | 33.33 | 23.74 | 34.40 | 43.16 | ndhH | 50.25 | 36.04 | 28.17 | 38.16 | 49.79 |
| rps2 | 43.46 | 43.04 | 27.43 | 37.97 | 46.26 | ndhG | 45.76 | 33.90 | 28.25 | 35.97 | 52.00 |
| rps18 | 34.31 | 42.16 | 25.49 | 33.99 | 44.76 | ndhF | 36.96 | 35.89 | 24.60 | 32.48 | 45.30 |
| rps14 | 44.55 | 47.52 | 33.66 | 41.91 | 39.24 | ndhE | 42.16 | 34.31 | 18.63 | 31.70 | 43.98 |
| rps11 | 54.74 | 55.47 | 27.01 | 45.74 | 50.38 | ndhC | 45.45 | 33.06 | 24.79 | 34.44 | 48.97 |
| rpoC2 | 46.52 | 37.49 | 30.45 | 38.15 | 50.87 | ndhB | 41.29 | 38.55 | 32.09 | 37.31 | 48.60 |
| rpoC1 | 50.29 | 36.52 | 26.23 | 37.68 | 48.88 | ndhA | 45.05 | 39.84 | 18.96 | 34.71 | 41.16 |
| rpoB | 50.52 | 37.32 | 27.80 | 38.55 | 48.50 | matK | 38.52 | 34.33 | 27.94 | 33.60 | 46.82 |
| rpoA | 44.35 | 32.14 | 29.76 | 35.42 | 51.62 | clpP | 59.39 | 38.07 | 30.46 | 42.64 | 50.07 |
| rpl22 | 39.35 | 40.00 | 23.87 | 34.41 | 39.13 | cemA | 39.13 | 28.26 | 30.87 | 32.75 | 49.80 |
| rpl20 | 37.80 | 45.67 | 27.56 | 37.01 | 46.96 | ccsA | 32.82 | 37.15 | 24.77 | 31.58 | 49.45 |
| rpl2 | 50.18 | 47.27 | 33.45 | 43.64 | 56.40 | atpI | 50.40 | 37.90 | 28.23 | 38.84 | 46.54 |
| rpl14 | 56.10 | 37.40 | 26.83 | 40.11 | 45.15 | atpF | 46.49 | 32.43 | 32.97 | 37.30 | 51.82 |
| rbcL | 57.00 | 43.00 | 30.86 | 43.62 | 47.42 | atpE | 52.96 | 41.04 | 25.37 | 39.80 | 51.30 |
| psbD | 52.26 | 43.22 | 32.49 | 42.66 | 44.10 | atpB | 55.71 | 41.68 | 29.06 | 42.15 | 48.21 |
| psbC | 53.38 | 46.20 | 29.54 | 43.04 | 43.82 | atpA | 56.19 | 39.49 | 25.15 | 40.27 | 46.83 |
| psbB | 53.83 | 45.97 | 30.45 | 43.42 | 47.88 | accD | 42.20 | 35.76 | 30.35 | 36.11 | 50.67 |
| psbA | 50.00 | 43.50 | 33.05 | 42.18 | 41.16 | 平均Average | 47.06 | 39.29 | 28.36 | 38.24 | 47.62 |
| psaB | 48.03 | 43.40 | 29.25 | 40.23 | 46.17 |
对JK1408叶绿体基因组筛选出的50条CDS的GC含量和ENC值进行相关性分析(表3),GC1与GC2、GC1与GCall、GC2与GCall、GC3与GCall、GC3与ENC相关性为极显著水平,密码子第1、2位碱基的成分相似,而第3位碱基组成与前2位碱基具有一定差异,这导致GC3与GC1、GC2二者含量之间的相关性均不显著;ENC与GC1、GC2之间无显著相关性,但与GC3含量具有极显著相关性,说明第3位碱基含量变化会对JK1408叶绿体基因组密码子偏好性产生重要影响。
表3 JK1408叶绿体基因组密码子数各位置GC含量与ENC值的相关性分析
Table 3
| 指标 Index | GC1 | GC2 | GC3 | GCall | 有效密码子数 ENC |
|---|---|---|---|---|---|
| GC2 | 0.389** | ||||
| GC3 | 0.210 | 0.111 | |||
| GCall | 0.838** | 0.737** | 0.498** | ||
| 有效密码子数ENC | 0.084 | -0.114 | 0.391** | 0.121 | |
| 密码子数N | -0.141 | -0.277* | 0.280 | -0.123 | 0.157 |
“*”代表显著相关(P < 0.05);“**”代表极显著相关(P < 0.01)。
“*”indicates significant correlation (P < 0.05);“**”indicates extremely significant correlation (P < 0.01).
通过对氨基酸密码子的相对同义密码子(RSCU)值分析(表4)可知,50条CDS共有20 244个密码子,亮氨酸(Leu)数量最多,具有2154个密码子,占比10.7%;半胱氨酸(Cys)数量最少,只有217个密码子,占比1.1%。结果显示,RSCU值>1的密码子有32个,其中16个密码子的第3位碱基为U,13个密码子的第3位碱基为A,3个密码子的第3位碱基为G,表明JK1408偏好于使用A/U作为密码子的末尾碱基。
表4 JK1408叶绿体基因组各氨基酸的RSCU分析
Table 4
| 氨基酸 Amino acid | 密码子 Codon | 数目 Number | 相对 同义 密码子 RSCU | 氨基酸 Amino acid | 密码子 Codon | 数目 Number | 相对 同义 密码子 RSCU |
|---|---|---|---|---|---|---|---|
| 苯丙氨酸 Phe | UUU | 768 | 1.35 | 丙氨酸 Ala | GCU | 509 | 1.77 |
| UUC | 368 | 0.65 | GCC | 182 | 0.63 | ||
| 亮氨酸 Leu | UUA | 690 | 1.92 | GCA | 335 | 1.16 | |
| UUG | 458 | 1.28 | GCG | 127 | 0.44 | ||
| CUU | 458 | 1.28 | 酪氨酸 Tyr | UAU | 628 | 1.63 | |
| CUC | 128 | 0.36 | UAC | 142 | 0.37 | ||
| CUA | 289 | 0.81 | 组氨酸 His | CAU | 357 | 1.5 | |
| CUG | 131 | 0.36 | CAC | 119 | 0.5 | ||
| 异亮氨酸 Ile | AUU | 836 | 1.46 | 谷氨酰胺 Gln | CAA | 570 | 1.53 |
| AUC | 340 | 0.6 | CAG | 176 | 0.47 | ||
| AUA | 536 | 0.94 | 天冬酰胺 Asn | AAU | 790 | 1.57 | |
| 甲硫氨酸 Met | AUG | 473 | 1 | AAC | 214 | 0.43 | |
| 缬氨酸 Val | GUU | 395 | 1.46 | 赖氨酸 Lys | AAA | 788 | 1.49 |
| GUC | 133 | 0.49 | AAG | 267 | 0.51 | ||
| GUA | 410 | 1.51 | 天冬氨酸 Asp | GAU | 663 | 1.58 | |
| GUG | 146 | 0.54 | GAC | 176 | 0.42 | ||
| 丝氨酸 Ser | UCU | 441 | 1.77 | 谷氨酸 Glu | GAA | 822 | 1.51 |
| UCC | 219 | 0.88 | GAG | 267 | 0.49 | ||
| UCA | 304 | 1.22 | 半胱氨酸 Cys | UGU | 157 | 1.45 | |
| UCG | 119 | 0.48 | UGC | 60 | 0.55 | ||
| AGU | 326 | 1.31 | 色氨酸 Trp | UGG | 367 | 1 | |
| AGC | 82 | 0.33 | 精氨酸 Arg | AGA | 362 | 1.84 | |
| 脯氨酸 Pro | CCU | 344 | 1.61 | AGG | 123 | 0.62 | |
| CCC | 145 | 0.68 | CGU | 275 | 1.39 | ||
| CCA | 234 | 1.09 | CGC | 78 | 0.4 | ||
| CCG | 134 | 0.63 | CGA | 260 | 1.32 | ||
| 苏氨酸 Thr | ACU | 407 | 1.64 | CGG | 85 | 0.43 | |
| ACC | 187 | 0.75 | 甘氨酸 Gly | GGU | 470 | 1.35 | |
| ACA | 303 | 1.22 | GGC | 159 | 0.46 | ||
| ACG | 94 | 0.38 | GGA | 536 | 1.53 | ||
| GGG | 232 | 0.66 |
2.4 ENC-plot分析
图2
图2
JK1408叶绿体基因组ENC-plot分析
Fig.2
ENC-plot analysis in the chloroplast genome of JK1408
表5 JK1408叶绿体基因组ENC比值频数分布表
Table 5
| 组段 Class range | 组中值 Class mid value | 频数 Frequency number | 频率 Frequency (%) |
|---|---|---|---|
| [-0.15,-0.05) | -0.10 | 1 | 2 |
| [-0.05,0.05) | 0.00 | 22 | 44 |
| [0.05,0.15) | 0.10 | 20 | 40 |
| [0.15,0.25) | 0.20 | 6 | 12 |
| [0.25,0.35) | 0.30 | 1 | 2 |
| 合计Total | 0.50 | 50 | 1 |
2.5 中性绘图及PR2-plot分析
图3
图3
JK1408叶绿体基因组中性绘图分析
Fig.3
Neutrality plot analysis in the chloroplast genome of JK1408
图4
图4
JK1408叶绿体基因组PR2-plot偏倚分析
Fig.4
PR2-plot bias analysis in the chloroplast genome of JK1408
2.6 最优密码子的确定
按照50条CDS的ENC值由小到大排序,用ENC值小的高表达基因(rpl22、rps14、petD、psbA、ndhA)和ENC值大的低表达基因(ndhD、rps4、ycf2、rpl2、ycf3)建立高、低表达库(表6),计算ΔRSCU(ΔRSCU=RSCUhigh-RSCUlow),结果显示,ΔRSCU≥0.08的密码子有32个。当密码子同时满足2个条件即密码子高表达(ΔRSCU≥0.08)和密码子高频出现(RSCU≥1)时,该密码子即为最优密码子[10]。经过计算,JK1408的叶绿体基因密码子偏爱A/U结尾,最优密码子为CUU、CCA、ACU、GCU、UAU、CAU、AAA、GAU和GGA。
表6 JK1408叶绿体基因组最优密码子的确定
Table 6
| 氨基酸 Amino acid | 密码子 Codon | 高表达基因 High expression gene | 低表达基因 Low expression gene | 相对 使用度 差值 ΔRSCU | 氨基酸 Amino acid | 密码子 Codon | 高表达基因 High expression gene | 低表达基因 Low expression gene | 相对 使用度 差值 ΔRSCU | ||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 数目 Number | 相对同义 密码子 RSCU | 数目 Number | 相对同义 密码子 RSCU | 数目 Number | 相对同义 密码子 RSCU | 数目 Number | 相对同义 密码子 RSCU | ||||||||
| 苯丙氨酸 Phe | UUU | 91 | 1.33 | 36 | 1.57 | -0.24 | 丙氨酸 Ala | GCU* | 46 | 1.61 | 40 | 1.47 | 0.14 | ||
| UUC* | 86 | 0.67 | 24 | 0.43 | 0.24 | GCC*** | 22 | 0.90 | 7 | 0.28 | 0.62 | ||||
| 亮氨酸 Leu | UUA | 67 | 1.74 | 51 | 2.33 | -0.59 | GCA | 31 | 0.90 | 28 | 1.66 | -0.76 | |||
| UUG | 77 | 1.24 | 25 | 1.39 | -0.16 | GCG | 13 | 0.59 | 9 | 0.59 | 0.00 | ||||
| CUU* | 82 | 1.31 | 28 | 1.08 | 0.23 | 酪氨酸 Tyr | UAU* | 91 | 1.45 | 28 | 1.34 | 0.11 | |||
| CUC** | 28 | 0.48 | 2 | 0.15 | 0.33 | UAC | 24 | 0.55 | 15 | 0.66 | -0.11 | ||||
| CUA | 48 | 0.84 | 27 | 0.90 | -0.06 | 组氨酸 His | CAU** | 62 | 1.66 | 11 | 1.17 | 0.49 | |||
| CUG* | 26 | 0.39 | 5 | 0.14 | 0.24 | CAC | 17 | 0.34 | 7 | 0.43 | -0.09 | ||||
| 异亮氨酸 Ile | AUU | 99 | 1.29 | 55 | 1.77 | -0.48 | 谷氨酰胺 Gln | CAA | 75 | 1.28 | 22 | 1.69 | -0.41 | ||
| AUC* | 58 | 0.59 | 18 | 0.34 | 0.25 | CAG** | 38 | 0.72 | 4 | 0.31 | 0.41 | ||||
| AUA* | 79 | 1.12 | 25 | 0.88 | 0.23 | 天冬酰胺 Asn | AAU | 130 | 1.40 | 34 | 1.44 | -0.04 | |||
| 甲硫氨酸 Met | AUG | 58 | 1.00 | 30 | 1.00 | 0.00 | AAC | 45 | 0.60 | 16 | 0.56 | 0.04 | |||
| 缬氨酸 Val | GUU | 36 | 0.98 | 27 | 1.85 | -0.87 | 赖氨酸 Lys | AAA* | 120 | 1.62 | 34 | 1.39 | 0.23 | ||
| GUC*** | 23 | 0.64 | 4 | 0.14 | 0.50 | AAG* | 62 | 0.38 | 6 | 0.21 | 0.17 | ||||
| GUA | 30 | 1.38 | 33 | 1.76 | -0.38 | 天冬氨酸 Asp | GAU* | 123 | 1.61 | 24 | 1.36 | 0.25 | |||
| GUG*** | 27 | 0.10 | 4 | 0.25 | 0.74 | GAC | 30 | 0.39 | 6 | 0.64 | -0.25 | ||||
| 丝氨酸 Ser | UCU | 67 | 1.31 | 27 | 1.24 | 0.08 | 谷氨酸 Glu | GAA | 100 | 1.48 | 46 | 1.68 | -0.19 | ||
| UCC | 47 | 1.05 | 16 | 1.31 | -0.26 | GAG* | 54 | 0.52 | 11 | 0.32 | 0.19 | ||||
| UCA | 57 | 1.41 | 19 | 1.65 | -0.24 | 半胱氨酸 Cys | UGU | 21 | 1.32 | 8 | 1.80 | -0.48 | |||
| UCG** | 30 | 0.71 | 3 | 0.27 | 0.44 | UGC** | 11 | 0.68 | 1 | 0.20 | 0.48 | ||||
| AGU* | 45 | 1.34 | 20 | 1.20 | 0.14 | 色氨酸 Trp | UGG | 47 | 1.00 | 26 | 1.00 | 0.00 | |||
| AGC | 14 | 0.17 | 6 | 0.32 | -0.16 | 精氨酸 Arg | CGU | 32 | 2.17 | 20 | 2.42 | -0.24 | |||
| 脯氨酸 Pro | CCU | 36 | 1.18 | 29 | 1.97 | -0.79 | CGC** | 18 | 0.56 | 3 | 0.25 | 0.31 | |||
| CCC* | 26 | 0.84 | 9 | 0.57 | 0.27 | CGA | 38 | 1.02 | 12 | 1.15 | -0.14 | ||||
| CCA* | 41 | 1.26 | 15 | 1.07 | 0.19 | CGG* | 17 | 0.41 | 3 | 0.26 | 0.15 | ||||
| CCG** | 18 | 0.72 | 6 | 0.39 | 0.33 | AGA | 61 | 1.25 | 15 | 1.59 | -0.34 | ||||
| 苏氨酸 Thr | ACU* | 48 | 1.59 | 22 | 1.40 | 0.19 | AGG* | 34 | 0.59 | 4 | 0.34 | 0.25 | |||
| ACC* | 22 | 0.77 | 11 | 0.64 | 0.12 | 甘氨酸 Gly | GGU | 45 | 1.05 | 48 | 1.77 | -0.72 | |||
| ACA | 40 | 1.25 | 23 | 1.60 | -0.35 | GGC | 9 | 0.19 | 10 | 0.32 | -0.13 | ||||
| ACG | 19 | 0.38 | 2 | 0.35 | 0.04 | GGA*** | 69 | 1.78 | 22 | 1.26 | 0.52 | ||||
| GGG** | 40 | 0.97 | 8 | 0.64 | 0.33 | ||||||||||
“*”:ΔRSCU≥0.08;“**”:ΔRSCU≥0.30;“***”:ΔRSCU≥0.50;下划线显示RSCU>1.00;加粗显示的密码子表示其为最优密码子。
The underlined indicates RSCU > 1; The bolded codon indicates the optimal codon.
3 讨论
叶绿体基因组拷贝数高、进化速率适中且全基因组序列更容易获取的特点使得其在物种鉴定、分类、系统发育分析和基因工程等方面呈现出巨大的利用价值。本研究通过高通量二代测序技术获取了JK1408的全基因组数据,组装过程确定MK341450作为最佳参考序列,MK341450是MAX1细胞质类型的向日葵栽培品种,初步判断JK1408为MAX1细胞质类型,更加清晰的证据需要后续线粒体基因组的研究补充。有关黎平瘤果茶[11]、蒺藜苜蓿[12]、再力花[13]、绣球[14]和西南牡蒿[15]的研究支持大多数高等植物的叶绿体基因组呈现双链环状四分体结构,本研究得出的双链环状四分体结构与其结果一致。研究[9]显示,10个一年生栽培型向日葵叶绿体基因组序列长度在151 100~151 150 bp,所有材料均注释出127个基因,而本研究组装的JK1408的叶绿体基因组大小为151 100 bp,注释出129个基因,由于油用和食用向日葵种质来源不同,系统发育定位不清晰,在组装注释过程中参考基因组的选择存在一定的局限性,注释结果因而产生偏差。
密码子偏好性问题在生物学研究中一直是热点话题,突变压力、自然选择、基因重组、基因遗传漂变以及基因水平转移等因素在物种的长期进化中均可能影响到密码子的偏好性,而突变压力和自然选择是主要因素[16]。本研究中JK1408的ENC平均值为47.62,CAI平均值为0.1719,FOP平均值为0.3537,表明该品种密码子使用偏好性较弱,最优密码子的使用程度并不高,在裸果木[1]、人参[17]、菘蓝[18]、百合属[19]及柏科[20]等大多数植物的叶绿体基因组密码子偏好性研究中均显示其偏好性较弱,这也揭示了植物叶绿体基因组密码子使用模式相对保守的现象。相关性分析表明第3位碱基含量变化会对JK1408叶绿体基因组密码子偏好性产生重要影响,多数研究支持这种现象,如梁山慈竹[21]、硬尖神香草和欧神香草[22]、栽培大麦和野生大麦[23]、裸果木[1]、人参[17]及菘蓝[18]。JK1408叶绿体基因组密码子ENC-plot图、中性绘图分析和PR2-plot偏倚分析显示自然选择和突变压力对不同基因的密码子的偏好性影响程度不同,自然选择对密码子使用模式产生了重要影响,这与王雪等[9]的研究结果一致。本研究确定了JK1408叶绿体基因组中均以A/U结尾的最优密码子CUU、CCA、ACU、GCU、UAU、CAU、AAA、GAU和GGA,为密码子偏好性方面的应用如预测未知蛋白基因位置、提高基因工程中目的基因表达量等提供了理论支撑。
4 结论
通过高通量测序技术获取了JK1408全基因组数据,组装、注释其叶绿体基因组并绘制了物理图谱,JK1408叶绿体基因组信息的释放丰富了向日葵叶绿体基因组构建的系统进化树,有助于理清向日葵品种的系统发育关系,寻找不同品种间的叶绿体基因组高变异区域还可以进行品种的鉴定,更好地服务于向日葵育种工程。系统分析JK1408的密码子偏好性,发现JK1408的密码子使用模式受自然选择主导,确定了9个均以A/U结尾的最优密码子CUU、CCA、ACU、GCU、UAU、CAU、AAA、GAU和GGA。
参考文献
Codon usage and genome volution
NOVOPlasty: de novo assembly of organelle genomes from whole genome data
GeSeq-versatile and accurate annotation of organelle genomes
黎平瘤果茶的叶绿体基因组特征及系统发育分析
蒺藜苜蓿叶绿体密码子偏好性分析
DOI:10.11686/cyxb2015016
[本文引用: 1]

本文对蒺藜苜蓿叶绿体基因组全序列密码子进行分析,筛选出50条CDS(coding DNA sequence)利用CodonW软件进行分析其密码子使用模式。结果显示,蒺藜苜蓿叶绿体基因组密码子第3位碱基GC含量为26.9%,即第3位密码子富含A和U,ENC值在37.11~51.91之间密码子偏好性较弱。相对同义密码子使用度分析显示RSCU值大于1的密码子有23个,其中以A和U为结尾20个。中性绘图分析显示GC<sub>12</sub>与GC<sub>3</sub>的相关系数为0.341,相关性不显著,回归系数为0.4843;单基因ENC比值多分布在-0.05~0.05,即大部分基因ENC值离ENC期望值较近;对应性分析,第一轴显示了12.50%的差异为主要影响因素,第一轴与ENC和GC<sub>3</sub>的相关系数分别为0.091和-0.092,均相关不显著。综合这几项分析发现蒺藜苜蓿叶绿体基因组密码子偏好性主要受到突变的影响,但是并不是唯一的影响因素,其他因素对密码子偏好性也可能有一定的影响。最终通过高表达优越密码子方法确定得出UUA、UUG、CCU等23个密码子为最优密码子,为之后对外源基因进行改造,提高其在叶绿体中的表达效率奠定了基础。
再力花叶绿体基因组特征和系统发育分析
绣球叶绿体基因组特征及密码子偏好性分析
Exposing synonymous mutations
DOI:10.1016/j.tig.2014.04.006
PMID:24954581
[本文引用: 1]

Synonymous codon changes, which do not alter protein sequence, were previously thought to have no functional consequence. Although this concept has been overturned in recent years, there is no unique mechanism by which these changes exert biological effects. A large repertoire of both experimental and bioinformatic methods has been developed to understand the effects of synonymous variants. Results from this body of work have provided global insights into how biological systems exploit the degeneracy of the genetic code to control gene expression, protein folding efficiency, and the coordinated expression of functionally related gene families. Although it is now clear that synonymous variants are important in a variety of contexts, from human disease to the safety and efficacy of therapeutic proteins, there is no clear consensus on the approaches to identify and validate these changes. Here, we review the diverse methods to understand the effects of synonymous mutations. Published by Elsevier Ltd.
人参叶绿体全基因组特征及密码子偏好性分析
菘蓝叶绿体基因组密码子使用偏好性分析
DOI:10.11733/j.issn.1007-0435.2024.08.005
[本文引用: 2]

为探究菘蓝(Isatis indigotica Fort.)叶绿体基因组密码子使用偏好性及其影响因素,本研究使用Codon W 1.4.2软件和在线软件CUSP对筛选到的52个蛋白质编码序列进行密码子使用模式分析。结果表明:菘蓝叶绿体基因组密码子总GC含量(Total GC content of codon,GC<sub>all</sub>)为37.59%,密码子第1位碱基GC含量(GC content of the first base of codon,GC<sub>1</sub>)>密码子第2位碱基GC含量(GC content of the second base of codon,GC<sub>2</sub>)>密码子第3位碱基GC含量(GC content of the third base of codon,GC<sub>3</sub>),且均小于50%,说明密码子第3位碱基拥有较低的GC含量且密码子偏好使用A或U。有效密码子数(Effective number of codon,ENC)值为35.92~55.32,密码子适应指数(Codon adaptation index,CAI)值为0.10~0.31,密码子偏好指数(Codon bias index,CBI)值为-0.24~0.20,最优密码子使用频率(Frequency of optimal codons,FOP)值为0.24~0.54,说明密码子使用偏好性较弱;通过中性绘图分析、PR2-plot分析、ENC-plot分析和对应性分析,发现菘蓝叶绿体基因组密码子使用偏好性受选择压力和突变等因素的共同影响,主要因素为自然选择。最优密码子分析共筛选出14个最优密码子,且均以A或U结尾。以上研究可为菘蓝的系统进化、环境适应性和种质改良提供理论基础。
硬尖神香草和欧神香草叶绿体基因组密码子偏好性分析