作物杂志,2022, 第3期: 73–79 doi: 10.16035/j.issn.1001-7283.2022.03.010
苦荞漆酶基因的克隆与生物信息学分析
杨晓琳1( ), 段迎1, 蔡苏云1, 贺润丽1(
), 段迎1, 蔡苏云1, 贺润丽1( ), 尹桂芳2, 王艳青2, 卢文洁2, 孙道旺2, 王莉花2(
), 尹桂芳2, 王艳青2, 卢文洁2, 孙道旺2, 王莉花2( )
)
- 1山西中医药大学中药与食品工程学院,030619,山西太原
2云南省农业科学院生物技术与种质资源研究所/云南省农业生物技术重点实验室/农业农村部西南作物基因资源与种质创制重点实验室,650221,云南昆明
Molecular Cloning and Bioinformatics Analyzing of Laccase in Fagopyrum tataricum
Yang Xiaolin1( ), Duan Ying1, Cai Suyun1, He Runli1(
), Duan Ying1, Cai Suyun1, He Runli1( ), Yin Guifang2, Wang Yanqing2, Lu Wenjie2, Sun Daowang2, Wang Lihua2(
), Yin Guifang2, Wang Yanqing2, Lu Wenjie2, Sun Daowang2, Wang Lihua2( )
)
- 1College of Traditional Chinese Medicine and Food Engineering, Shanxi University of Traditional Chinese Medicine, Taiyuan 030619, Shanxi, China
2Biotechnology and Germplasm Resources Institute, Yunnan Academy of Agricultural Sciences/Yunnan Provincial Key Laboratory of Agricultural Biotechnology/Key Laboratory of Southwestern Crop Gene Resources and Germplasm Innovation, Ministry of Agriculture and Rural Affairs, Kunming 650221, Yunnan, China
摘要:
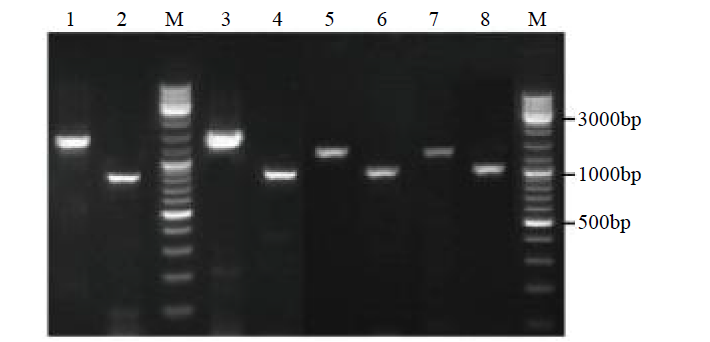
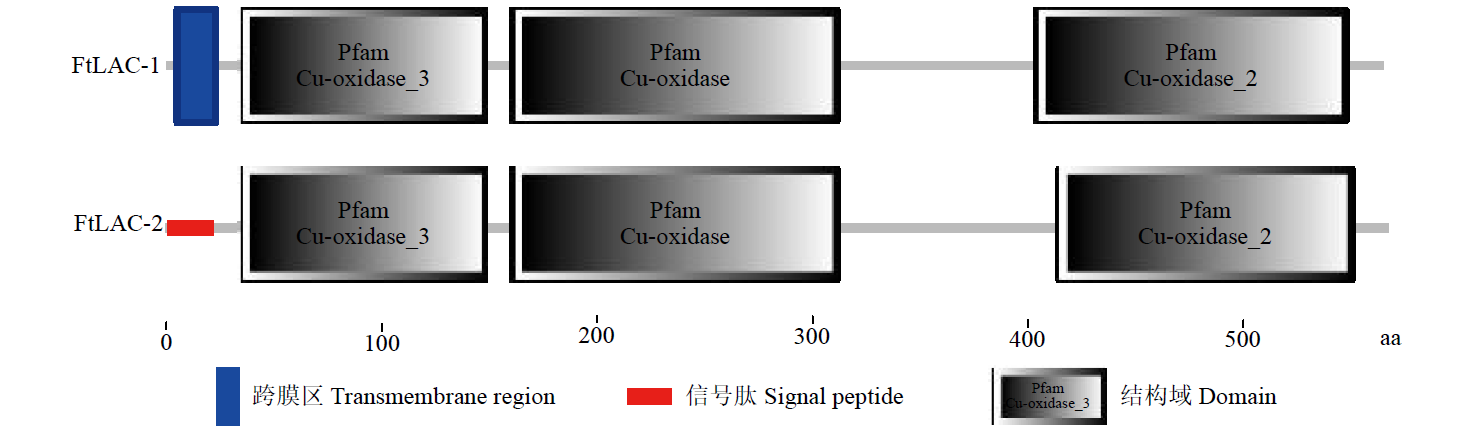
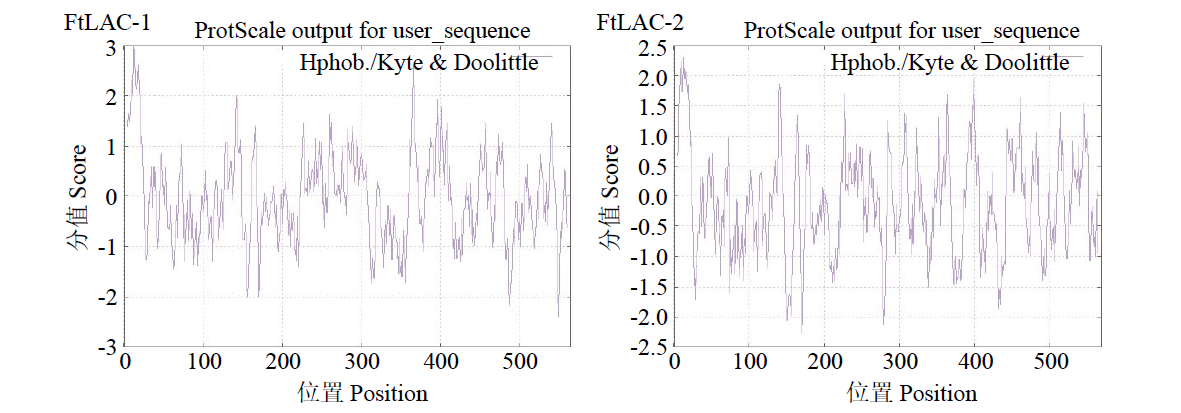
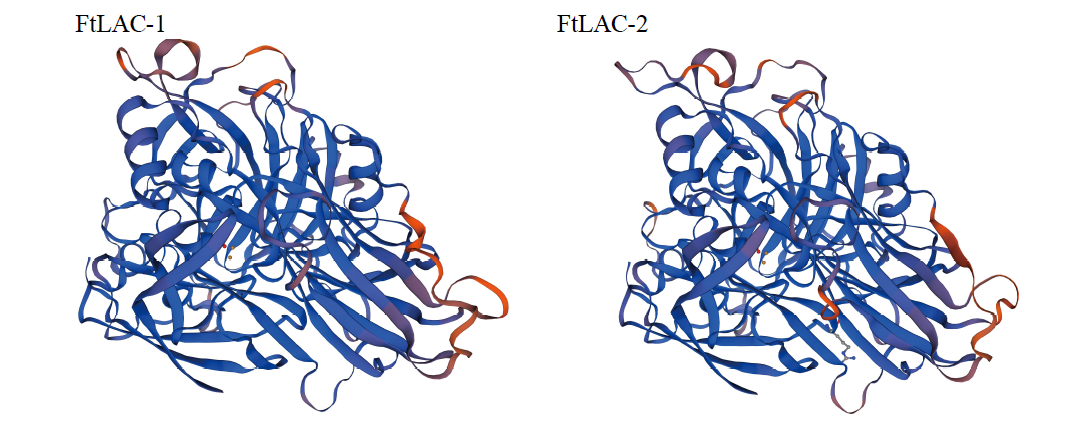
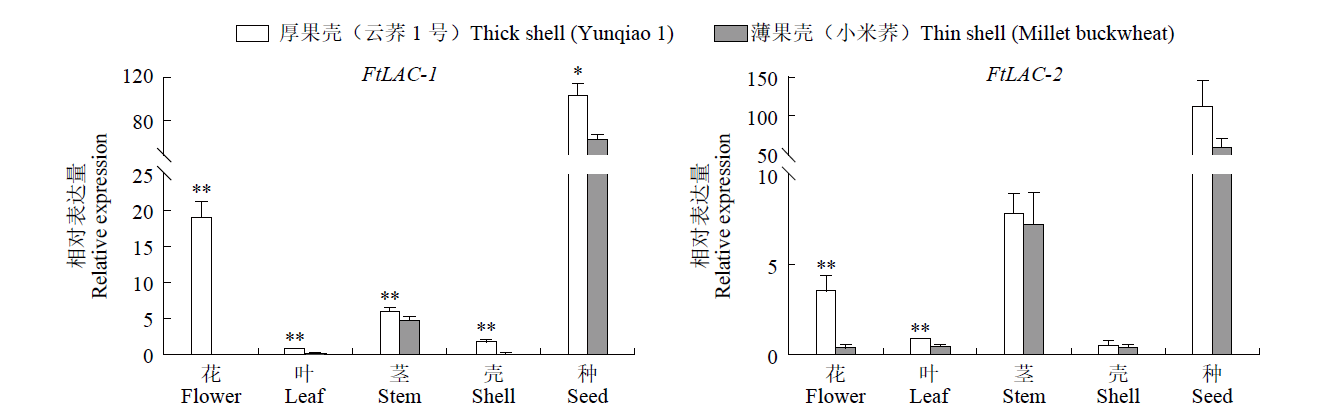
利用RT-PCR技术从苦荞中克隆出2条漆酶(laccase)基因,运用生物信息学技术对2个基因序列进行分析,预测结构域、二级和三级蛋白结构,进行蛋白同源比对及进化树分析。结果表明,FtLAC-1基因序列开放阅读框为1695bp,编码564个氨基酸,FtLAC-2基因序列开放阅读框为1707bp,编码568个氨基酸。FtLAC-1和FtLAC-2蛋白预测分子量分别为61.41和62.47kDa,理论等电点分别为9.45和9.41,为亲水性蛋白,2个基因序列相似性较低,氨基酸序列同源性不高。qRT-PCR分析出其在苦荞不同组织器官存在差异性表达。结论为进一步探索FtLAC-1和FtLAC-2在苦荞薄果壳形成中的功能提供理论基础。
| [1] | 陈庆富. 荞麦属植物科学. 北京: 科学出版社, 2012. |
| [2] | 陈曦. 苦荞功能成分研究文献综述. 食品安全导刊, 2018, 33(11):162. |
| [3] | 朱云辉, 郭元新. 我国苦荞资源的开发利用研究进展. 食品工业科技, 2014, 35(24):360-365. |
| [4] | 张余, 黄小燕, 昌敏, 等. 苦荞营养保健成分及其食品开发研究进展与展望. 粮食与油脂, 2019, 32(8):12-14. |
| [5] | 陈庆富, 陈其饺, 石桃雄, 等. 苦荞厚果壳性状的遗传及其与产量因素的相关性研究. 作物杂志, 2015(2):27-31,2. |
| [6] |
张盛春, 鞠常亮, 王小菁. 拟南芥漆酶基因AtLAC4参与生长及非生物胁迫响应. 植物学报, 2012, 47(4):357-365.
doi: 10.3724/SP.J.1259.2012.00357 |
| [7] |
Turlapati P V, Kim K W, Davin L B, et al. The laccase multigene family in Arabidopsis thaliana:towards addressing the mystery of their gene function(s). Planta, 2011, 233(3):439-470.
doi: 10.1007/s00425-010-1298-3 |
| [8] |
Schuetz M, Benske A, Smith R A, et al. Laccases direct lignification in the discrete secondary cell wall domains of protoxylem. Plant Physiology, 2014, 166(2):798-807.
doi: 10.1104/pp.114.245597 |
| [9] | 冯鹤翔, 涂轶. 木质素生物合成的研究. 青岛大学学报(自然科学版), 2018, 31(1):46-54. |
| [10] |
Vanholme R, Morreel K, Darrah C, et al. Metabolic engineering of novel lignin in biomass crops. New Phytologist, 2012, 196(4):978-1000.
doi: 10.1111/j.1469-8137.2012.04337.x pmid: 23035778 |
| [11] | Ranocha P, Chabannes M, Chamayou S, et al. Laccase down-regulation causes alterations in phenolic metabolism and cell wall structure in poplar. Journal of Plant Physiology, 2002, 129(1):145-155. |
| [12] | 吴朝昕. 薄壳苦荞果壳结构及其发育中的转录组学分析. 贵阳:贵州师范大学, 2020. |
| [13] | 罗冬梅, 赵亚文, 荆永琳, 等. 牛大力漆酶基因CsLAC17的克隆与载体构建. 分子植物育种, 2019, 17(6):1906-1912. |
| [14] | 艾嘉, 刘坤, 张蕾, 等. 细菌漆酶的生物信息学分析. 基因组学与应用生物学, 2019, 38(3):1070-1078. |
| [15] | 乔永刚, 贾孟君, 陈亮, 等. 忍冬ICE1基因的cDNA克隆及表达分析. 植物生理学报, 2019, 55(11):1647-1654. |
| [16] | 邹春虹. 苦荞营养保健成分及其食品开发分析. 食品界, 2020(8):121. |
| [17] | 李利超, 孙化雨, 娄永峰, 等. 毛竹漆酶基因PeLAC的克隆与表达分析. 植物科学学报, 2017, 35(2):252-259. |
| [18] | 陈亮. 丹参漆酶基因的克隆表达和功能研究. 福州:福建中医药大学, 2017. |
| [19] | 高敏霞, 冯新, 赖瑞联, 等. 米良1号猕猴桃漆酶基因的克隆与表达分析. 农业生物技术学报, 2018, 26(1):64-76. |
| [20] | 刘月, 杨健, 王龙, 等. 木质素合成途径及其调控梨褐皮性状研究进展. 果树学报, 2018, 35(增1):17-25. |
| [21] | 汤崴, 王洁, 张文颖, 等. 木质素在果树果核退化的作用. 分子植物育种, 2018, 16(11):3752-3762. |
| [22] | 王文然, 王晨, 解振强, 等. VvmiR397a及其靶基因VvLACs在葡萄果实发育中的作用分析. 园艺学报, 2018, 45(8):1441-1455. |
| [23] | 帅良, 赵昱清, 廖玲燕, 等. 龙眼漆酶基因(DlLac)的克隆及表达分析. 食品工业科技, 2017, 38(13):95-100. |
| [24] | 薛辉. 软籽石榴种子硬度的影响因素及转录组分析. 北京: 中国农业科学院, 2016. |
| [25] | 谢小波, 黄云, 田胜平, 等. 软籽石榴种子硬度发育与种皮细胞壁显微结构的关系研究. 园艺学报, 2017, 44(6):1174-1180. |
| [1] | 周菲. 向日葵HaLACS7基因的生物信息学和表达分析[J]. 作物杂志, 2022, (3): 104–108 |
| [2] | 施娴, 李洪有, 卢丙越, 周云, 赵继菊, 赵孟丽, 梁京, 孟衡玲. 3个苦荞品种对盐胁迫的生理响应及耐受性评价[J]. 作物杂志, 2022, (3): 149–154 |
| [3] | 卢平, 康庆芳, 赵孟瑶, 张凤洁, 武强强, 马芳芳, 王宇珅, 韩渊怀, 王兴春, 李雪垠. 谷子miR169家族及其靶基因的鉴定与功能分析[J]. 作物杂志, 2022, (2): 54–63 |
| [4] | 尹桂芳, 段迎, 杨晓琳, 蔡苏云, 王艳青, 卢文洁, 孙道旺, 贺润丽, 王莉花. 苦荞FtC4H基因克隆与生物信息学分析[J]. 作物杂志, 2022, (1): 77–83 |
| [5] | 宋坤锋, 郝风声, 姚欣, 王静, 刘卫群. 烟草Nt14-3-3-like C基因的克隆与功能分析[J]. 作物杂志, 2021, (5): 6–13 |
| [6] | 翁文凤, 伍小方, 张凯旋, 唐宇, 江燕, 阮景军, 周美亮. 过表达FtbZIP5提高苦荞毛状根黄酮积累及其耐盐性[J]. 作物杂志, 2021, (4): 1–9 |
| [7] | 贾瑞玲, 赵小琴, 南铭, 陈富, 刘彦明, 魏立平, 刘军秀, 马宁. 64份苦荞种质资源农艺性状遗传多样性分析与综合评价[J]. 作物杂志, 2021, (3): 19–27 |
| [8] | 靳建刚, 田再芳. 山西北部地区引种苦荞品种的灰色关联度分析[J]. 作物杂志, 2021, (2): 52–56 |
| [9] | 刘晓丽, 韩利涛, 魏楠, 申飞, 蔡一林. 玉米ZmGS5基因克隆、分子特性分析及对拟南芥的遗传转化[J]. 作物杂志, 2021, (1): 16–25 |
| [10] | 马名川, 刘龙龙, 刘璋, 周建萍, 南成虎, 张丽君. 苦荞全基因组SSR位点特征分析与分子标记开发[J]. 作物杂志, 2021, (1): 38–46 |
| [11] | 王志龙, 薛应红, 郝月茹, 刘宝玲, 苑丽霞, 薛金爱, 李润植. 亚麻荠油脂相关转录因子CsLEC2基因家族的鉴定及表达分析[J]. 作物杂志, 2020, (5): 23–32 |
| [12] | 卢晓玲, 何铭, 张凯旋, 廖志勇, 周美亮. 苦荞鼠李糖基转移酶FtF3GT1基因的克隆与转化毛状根研究[J]. 作物杂志, 2020, (5): 33–40 |
| [13] | 杨学乐, 张璐, 李志清, 何录秋. 苦荞种质资源表型性状的遗传多样性分析[J]. 作物杂志, 2020, (5): 53–58 |
| [14] | 徐园园, 赵鹏, 洪权春, 朱晓琴, 裴冬丽. 小麦转录因子基因TaMYB70的分离和表达分析[J]. 作物杂志, 2020, (4): 84–90 |
| [15] | 李春花, 黄金亮, 尹桂芳, 王艳青, 卢文洁, 孙道旺, 王春龙, 郭来春, 洪波, 任长忠, 王莉花. 苦荞粒形相关性状的遗传分析[J]. 作物杂志, 2020, (3): 42–46 |
|
||