作物杂志,2021, 第1期: 38–46 doi: 10.16035/j.issn.1001-7283.2021.01.006
苦荞全基因组SSR位点特征分析与分子标记开发
马名川( ), 刘龙龙, 刘璋, 周建萍, 南成虎, 张丽君(
), 刘龙龙, 刘璋, 周建萍, 南成虎, 张丽君( )
)
- 山西农业大学农业基因资源研究中心/农业农村部黄土高原作物基因资源与种质创制重点实验室/杂粮种质资源发掘与遗传改良山西省重点实验室,030031,山西太原
Analysis of SSR Loci in Whole Genome and Development of Molecular Markers in Tartary Buckwheat
Ma Mingchuan( ), Liu Longlong, Liu Zhang, Zhou Jianping, Nan Chenghu, Zhang Lijun(
), Liu Longlong, Liu Zhang, Zhou Jianping, Nan Chenghu, Zhang Lijun( )
)
- Center for Agricultural Genetic Resources Research, Shanxi Agricultural University/Key Laboratory of Crop Gene Resources and Germplasm Enhancement on Loess Plateau, Ministry of Agriculture and Rural Affairs/Shanxi Key Laboratory of Genetic Resources and Genetic Improvement of Minor Crops, Taiyuan 030031, Shanxi, China
摘要:
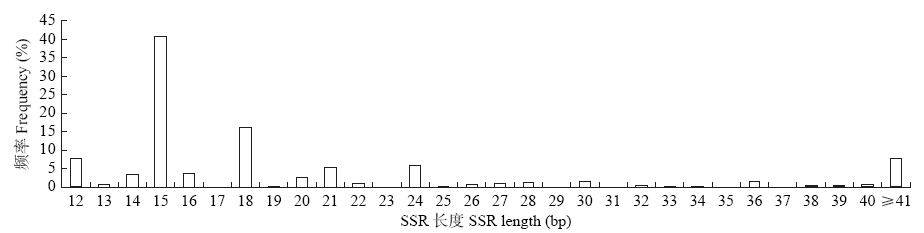
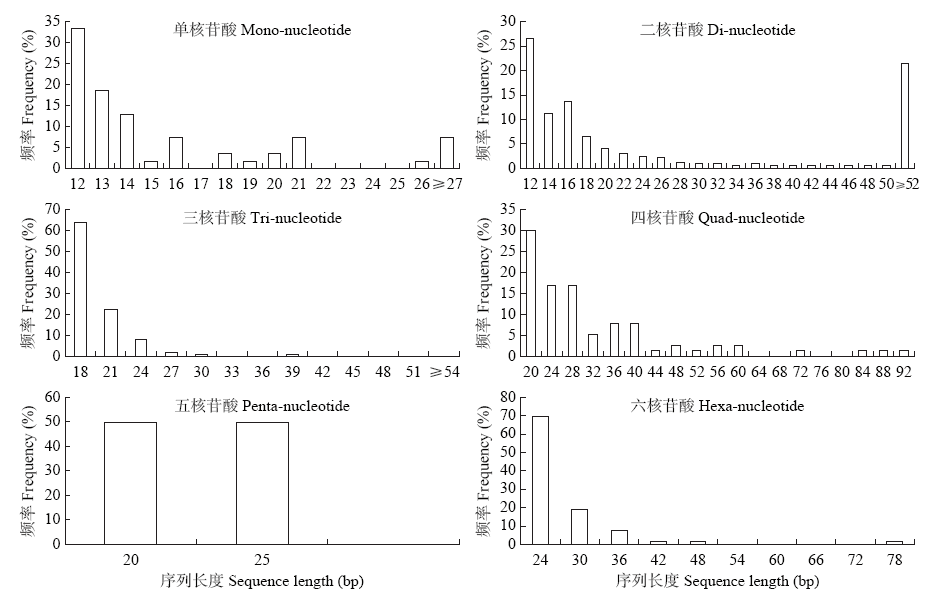
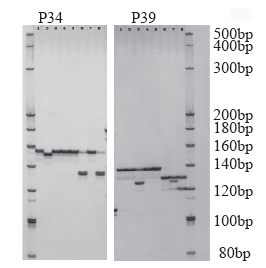
目前苦荞SSR多态性标记数量较少,根据已发表的苦荞基因组测序数据,利用MISA软件对1~6核苷酸重复的SSR位点进行了查找和序列特征分析,批量设计引物并对引物进行了有效性和多态性检测。结果表明,苦荞基因组中共检测到1 640个SSR位点,其中三核苷酸重复型SSR最多,占比63.29%,五核苷酸重复型最少,仅占0.12%。AT/TA、AAG/CTT、ACC/GGT和ATC/GAT为出现频率较高的重复基序。苦荞基因组SSR序列长度变化范围为12~476bp,平均长度23.14bp,长度12~19bp的占比71.71%,长度≥20bp的占比28.29%。根据不同类型SSR位点设计并合成引物479对,选择200对引物对5份苦荞资源和3份甜荞资源进行多态性检测,有56对扩增出多态性条带,17对在苦荞种质中产生多态性条带,48对在甜荞种质中产生多态性条带,9对同时在两种种质中产生多态性条带。利用苦荞全基因组序列可实现SSR标记的批量开发,可鉴定出适用于苦荞和甜荞遗传多样性分析、遗传图谱构建和品种鉴定等研究的SSR引物。
| [1] | 胡银岗, 冯佰利, 周济铭, 等. 荞麦遗传资源利用及其改良研究进展. 西北农业学报, 2005,14(5):101-109. |
| [2] | 张丽君, 马名川, 刘龙龙, 等. 山西省苦荞品种资源研究. 河北农业科学, 2015,19(1):69-74. |
| [3] | 李秀莲, 史兴海, 朱慧珺. 苦荞产品开发应用现状及发展对策. 山西农业科学, 2011,39(8):908-910. |
| [4] | 王玲玲, 陈东亮, 黄丛林, 等. SSR分子标记技术在植物研究中的应用. 安徽农业科学, 2017,45(36)::123,126,130. |
| [5] | 杨梦婷, 黄洲, 干建平, 等. SSR分子标记的研究进展. 杭州师范大学学报(自然科学版), 2019,18(4):429-436. |
| [6] |
Iwata H, Imon K, Tsumura Y, et al. Genetic diversity among Japanese indigenous common buckwheat (Fagopyrum esculentum) cultivars as determined from amplified fragment length polymorphism and simple sequence repeat markers and quantitative agronomic traits. Genome, 2005,48(3):367-377.
doi: 10.1139/g04-121 pmid: 16121234 |
| [7] |
Konishi T, Yasui Y, Ohnishi O. Original birthplace of cultivated common buckwheat inferred from genetic relationships among cultivated populations and natural populations of wild common buckwheat revealed by AFLP analysis. Genes and Genetic Systems, 2005,80(2):113-119.
doi: 10.1266/ggs.80.113 pmid: 16172523 |
| [8] | 候雅君, 张宗文, 吴斌. 苦荞种质资源AFLP标记遗传多样性分析. 中国农业科学, 2009,42(12):4166-4174. |
| [9] | 王莉花, 殷富有, 刘继梅. 利用RAPD分析云南野生荞麦资源的多样性和亲缘关系. 分子植物育种, 2004,2(6):807-815. |
| [10] |
Tsuji K, Ohnishi O. Origin of cultivated tartary buckwheat (Fagopyrum tataricum Gaertn.) revealed by RAPD analyses. Genetic Resources and Crop Evolution, 2000,47:431-438.
doi: 10.1023/A:1008741513277 |
| [11] |
Kump B, Javornik B. Genetic diversity and relationships among cultivated and wild accessions of tartary buckwheat (Fagopyrum tataricum Gaertn.) as revealed by RAPD markers. Genetic Resources and Crop Evolution, 2002,49(6):565-572.
doi: 10.1023/A:1021250300572 |
| [12] | 邓琳琼, 张奎, 黄凯丰, 等. 甜荞和苦荞品种遗传多样性的RAPD分析. 安徽农业科学, 2011,39(15):8895-8898. |
| [13] |
Li Y Q, Fan X L, Shi T L, et al. SRAP marker reveals genetic diversity in tartary buckwheat in China. Frontiers of Agriculture in China, 2009,3(4):383-387.
doi: 10.1007/s11703-009-0071-5 |
| [14] |
Konishi T, Iwata H, Yashiro K, et al. Development and characterization of microsatellite markers for common buckwheat. Breed Science, 2006,56:277-285.
doi: 10.1270/jsbbs.56.277 |
| [15] |
Ma K H, Kim N S, Lee G A, et al. Development of SSR markers for studies of diversity in the genus Fagopyrum. Theoretical and Applied Genetics, 2009,119:1247-1254.
doi: 10.1007/s00122-009-1129-8 pmid: 19680622 |
| [16] |
Li Y Q, Shi T L, Zhang Z W. Development of microsatellite markers from tartary buckwheat. Biotechnology Letters, 2007,29(5):823-827.
doi: 10.1007/s10529-006-9293-2 pmid: 17216537 |
| [17] |
高帆, 张宗文, 吴斌. 中国苦荞SSR分子标记体系构建及其在遗传多样性分析中的应用. 中国农业科学, 2012,45(6):1042-1053.
doi: 10.3864/j.issn.0578-1752.2012.06.002 |
| [18] | 韩瑞霞, 张宗文, 吴斌. 苦荞SSR引物开发及其在遗传多样性分析中的应用. 植物遗传资源学报, 2012,13(5):759-764. |
| [19] | 石桃雄, 黎瑞源, 郭菊卉, 等. 基于普通荞麦种子表达序列标签微卫星标记的开发. 贵州农业科学, 2014,42(3):1-5. |
| [20] | 黎瑞源, 潘凡, 陈庆富, 等. 苦荞转录组EST-SSR发掘及多态性分析. 中国农业科技导报, 2015,17(4):42-52. |
| [21] | 杜晓磊, 张宗文, 吴斌, 等. 苦荞SSR分子遗传图谱的构建及分析. 中国农学通报, 2013,29(21):61-65. |
| [22] | 黎瑞源, 梁龙兵, 石桃雄, 等. 苦荞重组自交系群体F5代SSR遗传图谱的构建. 贵州师范大学学报(自然科学版), 2017,35(4):31-45. |
| [23] | 莫日更朝格图, 王鹏科, 高金锋, 等. 苦荞地方种质资源的遗传多样性分析. 西北植物学报, 2010,30(2):255-261. |
| [24] | 田晓庆, 徐宏亚, 汪灿, 等. 用SSR标记分析荞麦栽培种质资源的遗传多样性. 作物杂志, 2013(5):28-32. |
| [25] | 杨学文, 丁素荣, 胡陶, 等. 104份苦荞种质的遗传多样性分析. 作物杂志, 2013(6):13-17. |
| [26] | 徐笑宇, 方正武, 杨璞, 等. 苦荞遗传多样性分析与核心种质筛选. 干旱地区农业研究, 2015,33(1):268-277. |
| [27] |
Zhang L, Li X, Ma B, et al. The tartary buckwheat genome provides insights into rutin biosynthesis and abiotic stress tolerance. Molecular Plant, 2017,10(9):1224-1237.
pmid: 28866080 |
| [28] |
Varshney R K, Graner A, Sorrells M E. Genic microsatellite markers in plants:features and applications. Trends in Biotechnology, 2005,23(1):48-55.
pmid: 15629858 |
| [29] | Lawson M J, Zhang L Q. Distinct patterns of SSR distribution in the Arabidopsis thaliana and rice genomes. Genome Biology, 2006,7(2):R14.1-R14.11. |
| [30] | 原志敏. 玉米全基因组 SSRs 分子标记开发与特征分析. 雅安:四川农业大学, 2013. |
| [31] |
Kantety R V, Rota M L, Matthews D E, et al. Data mining for simple sequence repeats in expressed sequence tags from barley,maize,rice,sorghum and wheat. Plant Molecular Biology, 2002,48:501-510.
pmid: 11999831 |
| [32] |
王玉龙, 黄冰艳, 王思雨, 等. 四倍体野生种花生A. monticola全基因组SSR的开发与特征分析. 中国农业科学, 2019,52(15):2567-2580.
doi: 10.3864/j.issn.0578-1752.2019.15.002 |
| [33] | 李海洋, 李荣华, 夏岩石, 等. 基于RAD-seq数据开发烟草多态性SSR标记. 中国烟草科学, 2018,39(1):1-9. |
| [34] | Song Q, Jia G, Zhu Y, et al. Abundance of SSR motifs and development of candidate polymorphic SSR markers (BARCSOYSSR1.0) in soybean. Crop Science, 2010,50(5):1950-1960. |
| [35] | 郑燕, 张耿, 吴为人. 禾本科植物微卫星序列的特征分析和比较. 基因组学与应用生物学, 2011,30(5):513-520. |
| [36] |
Morgante M, Hanafey M, Powell W. Microsatellites are preferentially associated with nonrepetitive DNA in plant genomes. Nature Genetics, 2002,30(2):194-200.
doi: 10.1038/ng822 pmid: 11799393 |
| [37] | Dreisigacker S, Zhang P, Warburton M L, et al. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different mega environments. Crop Science, 2004,44(2):381-388. |
| [38] |
Temnykh S, DeClerck G, Lukashova A, et al. Computational and experimental analysis of microsatellites in rice (Oryza sativa L.):Frequency,length variation,transposon associations,and genetic marker potential. Genome Research, 2001,11(8):1441-1452.
doi: 10.1101/gr.184001 pmid: 11483586 |
| [39] |
陈仕勇, 马啸, 张新全, 等. 不同来源SSR和EST-SSR在披碱草属和鹅观草属物种中的通用性分析. 草业学报, 2016,25(2):132-140.
doi: 10.11686/cyxb2015324 |
| [40] | 卢玉飞, 蒋建雄, 易自力. 玉米SSR引物和甘蔗EST-SSR引物在芒属中的通用性研究. 草业学报, 2012,21(5):674-679. |
| [1] | 靳建刚, 田再芳. 山西北部地区引种苦荞品种的灰色关联度分析[J]. 作物杂志, 2021, (2): 52–56 |
| [2] | 林淼, 张秋芝, 史利玉, 刘蓓, 王红武, 潘金豹. 玉米秸秆消化率全基因组关联分析[J]. 作物杂志, 2020, (6): 23–29 |
| [3] | 卢晓玲, 何铭, 张凯旋, 廖志勇, 周美亮. 苦荞鼠李糖基转移酶FtF3GT1基因的克隆与转化毛状根研究[J]. 作物杂志, 2020, (5): 33–40 |
| [4] | 杨学乐, 张璐, 李志清, 何录秋. 苦荞种质资源表型性状的遗传多样性分析[J]. 作物杂志, 2020, (5): 53–58 |
| [5] | 公丹, 王素华, 程须珍, 王丽侠. 普通豇豆应用核心种质的SSR指纹图谱构建及多样性分析[J]. 作物杂志, 2020, (4): 79–83 |
| [6] | 李春花, 黄金亮, 尹桂芳, 王艳青, 卢文洁, 孙道旺, 王春龙, 郭来春, 洪波, 任长忠, 王莉花. 苦荞粒形相关性状的遗传分析[J]. 作物杂志, 2020, (3): 42–46 |
| [7] | 李红琴, 刘宝龙, 张波, 张怀刚. 青海省审定小麦品种SSR遗传多样性分析及分子身份证的建立[J]. 作物杂志, 2020, (3): 60–65 |
| [8] | 马孟莉,王田涛,雷恩,孟衡玲,张薇,张婷婷,卢丙越. 基于表型和SSR标记的金平草果遗传多样性分析[J]. 作物杂志, 2020, (2): 54–59 |
| [9] | 马成瑞,向达兵,万燕,欧阳建勇,宋月,唐正松,刘建英,赵钢. 不同苦荞品种花和籽粒空间分布特征及差异分析[J]. 作物杂志, 2020, (1): 35–40 |
| [10] | 杨甜,张永清,董馥慧,马星星,薛小娇. 不同水分条件下不同抗旱性苦荞根系生长规律研究[J]. 作物杂志, 2019, (6): 76–82 |
| [11] | 李松,张世成,董云武,施德林,史云东. 基于SSR标记的云南腾冲水稻的遗传多样性分析[J]. 作物杂志, 2019, (5): 15–21 |
| [12] | 宋丽芳,冯美臣,张美俊,肖璐洁,王超,杨武德,宋晓彦. 外源硒对苦荞生长发育及子粒硒含量的影响[J]. 作物杂志, 2019, (3): 150–154 |
| [13] | 马名川,刘龙龙,张丽君,崔林,周建萍. EMS诱变刺荞的形态突变体鉴定与分析[J]. 作物杂志, 2019, (3): 37–41 |
| [14] | 崔娅松, 王艳, 杨丽娟, 吴朝昕, 周飘, 冉盼, 陈庆富. 米苦荞果壳率及其相关性状的遗传研究[J]. 作物杂志, 2019, (2): 51–60 |
| [15] | 吴磊,王兰芬,武晶,王述民. 普通菜豆根系相关性状的关联分析[J]. 作物杂志, 2019, (2): 61–70 |
|